VIGILÂNCIA LABORATORIAL DOS ENTEROVÍRUS JUNTO AOS REFUGIADOS/REPATRIADOS ADVINDOS DE ÁREAS DE CIRCULAÇÃO DE POLIOVÍRUS – 2022
- Home
- /
- Blog
- /
- Livros
- /
- Capítulo de Livro IV Mostra
- /
- VIGILÂNCIA LABORATORIAL DOS ENTEROVÍRUS JUNTO AOS REFUGIADOS/REPATRIADOS ADVINDOS DE ÁREAS DE CIRCULAÇÃO DE POLIOVÍRUS – 2022
Capítulo de livro publicado no Livro da IV Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
doi.org/10.53934/9786599965821-05
Este trabalho foi escrito por:
Kelly Eduarda da Silva Sanches1; Bráulio Caetano Machado2; Rita de Cássia Compagnoli Carmona2
1Estudante do Curso de Especialização Adolfo Lutz – Núcleo de Doenças Entéricas/Centro de Virologia – Instituto Adolfo Lutz (NDE/CV-IAL); [email protected] 2Docente/pesquisador do NDE/CV – IAL; [email protected]
Resumo: A paralisia flácida aguda (PFA) é a apresentação clínica mais comum da infecção ocasionada pelo poliovírus 1, 2 e 3 (poliomielite). Em 1994 o Brasil recebeu da Organização Mundial de Saúde (OMS)/Organização Panamericana de Saúde (OPAS), a Certificação de área livre de circulação do Poliovírus Selvagem do seu território. À baixa taxa da cobertura vacinal e com a chegada de refugiados/repatriados de áreas com circulação do vírus, o Brasil corre risco de reintrodução do poliovírus. O objetivo deste estudo foi descrever os sorotipos de enterovírus não pólio (EVNP) a partir da vigilância dos poliovírus, fazendo a caracterização genotípica dos materiais de fezes de todo refugiado/repatriado positivo para EVNP, a fim de aumentar a sensibilidade da rede no Estado de São Paulo. As amostras de fezes foram sujeitas ao rastreamento de poliovírus pelo laboratório Instituto Oswaldo Cruz (IOC) – FIOCRUZ, a partir deste resultado, as amostras que deram positivas para ENVP foram submetidas à genotipagem pelo Núcleo de Doenças Entéricas, Centro de Virologia, Instituto Adolfo Lutz (NDE/CV/IAL) utilizando os métodos de purificação e suspensão fecal, transcrição reversa seguida da reação em cadeia pela polimerase convencional (RT- snPCR) para detecção do EVNP e reação de sequenciamento plataforma Sanger para determinação dos genótipos de EVNP. Foram recebidas pelo NDE/CV/IAL 340 amostras de fezes, os EVNP foram detectados em 5,3% (n=18/340) das amostras analisadas, destas 55,5% (N=10/18) foram detectadas e genotipadas, correspondendo ao CVA8 o tipo mais identificado, seguidos por E14, CVA19, E21 e EV-B84. Os dados obtidos contribuem para o conhecimento de quais EVNP estão circulando entre refugiados/repatriados durante janeiro a outubro de 2022 no Estado de São Paulo, Brasil.
Palavras–chave: enterovírus, enterovírus não pólio, paralisia flácida aguda, poliomielite, poliovírus
INTRODUÇÃO
Paralisia Flácida Aguda (PFA) é a manifestação clínica mais comum das infecções causadas pelo poliovírus 1, 2 e 3 (PV) conhecida como poliomielite. O vírus pertence ao
gênero Enterovirus (EV), família Picornaviridae, ordem Picornavirales. A família atualmente consiste em 158 espécies agrupadas em 68 gêneros. O gênero Enterovirus consiste em 15 espécies. Os EVs que infectam humanos são classificados em quatro espécies (EV-A, EV-B, EV-C e EV-D) (Tabela 1). Os sorotipos do poliovírus se encontram na espécie
C. Espécie EV-B tem sido descrito como principais agentes causadores de infecções do sistema nervoso (SNC) e meningite asséptica (MA). Entre esses tipos, os mais comuns são coxsackievirus B (CVB); coxsackievirus A9 (CVA9); echovirus (E) 4, 6, 9, 11, 13, 18, e 30 (CARMONA, R. de C.C, et al., 2020; HARVALA, H. et al., 2018; ICTV, 2020; RACANIELLO, V. R., 2006).
Após o controle dos PV os EVNP emergiram, causando surtos com manifestações clínicas muito similares à PFA, por isso o conhecimento desses vírus contribui para um diagnóstico diferencial, além de detectar e dar respostas a surtos.
OBJETIVO
O objetivo do trabalho é rastrear os enterovírus não pólio a partir da vigilância dos poliovírus, fazendo a caracterização genotípica dos materiais de fezes de todo refugiado/repatriado positivo para EVNP a fim de aumentar a sensibilidade da rede no Estado de São Paulo.
MATERIAL E MÉTODOS
Esse estudo foi realizado com 340 amostras fecais coletadas de refugiados/repatriados menores de 21 anos de idade, advindos de áreas de circulação de poliovírus, no período de janeiro a outubro de 2022.
A demanda de amostras foi estabelecida pela Nota Técnica Conjunta Nº 3/2022 (SES-SP/CCD/CVE/DDTHA/DVIMUNI/DVRESP/CIEVS-SP, 2022). As amostras fecais foram recebidas pelo Núcleo de Doenças Entéricas, Centro de Virologia, Instituto Adolfo Lutz (NDE/CV/IAL), Laboratório de Referência para os Enterovírus no estado de São Paulo e ponto focal da vigilância da PFA/Poliomielite, em seguida encaminhadas ao Laboratório de Referência Nacional das PFA/Poliomielite, Instituto Oswaldo Cruz (IOC), FIOCRUZ, Rio de Janeiro-RJ, para realização do diagnóstico laboratorial de poliovírus, conforme algoritmo preconizado pela OMS. Foram identificadas 18 amostras de fezes caracterizadas previamente como EVNP pelo IOC-FIOCRUZ, destas, 10 foram tipadas por caracterização molecular de genótipos de enterovírus pelo NDE/CV/IAL.
As amostras fecais estocadas a temperatura de -20°C foram preparadas de acordo com a metodologia descrita em Polio Laboratory Manual 2004 (WHO, 2004). Aproximadamente dois gramas de fezes foram adicionados a 10mL de uma solução tampão fosfato estéril, pH 7,4. As amostras foram agitadas e submetidas ao processo de centrifugação durante 60 minutos a 10.000xg em centrífuga refrigerada a 4°C. Em seguida, os sobrenadantes foram coletados e tratados com solução de penicilina (100U/mL) e estreptomicina (100ug/mL) por 30 minutos. Após este período os sobrenadantes foram estocados a baixas temperaturas -70° C até a realização dos testes.
O ssRNA dos EVNP foi extraído diretamente da suspensão fecal purificada, utilizando extrator automático Loccus, Kit – Extracta Fast 96 ou extração manual feita com o Kit Bio Gene ou Kit Qiamp viral RNA mini, de acordo com instruções do fabricante. A reação da transcrição reversa seguida da reação em cadeia pela polimerase convencional (RT-snPCR) foi realizada de acordo com o protocolo descrito por Nix, Oberste e Pallansch, (2006); WHO, (2015). A visualização dos produtos amplificados foi obtida por eletroforese em gel de agarose (1,5%) corado com corante de ácido nucleico (Gel Red® Nucleic Acid Stain – Biotium, INC., CA, USA). Foi utilizado marcador de peso molecular de 100pb (DNA Ladder, Invitrogen®). O sistema de corrida foi submetido a uma corrente elétrica constante de 100 Volts por 60 minutos. Após eletroforese os produtos amplificados foram observados em transiluminador de luminescência ultravioleta e o resultado captado pelo sistema de fotodocumentação digital UV Transilluminator, UVP Laboratory Products. A imagem salva foi impressa, analisada e arquivada.
Os produtos da RT-snPCR foram submetidos à reação de sequenciamento genômico pelo método de dideoxinucleotídeo, SANGER, F.; NICKLEN, S; COULSON, A.R. (1977), utilizando o kit BigDye® Terminator v3.1 Cycle Sequencing Ready Reaction Kit (Applied Biosystems®), com termociclagem de 25 ciclos de 96ºC por 10 segundos, 50°C por 5 segundos e 60°C por 4 minutos. Foram utilizados os mesmos primers da reação de RT- snPCR. O produto de sequenciamento foi precipitado utilizando o método de acetato e etanol. Os produtos precipitados foram ressuspendidos em 10μL de formamida Hi-Di® (Applied Biosystems®, Inc., USA) foram analisados no sequenciador automático modelo Applied Biosystems 3130xl Genetic Analyzer (Applied Biosystems®), utilizando o polímero POP-7® (Applied Biosystems®, Inc., USA).
As sequências dos genes obtidas foram editadas manualmente utilizando o programa Sequencher 4.7 (Gene Codes Corporation, Michigan, USA), para obtenção de sequências consensos. A ferramenta Enterovirus Genotyping Tool version 0.1 disponível no site http://www.rivm.nl/mpf/enterovirus/typingtool foi utilizada para confirmação dos genótipos de EVNP (Kroneman et al., 2011).
O TCC faz parte do Projeto de Pesquisa sob coordenação da Pesquisadora Científica Dra. Rita de Cássia Compagnoli Carmona, com aprovação quanto aos aspectos éticos pelo Comitê de Ética do Instituto Adolfo Lutz (IAL) e aprovação quanto ao mérito científico pelo Conselho Técnico Científico (CTC) do IAL – cadastro CTC 35-I /2016.
RESULTADOS E DISCUSSÃO
Entre os resultados positivos para EVNP disponibilizados pelo laboratório IOC- FIOCRUZ, 55,5% (n=10/18) foram caracterizados por sequenciamento genômico pelo NDE/CV/IAL (Tabela 1).
Os EVNP foram detectados em 5,3% (n=18/340) das amostras analisadas. 93,5% (n=318/340) negativas, n=4/340 (1,2%) poliovírus vacinal tipo 1 e 3, conforme representado no gráfico 1.
Dentre 5,3% (n=18/340) dos EVNP detectados, 55,5% (n=10/18) são genótipos caracterizados pelo NDE/CV/IAL. Cinco tipos distintos de EVNP foram identificados.
CVA8 espécie EV-A, foi o tipo mais comumente identificado (60%, n=6/10) seguido por E14 espécie EV-B, CVA19 espécie EV-C, E21 espécie EV-B, e EV-B84 espécie EV-B, cada um (10%, n=1/10) de acordo com o gráfico 2.
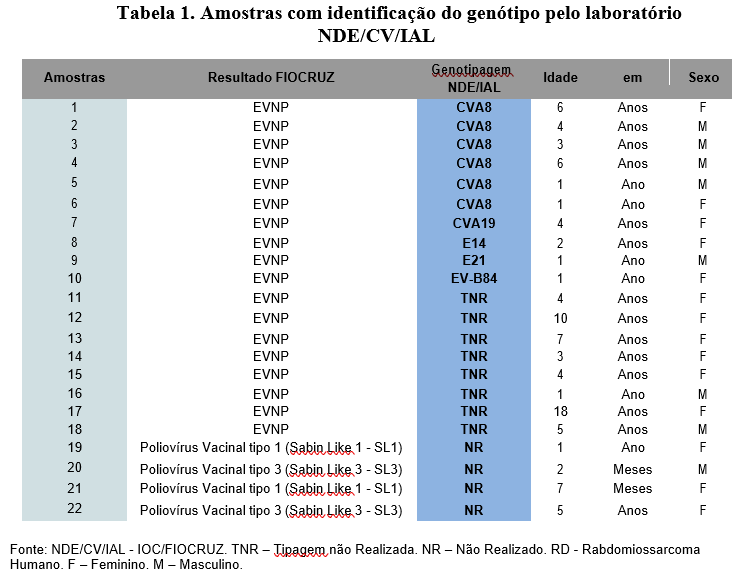
Gráfico 1 – Amostras de refugiados/repatriados menores de 21 anos entre janeiro a outubro de 2022
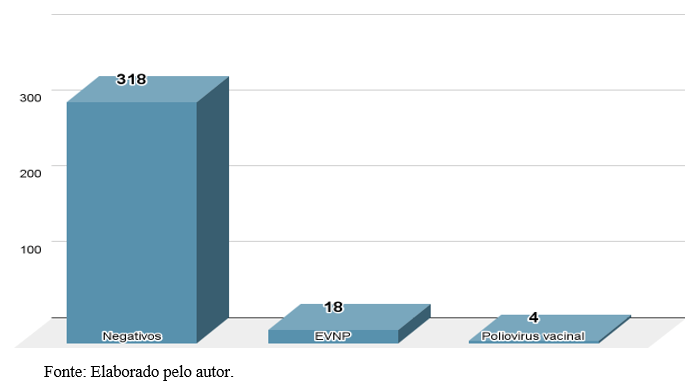
Gráfico 2 – Genótipos de EVNP caracterizados por sequenciamento genômico.
Os resultados obtidos neste estudo foram coletados de um grupo que não possui sintomas, com o objetivo de rastrear quais sorotipos de EVNP estão em circulação, diante da presença de pessoas advindas de área de circulação de poliovírus. É importante fazer o rastreamento dos EVNP, devido ao aumento dos casos de PFA ocasionados por esses vírus, relatando que nessa população estudada está ocorrendo à circulação de EVNP.
Diante da vigilância dos poliovírus no Brasil, os materiais coletados foram fezes, e por serem pessoas assintomáticas, que não possuem manifestações clínicas, não foi necessária a coleta de outro tipo de material biológico mais invasivo. Em casos de infecções neurológicas, as amostras de fezes e respiratórias têm sido consideradas as mais convenientes para serem submetidas ao teste de EV, além de LCR e sangue (CARMONA, R. de C.C, et al, 2020).
O coxsackievirus e echovirus foram relacionados a surtos com comprometimento motor (DIAS, A. P. M., 2018). O coxsackievirus A8 (CVA8), pertence à espécie EV-A foi o mais encontrado neste estudo, tem um perfil de causar doença febril, doença de mão pé boca, herpangina (HA), mas ocasionalmente levam a distúrbios neurológicos graves, como PFA. Em 2012 na Tailândia, o CVA8 foi a causa mais prevalente de HA. (SONG, Y. et al., 2020). O coxsackievirus A19 (CVA19) pertence à espécie EV-C, e tem sido associado a casos de doenças respiratórias e gastrointestinais, meningite asséptica, gastroenterite, PFA e enterite aguda (Yi, L., Zhang, L., Feng, L. et al, 2022).
O enterovirus B84 (EV-B84) pertence à espécie EV-B, causa viral mais comum de miocardite aguda, paralisia flácida aguda e meningite asséptica. A taxa de isolamento do vírus é extremamente baixa e não é um sorotipo prevalente no mundo. O primeiro relato do vírus na China foi de um paciente com PFA em 2004 (ZHENG, H. et al., 2016).
O echovirus 14 (E14) pertence à espécie EV-B está associado a casos de síndrome gastrointestinal, meningite asséptica, paralisia, necrose hepática neonatal fatal, o genótipo também é considerado raro (CHEN, P. et al., 2017). O echovirus 21 (E21) pertence à espécie EV-B, este genótipo não possui muitos isolados, mas já foi isolado de um paciente com PFA (LIU, H. et al., 2020).
No Brasil, um estudo de 1.215 amostras de paciente com suspeita de doenças infecciosas do SNC, realizado no Estado de São Paulo entre 2004 a 2014 por CARMONA, R. de C.C, et al (2020), identificou o E14 que foi associado a doença meningite asséptica (MA), e o E21 associado a meningoencefalite, ambos encontrados em (0,6%; n = 1/176) amostras positivas. E-6 foi o mais comumente identificado, seguido E-30; E18; CV-B5; E4; E11; CVB2 e E9; E7; CVA9, CVB1, CVB3, CVB4, E13, E14 e E21. Segundo CARMONA, R. de C.C, et al (2020) no Brasil os EVs foram detectados durante todo o ano e a variação sazonal é mais proeminente durante a primavera, já o pico de ocorrência foi no verão.
As amostras são de refugiados, sendo assim, esses genótipos podem ser comuns em seu país de origem, a chegada ao Brasil pode aumentar a probabilidade da introdução de genótipos não circulantes.
CONCLUSÕES
Os resultados encontrados de EVNP (CVA8, CVA19 E14, E21 e EV-B84), demonstram conhecimento de quais genótipos estão circulando entre refugiados/repatriados assintomáticos durante janeiro a outubro de 2022 no Estado de São Paulo, Brasil, demonstrando o perfil de circulação silenciosa dos EVs, que podem sofrer mutações até causarem manifestações clínicas mais graves, e em grandes surtos essas doenças podem ter uma grande proporção. Os achados não foram de casos de PFA, mas estudos anteriores relacionaram esses genótipos a casos de PFA. A vigilância epidemiológica e laboratorial tem feito um papel de extrema importância no controle e no rastreamento dos EVNP, a partir da grande comoção que a poliomielite tem causado com o risco de reintrodução no país, porém, vemos que outros agentes estão circulando com potencial de causar PFA e a probabilidade da introdução de novos genótipos no Brasil. Outros estudos precisam ser feitos para que os EVNP não venham causar grandes surtos semelhantes aos poliovírus.
AGRADECIMENTOS
Agradeço aos colegas Adriana Jorge, Amanda Dias, Antônio Erculiani, Daniele de Moraes, Fabricio Caldeiras, Gabriel Ferreira, Gisele Nemer e Lucas Leme do Núcleo de Doenças Entéricas, Centro de Virologia, Instituto Adolfo Lutz (IAL), que realizaram o diagnóstico dos Enterovírus Não Pólio, a Divisão de Doenças e Transmissão Hídrica e Alimentar, Centro de Vigilância Epidemiológica, Coordenadoria de Controle de Doenças, Secretaria de Estado da Saúde, São Paulo e o Laboratório de Enterovírus, Instituto Oswaldo Cruz (IOC), FIOCRUZ, Ministério da Saúde.
REFERÊNCIAS
CARMONA, R. de C.C, et al. Distribution of species enterovirus B in patients with central nervous system infections in São Paulo State, Brazil. Journal of Medical Virology. v. 92, ed. 12, 2020. Disponível em: <https://doi.org/10.1002/jmv.26131>. Acesso em: 02 nov. 2022.
HARVALA, H. et al. Recommendations for enterovirus diagnostics and characterisation within and beyond Europe. In: Journal of Clinical Virology. v. 101, p. 11-17, abr. 2018. Disponível em:
<https://doi.org/10.1016/j.jcv.2018.01.008>. Acesso em: 22 nov. 2022.
INTERNATIONAL COMMITTEE ON TAXONOMY OF VIRUSES EXECUTIVE COMMITTEE.
The new scope of virus taxonomy: partitioning the virosphere into 15 hierarchical ranks. Nat Microbiol. v. 5, ed. 5, p. 668-674, 2020. Disponível em: <doi: 10.1038/s41564-020-0709-x>. Acesso
em: 10 nov. 2022.
RACANIELLO, V. R. One hundred years of poliovirus pathogenesis. In: Virology. v. 344, ed. 1. 2006. Disponível em: <https://doi.org/10.1016/j.virol.2005.09.015>. Acesso em: 05 nov. 2022 SANGER, F.; NICKLEN, S; COULSON, A.R. DNA sequencing with chain-terminating inhibitors. Proc Natl Acad Sci U S A. ed.12, v. 74, p.5463-7. 1977.
SECRETARIA DE ESTADO DA SAÚDE DE SÃO PAULO. COORDENADORIA DE CONTROLE DE DOENÇAS – CCD. CENTRO DE VIGIL NCIA EPIDEMIOLÓGICA “PROF. ALEXANDRE VRANJAC”. DIVISÃO DE DOENÇAS DE TRANSMISSÃO HÍDRICA E ALIMENTAR – DDTHA. DIVISÃO DE IMUNIZAÇÃO – DVIMUNI. DIVISÃO DE DOENÇAS DE TRANSMISSÃO RESPIRATÓRIA – DVRESP. CENTRO DE INFORMAÇÕES ESTRATÉGICAS EM VIGIL NCIA EM SAÚDE –CIEVS-SP. NOTA CONJUNTA Nº 3/2022 –
DDTHA/CIEVS-SP/DVIMUNI/DVRESP/CVE/CCD/SES-SP. Alerta para risco de reintrodução da poliomielite no Brasil e recomendações para ações de Vigilância Epidemiológica das Paralisias Flácidas Agudas (PFA) junto aos refugiados/repatriados advindos de áreas de circulação de poliovírus (selvagem e/ou derivado vacinal). São Paulo, 2022.
WORLD HEALTH ORGANIZATION. Polio laboratory manual 4th edition.
Geneva 27, Switzerland: Department of Immunization, Vaccines and Biologicals CH- 1211, World Health Organization; p. 1–167. 2004.
NIX, W.A.; OBERSTE, M.S; PALLANSCH, M.A. Sensitive, seminested PCR amplification of VP1 sequences for direct identification of all enterovirus serotypes from original clinical specimens. J Clin Microbiol. v. 44, p. 2698–2704, 2006.
WORLD HEALTH ORGANIZATION. Enterovirus surveillance guidelines: Guidelines for enterovirus surveillance in support of the Polio Eradication Initiative, Geneva. World Health Organization; 2015. p.1-46.
SANGER, F.; NICKLEN, S; COULSON, A.R. DNA sequencing with chain-terminating inhibitors. Proc Natl Acad Sci U S A. ed.12, v. 74, p.5463-7. 1977.
KRONEMAN, A. et al. An automated genotyping tool for enteroviruses and noroviruses. Journal of Clinical Virology. v. 51, ed. 2, p. 121-5. jun. 2011. Disponível em: . Acesso em: 25 nov. 2022.
DIAS, A. P. M. Avaliação de um protocolo visando o diagnóstico rápido dos enterovírus associados a casos de paralisia flácida aguda. 2008. 73 f. Dissertação (Mestrado em Vigilância Sanitária) – Instituto Nacional de Controle de Qualidade em Saúde, Fundação Oswaldo Cruz, Rio de Janeiro, 2008.
SONG, Y. et al. Genetic Diversity Analysis of Coxsackievirus A8 Circulating in China and Worldwide Reveals a Highly Divergent Genotype. Viruses. v. 12, ed.10, p.1061, set. 2020. Disponível em: <https://www.mdpi.com/1999-4915/12/10/1061> . Acesso em: 25 dez. 2022.
ZHENG, H. et al. Isolation and Characterization of a Highly Mutated Chinese Isolate of Enterovirus B84 from a Patient with Acute Flaccid Paralysis. Sci Rep. v. 6, n. 31059, ago. 2016. Disponível em:
<https://www.nature.com/articles/srep31059> . Acesso em: 26 nov. 2022.
CHEN, P., et al. Filogeografia evolutiva e padrão de transmissão do echovirus 14: uma exploração da dinâmica espaço-temporal com base na vigilância da paralisia flácida aguda de 26 anos em Shandong, China. BMC Genomics. v. 18, n. 48, jan. 2017. Disponível em:<https://doi.org/10.1186/s12864-016-3418-3>. Acesso em: 27 nov. 2022.
LIU, H. et al. Caracterização de uma nova cepa de echovirus 21 isolada de uma criança saudável na China em 2013. Arch Virol. v. 165, p. 757–760, jan. 2020. Disponível em: <https://doi.org/10.1007/s00705-019-04506-4> Acesso em: 25 nov. 2022.

