AVALIAÇÃO ENTRE OS MÉTODOS DE RT-qPCR, RT-snPCR E ISOLAMENTO DE ENTEROVIRUS EM CULTURA CELULAR EM CASOS SUSPEITOS DE DOENÇA DE MÃO-PÉ-BOCA NO ESTADO DE SÃO PAULO
- Home
- /
- Blog
- /
- Resumo expandido TCC
- /
- AVALIAÇÃO ENTRE OS MÉTODOS DE RT-qPCR, RT-snPCR E ISOLAMENTO DE ENTEROVIRUS EM CULTURA CELULAR EM CASOS SUSPEITOS DE DOENÇA DE MÃO-PÉ-BOCA NO ESTADO DE SÃO PAULO
Resumo expandido publicado nos Anais da III Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
Este trabalho foi escrito por:
Gabriel Henriques Ferreira*¹, Bráulio Caetano Machado1, Rita de Cássia Compagnoli Carmona1
*Aluno do Curso de Especialização em Vigilância Laboratorial em Saúde Pública;1Núcleo de Doenças Entéricas, Centro de Virologia, Instituto Adolfo Luz; E-mail: [email protected]; [email protected]; [email protected]
RESUMO
A doença de mão-pé -boca (DMPB) é causada por vírus do gênero Enterovirus (EV), dentre eles destacam-se os sorotipos: coxsackievirus, echovirus, enterovirus A71. A doença acomete majoritariamente crianças de até 5 anos e os principais sintomas são: febre, erupção cutânea em mãos, pés e vesículas na boca. Geralmente a doença é autolimitada, mas pode evoluir para casos graves com tropismo ao sistema nervoso. Os EV possuem prevalência no mundo todo, e a DMPB é endêmica em países asiáticos e oeste do pacífico. No Brasil somente os surtos são de notificação compulsória. Desta forma, o objetivo deste trabalho foi comparar as metodologias utilizadas para o diagnóstico dos EV, reverse transcription quantitative PCR (RT-qPCR), isolamento viral em sistema de cultura celular e a reverse transcription semi-nested PCR (RT-snPCR) utilizando amostras clínicas de suspeitas de DMPB obtidas entre maio e outubro de 2021 no Estado de São Paulo. Os resultados mostraram boa concordância entre RT-qPCR e isolamento viral, assim como entre os dois ensaios de biologia molecular, em detrimento da concordância regular entre isolamento viral e RT-snPCR. Destacam-se maiores sensibilidade da RT-qPCR (92,3; 85,1%) e cultura celular (88,4%; 87,1%). Especificidade da RT-snPCR (84,3%; 91,6%), RT-qPCR (70,2%; 81,2%) e cultura celular (76,4%; 57,4%). Valor preditivo positivo da RT-snPCR (92,3%; 87,1%) cultura celular (72,0%; 88,4%)e RT-qPCR (85,1%; 62,9%). Os resultados corroboram aos relatos da literatura, sugerindo o ensaio de RT-qPCR para detecção de rotina de EV, mas expressa a importância do isolamento viral e da RT-snPCR para melhorar estratégias de vigilância epidemiológica, reconhecendo o isolamento de enterovirus em cultura celular como poderosa ferramenta.
Palavras-chave: Doença de Mão, Pé, Boca; Epidemiologia; PCR em tempo real; Enterovírus
INTRODUÇÃO
A doença de mão-pé-boca (DMPB) é causada por enterovírus humanos não-pólio, com sorotipos importantes como coxsackievirus, echovirus, enterovirus A71 (EVA71). O EV apresenta 15 espécies, sendo 7 descritas em humanos, apresentando mais de 100 sorotipos (TAPPAREL et al., 2013, COMPAGNOLI CARMONA et al., 2020). Não possuem envelope viral e seu genoma dispõe de fita única de RNA senso positivo (ssRNA+) (WELLS e CONEY, 2019). A DMPB acomete frequentemente crianças menores de 5 anos, causando manifestações clínicas leves e evolução autolimitada. Os EV da DMPB se replicam nas tonsilas palatinas, mucosa da orofaringe e substancialmente no intestino delgado. Os principais sintomas são febre, erupção cutânea em mãos e pés e vesículas na boca (MORTARI et al., 2018). Casos clínicos mais graves podem ocorrer incluindo meningite asséptica, encefalite e paralisia flácida aguda (TAPPAREL et al., 2013). A transmissão pode ocorrer por muco e saliva, mas primariamente pela via fecal-oral (WELLS e CONEY, 2019). Na China, Du e colaboradores (2017) relatam altíssimas taxas de infecções, R0 de até 30, ou seja, 1 pessoa pode transmitir para outras 30, considerando somente população suscetível, as crianças. Logo, as creches possuem altos índices de transmissão.
Isolamento viral em sistema de cultura celular, reverse transcription quantitative PCR(RT-qPCR) e a reverse transcription semi-nested PCR (RT-snPCR) estão entre os ensaios de identificação dos EV da DMPB. O ensaio RT-qPCR é recomendado por Harvala e colaboradores (2018) para uso na rotina de diagnóstico, por ser mais rápido e mais sensível e simples. Mas o isolamento viral em cultura celular é importante para laboratórios de referência permitindo o emprego junto a outros testes de identificação, como a RT-PCR convencional e o sequenciamento genômico (MORTARI et al., 2018). O ensaio RT-snPCR também é empregado para identificação de EV através de detecção da região da VP1. A proteína estrutural VP1 do capsídeo está localizada na região mais exterior ao vírus e é um antígeno de grande importância. O sequenciamento parcial do gene VP1 é empregado para busca de sorotipos, e o sequenciamento completo para identificação de novos sorotipos em trabalhos de vigilância epidemiológica (HARVALA et al., 2018). Já a RT-qPCR contribui de forma significativa para diagnóstico rápido de EV em saúde pública, diminuindo os custos sobre o uso indiscriminado de antibiótico e hospitalizações e melhorando a assistência ao paciente (MACHADO et al., 2016).
Os métodos de biologia molecular trouxeram inúmeras vantagens para a detecção dos EV, tanto para diagnóstico como para vigilância epidemiológica. Entretanto, observa-se algumas divergências, como resultados falsos negativos na RT-qPCR que são detectados no método de isolamento viral em cultura de células, levantando discussões sobretudo quanto à vigilância epidemiológica em elaboração de fluxogramas de trabalho. Com isso, o objetivo do trabalho foi comparar as metodologias de RT-qPCR, RT-snPCR e Isolamento viral em cultura de célula, sob perspectiva de detecção de EV em amostra clínica provenientes de surtos de DMPB ocorridos no Estado de São Paulo entre maio e outubro de 2021. Para isso foram calculados o índice Kappa, sensibilidade, especificidade, acurácia, valor preditivo positivo e negativo.
MATERIAL E MÉTODOS
Foram selecionadas 86 amostras clinicas, dentre elas 24 SOF (swabde orofaringe), 56 de fezes e 6 SNF (swab de nasofaringe). O uso destas amostras foi aprovado pelo Comitê de Ética em Pesquisa do Instituto Adolfo Lutz, CAAE: 28405120.2.0000.0059, cadastro: CTC 60-L/ 2019. As amostras de fezes passaram por clarificação antes de todos os ensaios, para obter somente o material líquido, de acordo com a metodologia descrita em Polio Laboratory Manual 2004 (WHO). Para proceder aos ensaios de biologia molecular os materiais clínicos passaram por extração de RNA por coluna de sílica, realizado com o kit comercial Bio Gene® de extração de DNA/RNA (Bioclin®, Belo Horizonte, Brasil) e adicionado inibidor de RNAse (RNA out) ao produto extraído.
O ensaio de RT-qPCR foi executado com sondas Taqman® e primers ambos desenhados para detecção universal de EV (HEV) por meio da região alvo 5’ NCR (Non-Coding Region) do genoma. Foi utilizada a reação em one step RT-qPCR, conforme descrito por MACHADO e colaboradores (2016). Para avaliar a qualidade das amostras processadas com relação à recuperação do RNA extraído e à presença de possíveis inibidores da PCR, foi realizada um controle interno endógeno cujo alvo foi a Rnase P humana seguindo o protocolo recomendado pelo CDC para detecção do vírus Influenza (CDC, 2009; SHU et al., 2011). Foram utilizados primer Senso CCCTGAATGCGGCTAATCC, primer anti-senso ATTGTCACCATAAGCAGCCA; sonda 6-FAM-AACCGACTACTTTGGGTGTCCGTGTTTC-BHQ1; RnaseP senso AGATTTGGACCTGCGAGCG; RnaseP anti-senso GAGCGGCTGTCTCCACAAGT; RnaseP sonda 6-FAM d(TTCTGACCTGAAGGCTCTGCGCG) BHQ-1.
Utilizou-se para a reação as concentrações dos reagentes da reação para detecção de EVs: 300 nM (primer senso), 900 nM (primer anti-senso), 150 nM (probe), 1× kit “SuperScript™ III Platinum™ One-Step qRT-PCR Kit” (Invitrogen ™) e 5 μL do RNA viral extraído, resultando no volume total de 25 μL. O controle interno endógeno: primers na concentração de 40 nM e a sonda, 10 nM. A reação foi realizada no equipamento 7500 Real Time PCR Systems (Applied Biosystems, Foster, CA, USA). A transcrição reversa foi realizada a 50ºC por 15 minutos, seguida de 1 ciclo a 95 ºC por 2 minutos, e 45 ciclos das temperaturas de 95 ºC por 15 segundos e 60 ºC por 1 minuto. Foram colocados 1 controle de reagentes e de ambiente (NTC1 – no template control) e 1 controle positivo em duplicata (PTC – positive template control). Foram consideradas positivas as amplificações em até 40 Ct (Cycle Threshold). Conforme predefinição laboratorial, Cts tardios (40+4) em contexto de surto foram considerados positivos.
A RT-snPCR utilizou como alvo a região VP1 do genoma dos EV e o ensaio foi realizado com reagentes do kit Invitrogen™. A primeira amplificação foi realizada em um ensaio de RT-PCR de tubo único contendo 1,25 mM de cada desoxinucleotídeos; Ditiotreitol 0,1 M; Tampão 1 × 5 × (Tris-HCl 300 mM, pH 9,0 e 62,5 mM NH4, 2SO4); Tampão 0,5 × 10 × (Tris-HCl 100 mM, pH 9,0, KCl 500 mM e Triton X-100 1%); MgCl2 1,25 mM; 20 μM de pool de primers 224-222 (SO 224, GCIATGYTIGGIACICAYRT do Gene VP3;SO 222, CICCIGGIGGIAYRWACAT do gene VP1); SuperScript III Reverse Transcriptase; 2 U de Platinum Taq DNA Polymerase 19; e RNAsin. Foram utilizados 5 μL de ssRNA extraidos. As reações foram incubadas a 42°C durante 50 minutos, seguidas de 94 ° C durante 5 minutos. A termociclagem: 35 ciclos de 94 ° C, 1 minuto; 50 ° C, 2 minutos; 72 ° C, 1 minuto; e uma incubação a 72 ° C, 5 minutos; em um termociclador modelo 9700 (Applied Biosystems, Foster City, CA). A 2ª reação de PCR, foram empregados 40 picomol de cada um dos primers AN89 (CCAGCACTGACAGCAGYNGARAYNGG do gene VP1) e AN88 (TACTGGACCACCTGGNGGNAYRWACAT do gene VP1), 200 µM de dNTP, 5 µL de 10X tampão de PCR, MgCl2 e 2,5 U de Platinum®Taq DNA polimerase. Um microlitro da 1ª PCR (dsDNA) foi adicionado a reação de 2ª PCR, com volume final de 50 µL e submetida a termociclagem de 40 ciclos de 95 °C por 30 segundos, 60 °C por 20 segundos, 72 °C por 15 segundos.
O produto amplificado foi submetido à eletroforese em gel de agarose a 1,5% diluída em 100mL de tampão TBE 1x (pH 8,0), acrescido de 5 μL de GelRed™ Nucleic Acid Gel Stain(Biotium, Inc., CA, EUA). Um volume de 7 μL do produto amplificado foi misturado com tampão de aplicação (azul de bromofenol 1% e sacarose proporção 1:1). Foi utilizado marcador de peso molecular de 100pb (DNA Ladder, Invitrogen™). O sistema de corrida foi submetido a uma corrente elétrica constante de 100 Volts por 60 minutos.
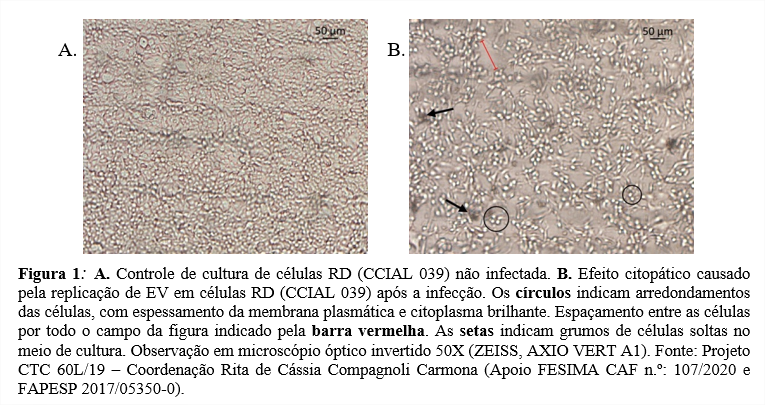
No isolamento de EV em Cultura de Células foram utilizadas células de RD (rabdomiossarcoma humano, CCIAL 039, linhagem pertencente à Coleção de Cultura de Células do Instituto Adolfo Lutz, registrado na World Federation Culture Collection). Primeiramente os tubos de cultura de células foram submetidos à lavagem com solução salina tamponada e posterior troca do meio de crescimento (Eagle + L15 + SFB 15%) para 1 mL meio de manutenção (Eagle sem SFB + L15 + Hepes + penicil./estreptom.). Sequencialmente foram feitas 3 passagens com as amostras clínicas em duplicata. 1ª passagem: para SOF e NSF realizou-se a adsorção: desprezou-se o meio de manutenção e aplicando amostra clínica, deixando em estufa a 37 °C por 30 minutos. Depois foi aplicado 1mL de meio de manutenção. As amostras foram deixadas em estufa a 37 °C por 7 dias realizando leituras diárias em microscópio invertido para investigação de efeito citopático. 2ª e 3ª passagens: após congelamento e descongelamento por 3 vezes, as amostras foram submetidas a novas passagens. 0,2 mL de passagens anteriores foram colocados em novos tubos de cultura de células em meio de manutenção (COMPAGNOLI CARMONA et al., 2020). Controles com meio de manutenção: lavou-se em solução salina e trocou-se para meio de manutenção. O controle com meio de crescimento: a cultura de células com o meio de crescimento foi colocada diretamente em estufa. Amostras foram consideradas positivas somente se visualizado o efeito citopático em pelo menos 1 das duplicatas durante as 3 passagens.
RESULTADOS E DISCUSSÕES
Analisando os dados, o ensaio RT-qPCR manteve uma boa concordância com os dois outros métodos (Tabela 1, índice Kappa 0,65 e 0,61), mostrando ser mais adequado para comparar os resultados de biologia molecular e o isolamento viral em cultura celular. Já a relação entre RT-snPCR e cultura de célula obteve concordância regular (índice Kappa entre 0,43) diminuindo a confiabilidade dos dados.
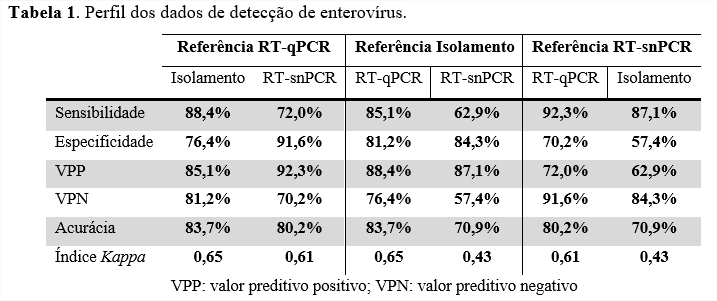
Ao colocar a RT-qPCR como referência observou maior sensibilidade da cultura de célula (88,4%) comparado ao RT-snPCR (72,0%). Isto sugere que os sorotipos isolados (Figura 1) correlacionaram com a detecção em rotina de diagnóstico, porém 6 amostras não foram cultiváveis, sendo 5 em SOF e 1 em fezes. No trabalho de Vijayaraghavan et al. (2012) relatou-se que durante um surto de DMPB na Índia o material de swab de faringe (material respiratório) alcançou uma das melhores taxas de detecção de EV em cultura de célula, mas trabalhou-se conjuntamente com células RD e Vero. Isto se alinha à recomendações sobre a utilização de mais de um tipo de linhagem celular para aumentar a sensibilidade na detecção de EV, porém é de pleno conhecimento que isto eleva os custos e tempo de trabalho substancialmente.
A maior sensibilidade do trabalho alcançado pela RT-qPCR (92,3%), sob referência da RT-snPCR, sugere que a detecção da região 5’NCR pela PCR em tempo real está de acordo com a detecção da região VP1 do ensaio de semi-nested. Porém o inverso não alcançou nível semelhante (sensibilidade 72,0% da RT-snPCR tendo a RT-qPCR como referência). Isto demonstra que os ensaios em tempo real com detecção Taqman são mais eficientes devido ao tamanho do fragmento alvo de amplificação ser menor (por volta de 100 pb). Já no RT-snPCR, 750 pb (1ªPCR) e 360 pb (2ª PCR) (OBERSTE et al., 2010).
Os casos positivos da PCR em tempo real com Cts tardios (40+4) somaram-se 9 casos e somente 1 destes não foi detectado no isolamento viral em cultura de célula nem em RT-snPCR. Desta forma, os Cts tardios podem ter a relação com a qualidade da extração de RNA ou a qualidade da amostra no momento da coleta. O Kit de extração Biogene utilizado no trabalho é também empregado para extração do vírus SARS-CoV-2 obtendo bons resultados, com Cts positivos ≤ 40 (CAMPOS et al., 2021). Porém isto não foi observado em EV no presente trabalho. Portanto, foi predefinido que Cts tardios inseridos em contexto de surto de DMPB estariam classificados como ‘’detectado’’.
Os dados apresentados se aproximam da recomendação de Harvala et al. (2018) na qual a RT-qPCR é indicado para rotina de diagnóstico por ser rápido, simples e sensível. Com o mesmo pensamento, o trabalho de Kabuga e et al. (2021) também sugerem a aplicação do ensaio em tempo real para diagnóstico dos EV, mesmo o método de isolamento superando-o em sensibilidade. Mas isto demonstra, assim como o presente trabalho, a poderosa ferramenta de detecção que é o isolamento viral, sendo importante para estudos de vigilância, sobretudo para aumentar a carga viral em amostras positivas na RT-qPCR para caracterização posterior por sequenciamento genômico. Da mesma forma, a RT-snPCR, confere informações importantes para posteriores caracterizações moleculares.
CONCLUSÕES
Os resultados indicam que a RT-qPCR e a metodologia de isolamento viral em cultura celular obtiveram boa confiabilidade com altos níveis de sensibilidade na detecção de EV para a DMPB, em detrimento de menor confiabilidade entre RT-snPCR e isolamento em cultura celular. Desta forma sugere-se o ensaio de RT-qPCR como referência para detecção de rotina de EVs, enquanto que para trabalhos de vigilância epidemiológica, a cultura de célula e a RT-snPCR. Reconhecendo o isolamento em cultura de células como poderosa ferramenta na pesquisa dos EVs.
AGRADECIMENTOS
Agradecemos aos colegas Adriana Jorge, Amanda Dias, Fabricio Caldeiras, Lucas Leme, Daniele de Moraes, Isabella Monteiro, Antônio Junior e Dra. Audrey C. Chirelli do Núcleo de Doenças Entéricas, Centro de Virologia, Instituto Adolfo Lutz (IAL) pelo apoio técnico e oportunidade de desenvolver o trabalho e ao Núcleo de Cultura de Células (IAL) também pelo apoio técnico. Apoio: Fundo Especial de Saúde para Imunização em Massa e Controle de Doenças (Fesima). Coordenadoria de Controle de Doenças. Secretaria de Estado da Saúde. São Paulo (CAF n.º: 107/2020) e FAPESP (2017/05350-0).
REFERÊNCIAS
CAMPOS, K. R. et al. COVID-19 laboratory diagnosis: comparative analysis of different RNAextraction methods for SARS-CoV-2 detection by two amplification protocols. Rev. Inst. Med. trop. v. 63, ed. 52. 2021.
CDC. Protocol of realtime RTPCR for swine influenza A(H1N1). CDC Atlanta, EUA. p. 8. 2009.
COMPAGNOLI CARMONA, R.C. et al. Distribution of species enterovirus B in patients with central nervous system infections in São Paulo State, Brazil. J Med Virol. 2020.
DU, Z. et al. Estimating the basic reproduction rate of HFMD using the time series SIR model in Guangdong, China. PloS one. v. 12, ed. 0179623. 2017.
HARVALA, H. et al. Recommendations for enterovirus diagnostics and characterisation within and beyond Europe. J Clin Virol. v. 101, p. 11-17. 2018.
KABUGA, A. I, et al. Cell culture demonstrates superior sensitivity over one step real time RT PCR and nested VP1 amplification for Enteroviruses. Journal of Virological Methods. v. 287, 2021.
MACHADO, B.C.; ALVES, M.R.M.; CARMONA, R.C.C. Avaliação do desempenho da PCR em Tempo Real para o diagnóstico de meningite por enterovírus. Rev Inst Adolfo Lutz. v. 75, n. 1695, p.6. 2016.
MORTARI, N. et al. Hand-Foot-Mouth Disease: guidelines and outbreaks management. BEPA, Boletim Epidemiológico Paulista (Impresso), v. 15, n.173, p. 11-28, 2018.
OBERSTE, M. S. et al. Comparative evaluation of Taqman real-time PCR and semi-nested VP1 PCR for detection of enteroviruses in clinical specimens. Journal of Clinical Virology. v. 49, issue 1, p. 73-74. 2010.
SHU, B. Design and Performance of the CDC Real-Time Reverse Transcriptase PCR Swine Flu Panel for Detection of 2009 A (H1N1) Pandemic Influenza Virus. Journal of Clinical Microbiology. v. 49, n. 7, p. 2614-2619. 2011.
VIJAYARAGHAVAN, P. M., et al. Virological investigation of hand, foot, and mouth disease in a tertiary care center in South India. Journal of global infectious diseases, v. 4, n. 3, p. 153–161, 2012.
WELLS, AI; COYNE, CB. Enteroviruses: A Gut-Wrenching Game of Entry, Detection, and Evasion. Viruses. v.11, n.5, p. 460. 2019.
WORLD HEALTH ORGANIZATION. Polio laboratory manual 4th edition. Geneva 27, Switzerland: Department of Immunization, Vaccines and Biologicals CH-1211, World Health Organization; p. 1–167. 2004.
