Capítulo de livro publicado no livro do I CONGEB. Para acessa-lo clique aqui.
DOI: https://doi.org/10.53934/9786585062046-19
Este trabalho foi escrito por:
Davi Bento Antonucci*; Kássia Ayumi Segawa do Amaral; Adriele Rodrigues dos Santos; Isadora Oliveira Gondim; Priscilla Zwiercheczewski de Oliveira; Juliana Morini Küpper Cardoso Perseguini
*Autor correspondente (Corresponding author) – Email: [email protected]
Resumo: A celulose é o recurso natural mais abundante e um componente chave da biomassa vegetal, podendo ser uma fonte de energia econômica e ambientalmente viável e renovável. Atualmente, as celulases são produzidas comercialmente a partir de fungos, sendo seu alto custo atribuído principalmente aos substratos utilizados na produção e à lenta taxa de crescimento dos fungos. A produção das enzimas celulolíticas em hospedeiros bacterianos, utilizando a técnica do DNA recombinante, é uma estratégia para reduzir os custos da produção de enzimas. O sistema de expressão procariótico está sendo preferido em relação ao sistema de expressão eucariótico devido ao seu baixo custo e taxa de crescimento rápido. A Escherichia coli é considerada uma boa escolha porque sua expressão gênica e fisiologia são bem conhecidas, cresce em altas densidades celulares e em meios de cultura acessíveis. Dessa forma, neste artigo abordaremos informações sobre a técnica do DNA recombinante para produção mais eficaz e viável das enzimas celulolíticas.
Palavras-chave: celulase; clonagem; DNA recombinante; engenharia genética; enzimas celulolíticas
INTRODUÇÃO
As enzimas ocorrem em todos os organismos vivos e catalisam reações bioquímicas necessárias para sustentar a vida, fazem parte de uma classe altamente seletiva de proteínas que desempenham um papel vital em funções fisiológicas que variam desde a digestão de alimentos até a síntese de DNA (1). Um grupo de enzimas presente em abundância em micróbios, plantas e animais são as celulases. O sistema enzimático celulolítico é composto por três enzimas: β-1,4-endoglucanases, β-1,4-exoglucanases e β-glucosidases, que funcionam de forma coordenada para a degradação da celulose em unidades de glicose (2).
A celulose é o recurso natural mais abundante e um componente chave da biomassa vegetal, podendo ser uma fonte de energia econômica e ambientalmente viável e renovável. Porém, a utilização econômica desse recurso natural depende principalmente do ritmo de hidrólise dos resíduos lignocelulósicos para a produção dos metabólitos e biocombustíveis desejados (3) e, nesse sentido, o uso das enzimas celulolíticas se torna uma opção.
Entretanto, o custo das enzimas para converter materiais de biomassa vegetal em açúcares fermentáveis é um grande impedimento para o desenvolvimento da indústria de biocombustíveis (4,5). Atualmente, as celulases são produzidas comercialmente a partir de fungos e, embora as cepas fúngicas já tenham sofrido uma extensa melhoria, é difícil alcançar um rendimento de proteína superior a 100 mg/l usando os métodos existentes (3,6).
A produção das enzimas celulolíticas em hospedeiros bacterianos, utilizando a técnica do DNA recombinante, é uma estratégia para reduzir os custos da produção de enzimas (7,8), uma vez que o sistema bacteriano alcança a alta densidade populacional em pouco tempo e, a produção extracelular das celulases é comparativamente maior do que a do sistema fúngico (3,6,8). Isso permitiria a eliminação de enzimas não essenciais e focaria na melhoria racional de enzimas-chave necessárias para a quebra eficiente de celuloses (3,6).
Dessa forma, neste artigo abordaremos informações sobre a técnica do DNA recombinante para produção mais eficaz e viável das enzimas celulolíticas.
TÉCNICAS TRADICIONAIS DE OBTENÇÃO DA ENZIMA CELULASE
As celulases são a chave principal das biotransformações bioquímicas, porém, possuem um custo elevado para a sua produção, que se transforma em uma barreira para a comercialização dos bioprodutos. Um sistema enzimático eficiente, balanceado e de baixo custo se torna possível com a utilização dos microrganismos (9). Na literatura encontram-se relatos da utilização de vários microrganismos para a produção de celulases, dentre os quais, os mais citados são os fungos: Trichoderma reesei, Trichoderma koningii, Trichoderma lignorum, Sporotrichum pulverulentum, Penicillium funiculosum, Penicillium iriensis, Aspergillus sp, Schizophyllumm sp, Chaetomium sp e Humicola sp (10).
De toda a diversidade microbiana, os fungos filamentosos são os mais utilizados devido às suas características morfológicas. A extensão das hifas permite ao fungo a penetração no substrato de crescimento, formando uma estrutura de apoio sólida. Para isso, prefere-se a fermentação em estado sólido, que permite o acesso de nutrientes e a excreção das enzimas pelas hifas, que não são diluídas ao meio, além de simular o habitat natural dos fungos filamentosos (11,12).
A manipulação de microrganismos visando a obtenção de produtos é conhecida desde antigamente, mas é a partir da década de 1990 que ela se intensifica devido ao avanço da engenharia genética com o potencial de ampliação de escala da produção. Dois métodos são largamente utilizados, a fermentação em estado sólido e a fermentação submersa (13).
A fermentação em estado sólido possui vantagens econômicas e simplicidade de produção sobre todos os outros métodos de fermentação existentes por cultivar o microrganismo em substratos sólidos com ausência de água livre, propiciando o aproveitamento de resíduos agroindustriais (14). Também proporciona um maior rendimento da excreção enzimática, maior estabilidade das enzimas em variações de temperatura e pH com menos susceptibilidade à inibição (14).
Para a fermentação submersa, o crescimento do microrganismo ocorre em um meio propício com concentração de oxigênio controlada e permite o escalonamento da produção com facilidade por utilizar meios líquidos de cultivo (15,16). Os processos mais utilizados são a fermentação contínua, batelada alimentada e batelada simples, seguidos dos parâmetros de controle de pH, temperatura, difusão de oxigênio, formação de dióxido de carbono e taxa de alimentação do biorreator (13). Uma das desvantagens atrelada a essa técnica é o alto gasto energético associado aos sistemas de esterilização, purificação e agitação (16). Para a remoção das enzimas secretadas em um meio líquido, é necessária uma etapa de filtração ou centrifugação com consequente remoção das células suspensas, por serem enzimas extracelulares, elas ainda permanecem no meio após a remoção da biomassa, necessitando de uma etapa de concentração enzimática geralmente por filtração de membranas, sendo essa a maior dificuldade de uma fermentação submersa (13).
A maioria dos fungos apresenta um sistema celulolítico e que precisa ser induzido pela presença do substrato contendo celulose (17). Diversos resíduos apresentam celulose complexa em sua composição, além de alguns açúcares simples, o mais utilizado é o bagaço da cana-de-açúcar pela alta disponibilidade e pelo baixo custo. A Tabela 1 lista os principais resíduos disponíveis pela indústria e suas composições.
Tabela 1. Composição de alguns materiais lignocelulósicos.
| Material | Celulose (%) | Hemicelulose (%) | Lignina (%) |
| Bagaço de cana | 40 | 26 | 25 |
| Bagaço de laranja | 16 | 14 | 1 |
| Casca de amendoim | 38 | 36 | 16 |
| Casca de arroz | 36 | 20 | 19 |
| Casca de aveia | 31 | 29 | 23 |
| Palha de cana | 36 | 21 | 16 |
| Palha de milho | 36 | 28 | 29 |
| Palha de trigo | 39 | 36 | 10 |
| Palha de cevada | 44 | 27 | 7 |
| Palha de arroz | 43 | 22 | 17 |
| Palha de algodão | 42 | 12 | 16 |
| Farelo de cevada | 23 | 33 | 21 |
| Sabugo de milho | 36 | 28 | NR* |
*NR: Valores não reportados. Fonte: Adaptado de Santos (18).
Embora os fungos tenham a capacidade de melhor produção de celulase, um único fungo com todos os componentes do sistema de celulase para a hidrólise eficaz da biomassa é muito raro (19–21). Além disso, a produção comercial de celulase é realizada usando diferentes linhagens de fungos para desenvolver todas as três principais enzimas eficientes do sistema celulase (exo, endo e β-g) (19).
A celulase é uma enzima de importância industrial e subsidia cerca de 20% do mercado global de enzimas em todo o mundo. Portanto, a produção e eficiência da enzima celulase tornaram-se um dos principais pontos de atenção a serem focados em escala industrial (16).
USO DO DNA RECOMBINANTE PARA OBTENÇÃO DE CELULASES
A celulase tem sido amplamente utilizada em aplicações industriais e diversas commodities, incluindo biocombustíveis de segunda geração, alimentos e recursos bioquímicos (22,23). Estas enzimas são produzidas em larga escala usando fungos, sendo seu alto custo atribuído principalmente aos substratos utilizados na produção e à lenta taxa de crescimento dos fungos (24). Contudo, para aumentar a produção de celulase e minimizar o custo total das aplicações industriais de celulose, a produção de celulase microbiana recombinante via engenharia genética foi proposta e rapidamente desenvolvida (22).
Dentre as bactérias, Bacillus spp. representa um dos gêneros mais importantes para produção de enzimas celulolíticas e, numerosos genes de celulases foram clonados de várias espécies de Bacillus, incluindo: B. licheniformis (22,25), B. subtilis (24,26–29), B. polymyxa (30,31), B. circulans (30) e B. macerans (32). As celulases de Bacillus foram relatadas como pertencentes às famílias da glicosil hidrolase 5 (GH5) e tentativas foram feitas para clonar e expressar os genes que codificam essas enzimas em um hospedeiro heterólogo, Escherichia coli (5,29). A clonagem e superexpressão de genes de celulases em hospedeiros heterólogos podem ser benéficas para sua aplicação industrial direta (5).
Assim, os pesquisadores se concentraram no desenvolvimento de celulase recombinante usando o sistema de expressão de E. coli (24). Esta bactéria como host, não requer meio de cultura específico e cresce de forma robusta (5). Portanto, a principal vantagem da celulase recombinante é o aumento de escala no nível de produção sem o uso de substrato caro (28).
TÉCNICA DO DNA RECOMBINANTE
A tecnologia de DNA recombinante (rDNA) compreende a alteração do material genético fora de um organismo para obter características aprimoradas e desejadas em organismos vivos ou como seus produtos (33). Essa tecnologia envolve a inserção de fragmentos de DNA de diversas fontes, possuindo uma sequência gênica desejável via vetor apropriado (33,34). Esta técnica permite amplificar determinado locus, desde que apresente quantidades mínimas de DNA genômico. Esta amplificação é feita com uma polimerase extraída da bactéria Thermus aquaticus, em cerca de 20 a 30 ciclos, podendo obter-se milhões de cópias do fragmento de DNA, levando apenas algumas horas (35).
Esta técnica começou a ser desenvolvida no ano de 1970, utilizando as enzimas de restrição, que são utilizadas e permitem cortar o DNA em pontos específicos, e vetores de clonagem, como plasmídeos e genomas virais (36). As primeiras moléculas de rDNA foram geradas em 1973 por Paul Berg, Herbert Boyer, Annie Chang e Stanley Cohen da Universidade de Stanford e da Universidade da Califórnia em São Francisco (33). Em 1975, durante a “The Asilomar Conference” foi discutida a regulamentação e o uso seguro da tecnologia rDNA (33,37). E, desde meados da década de 1980, o número de produtos como hormônios, vacinas, agentes terapêuticos e ferramentas de diagnóstico tem sido desenvolvido continuamente para melhorar a saúde (33).
ETAPAS DA TÉCNICA DE DNA RECOMBINANTE
Uma vez que o microrganismo de origem (B. subtilis), o gene alvo (celulase) e o vetor de expressão (E. coli) foram identificados dá-se início à seleção dos reagentes. Inúmeros materiais podem ser adquiridos comercialmente tais como: kit de extração de DNA bacteriano, kit de purificação, kit de preparação plasmidial, kits de amplificação para a reação em cadeia de polimerase, cepas hospedeiras com marcadores de resistência, vetores de clonagem, enzimas de restrição, entre outros (22,24).
EXTRAÇÃO DO DNA TOTAL
A extração do DNA cromossômico total é a primeira etapa a ser concluída e para tal pode-se fazer uso de kits comerciais (22,24).
AMPLIFICAÇÃO DO GENE DE INTERESSE
Os primers devem permear todo o segmento de interesse incluindo os códons de iniciação e terminação. A sequência do gene alvo pode ser localizada em plataformas de banco de dados genômicos como o GenBank (gi: 39776). Caso o gene de interesse não possua sítios de restrição idênticos aos do plasmídeo é necessário, para facilitar as etapas posteriores de produção do microrganismo recombinante, adicionar ao primer a sequência correspondente a esses sítios (2,24). Este procedimento irá fusionar extremidades no amplicon que serão complementares aos sítios de restrição do DNA plasmidial do vetor de clonagem (2,24). Sendo que, após rodar a PCR (Polymerase Chain Reaction) a etapa de isolamento e purificação do amplicon deve ser conduzida.
ISOLAMENTO E PURIFICAÇÃO DO AMPLICON
Para o isolamento dos fragmentos amplificados faz-se a digestão destes pelas enzimas de restrição para os quais o amplicon e o plasmídeo possuam sítios de restrição (Tabela 2). Estas enzimas bacterianas reconhecem sequências de pares de bases específicas nas moléculas de DNA e as cortam nesses pontos. Após a digestão, faz-se uma corrida em gel de agarose a 1% para confirmação (24). Etapas de sequenciamento para análise comparativa e construção de vetor para caracterizar a produção da proteína é algo desejável (2,22).
Tabela 2. Principais enzimas de restrição, organismo de origem e sítios de clivagem.
| Nome de enzima | Fonte | Local de reconhecimento e de clivagem | Natureza das pontas cortadas |
| Eco R1 | E. coli RY13 | 5’-G|AATTC-3’ 3’-CTTAA|G-5’ | Coesiva |
| Hind III | Haemophilus influenzae Rd | 5’-A|AGCTT -3’ 3’-TTCGA|A-5’ | Coesiva |
| Bam HI | Bacillus amyloliquefaciens H | 5’-G|GATCC-3’ 3’-CCTAG|G-5’ | Coesiva |
| Alu I | Arthrobacter luteus | 5’-AG|CT -3’ 3’-TC|GA-5’ | Cega |
| Not I | Nocardia otitidis-caviarum | 5’-GC|GGCCGC -3’ 3’-CGCCGG|CG -5’ | Coesiva |
| Sau3AI | Staphylococcus aureus | 5’-|GATC -3’ 3’-CTAG|-5’ | Coesiva |
| Sma I | Serratia marcescens | 5’-CCC|GGG-3’ 3’-GGG|CCC-5’ | Cega |
Fonte: (38)
Como as enzimas de restrição clivam em regiões palindrômicas e deixam segmentos coesivos expostos, para evitar a circularização e formação de estruturas secundárias, a etapa de desfosforilação e fosforilação com o uso de fosfatase alcalina pode ser necessária para diminuir a perda de produto e fundo sujo (39,40). Enzimas como TSAP e T4 PNK são opções para esse processo.
CONSTRUÇÃO DO VETOR DE CLONAGEM
Os vetores de clonagem (Figura 1) podem ser produzidos em laboratório ou adquiridos de empresas especializadas como Novagen, Invitrogen e Promega (22,24). A construção do vetor se inicia com a adição da endonuclease utilizada para a digestão do amplicon (24) em um tubo juntamente com o inserto e o vetor. A enzima DNA ligase é adicionada para refazer as ligações fosfodiéster entre os segmentos de DNA plasmidial e amplificado, e obter o vetor recombinante (vetor de clonagem) (2).
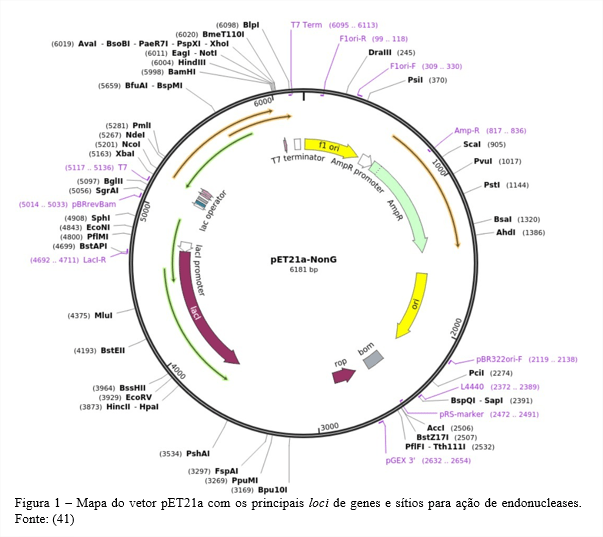
O sítio de inserção do gene exógeno deve ser analisado para se evitar a quebra da codificação de proteínas essenciais. O posicionamento do inserto após um promotor correto também é etapa que exige estudo (42).
TRANSFORMAÇÃO DO HOSPEDEIRO
A célula hospedeira ou vetor de expressão, neste caso a E. coli, pode ser adquirida ou desenvolvida em laboratório. Esta célula deve conter em seu genoma um sítio de resistência a antibióticos como a canamicina, ampicilina ou cloranfenicol para a seleção dos hospedeiros recombinantes (43).
Os vetores de clonagem são acondicionados juntamente às células hospedeiras e uma das opções de transformação bacteriana é desencadeada. Os principais métodos de transformação adotados em laboratórios são: eletroporação (2) e choque térmico.
SELEÇÃO DAS CÉLULAS TRANSFORMADAS
Após a etapa de transformação do hospedeiro é necessário proceder à seleção destes. As células transformadas são colocadas em meio com o agente antimicrobiano para o qual as células recombinantes bem-sucedidas possuem resistência e a seleção das colônias é realizada. Os clones bacterianos positivos, ou seja, com a inserção do DNA recombinante e do gene de resistência crescerão neste meio (2).
Para a confirmação do sucesso no processo de transformação pode-se realizar, a partir de uma alíquota das colônias selecionadas, uma rodada de amplificação com primers específicos e comparar, através de corrida em gel de agarose, com o controle (DNA genômico do B. subtilis) (24). Adicionalmente pode-se sequenciar o amplificado (2) e com uso de ferramentas de bioinformática alinhar e comparar com o controle.
Para a extração do DNA plasmidial pode-se optar pela lise alcalina e centrifugação. Uma vez que o DNA plasmidial possui menor peso molecular que o cromossomal, o primeiro estará no sobrenadante (44).
EXCISÃO DO GENE DE RESISTÊNCIA
A remoção dos marcadores selecionáveis, não mais necessários, pode ser benéfica, uma vez que estes limitam o empilhamento de genes adicionais em transformações subsequentes e podem representar uma carga metabólica excessiva na maquinaria celular (45) e evitar questões regulatórias (46).
EXPRESSÃO E PURIFICAÇÃO DA ENZIMA DE INTERESSE
Uma das etapas finais do processo de produção de um microrganismo recombinante é a verificação da expressão gênica, secreção da proteína alvo para o meio e o estabelecimento da produção em larga escala (2,22).
PRINCIPAIS CEPAS HOSPEDEIRAS E SUAS CARACTERÍSTICAS
As principais características para uma célula hospedeira é ter fisiologia celular bem documentada, sequências completas do genoma, informações do mecanismo de regulação, expressão gênica e propriedades de recombinação disponíveis.
Existem diversas cepas de E. Coli que são utilizadas como hospedeiros para produção de proteínas heterólogas, as mais conhecidas são a K-12 e atualmente a BL21-(DE3). A E. coli K-12 é uma das bactérias mais extensivamente estudadas, o seu genoma foi sequenciado em 1997 e consiste em um único cromossomo circular de 4.6 Mbp. Tem um histórico de uso seguro na produção de especialidades químicas e medicamentos humanos por não possuir as estruturas de superfície necessárias para a colonização eficiente do intestino humano e também carecer dos genes que codificam toxinas (47). A cepa BL21(DE3), também conhecida como células quimicamente competentes e igualmente adequada para clonar segmentos de DNA instáveis, emergiu como destaque devido seu crescimento ser mais rápido que outras linhagens (por não terem flagelos e serem imóveis) e tornou-se o hospedeiro preferido para a produção de proteínas recombinantes. O sequenciamento do genoma de cepas da linha B tem ajudado a entender a base molecular de fenótipos úteis para a síntese de proteínas heterólogas (48).
A E. coli é considerada uma boa escolha porque sua expressão gênica e fisiologia são bem conhecidas, tem rápido crescimento, cresce em altas densidades celulares e em meios de cultura acessíveis, não formam aglomerados celulares, há a possibilidade de uso de cepas com baixa atividade proteolítica, além de serem de fácil manipulação e custo-benefício. É o hospedeiro mais bem caracterizado com muitas alternativas de ferramentas de expressão e regulação disponíveis. Entretanto são incapazes de degradar polímeros de celulose devido à falta de celulases endógenas e são pobres secretores de proteínas sob condições normais de cultura devido seu envelope celular com duas membranas; as proteínas recombinantes produzidas em E. coli, inclusive na BL21(DE3), geralmente são encontradas intracelularmente na forma de corpos de inclusão insolúveis e, por serem procariotos, não podem introduzir modificações pós-tradução como pontes dissulfeto ou glicosilação (48). Vários estudos estão sendo conduzidos para inserir modificações (ancorar proteínas na membrana externa) para que a proteína heteróloga seja produzida no meio externo (22).
Os hospedeiros do gênero Bacillus são úteis para a produção de enzimas heterólogas e outras proteínas procarióticas e eucarióticas em larga escala secretando proteínas diretamente no meio de fermentação, porém tem como desvantagem possuírem vários genes de resistência a antibióticos (8,49,50). Pseudomonas fluorescens é considerada segura, porém acumula proteínas heterólogas intracelularmente como corpos de inclusão (8).
As leveduras são muito utilizadas para a produção de biocombustíveis a partir de resíduos industriais. A Saccharomyces cerevisiae já está bem documentada e Chang et al. (43) desenvolveu uma técnica de engenharia de hospedeiro para ajustar os níveis de expressão de genes heterólogos na levedura do kefir, Kluyveromyces marxianus, para obter alta eficiência celulolítica, as leveduras possuem alta eficiência secretória e podem fazer modificações pós-traducionais (49).
Os fungos são produtores e secretores eficazes de celulases devidos sua fisiologia e nicho ecológico que ocupam. A vantagem dos fungos sobre outros microrganismos industriais é sua capacidade de secretar grandes quantidades de proteínas (até 120-150 g/litro) (51). No entanto podem ter limitações como proteólise das proteínas heterólogas, produção de micotoxinas (metabólitos secundários) (8,51) e a ausência de promotores de RNA polimerase III, que pode ser contornada usando o promotor RNA polimerase II e ribozimas ou o promotor 5S rRNA (51,52).
As células vegetais estão sendo utilizadas para produzir alimentos com melhores características nutricionais (conteúdo lipídico ou de vitaminas), na otimização de absorção de nutrientes do solo, resistência a pragas e menor uso de pesticidas, resistência a estresses abióticos, entre outros. Mas são poucos os cultivares que tem informações mais detalhadas acerca do seu genoma (53,54).
As células de mamíferos também são capazes de inserir modificações pós-traducionais, entretanto necessitam de fatores mais complexos (nutrientes essenciais, fatores de crescimento, hormônios, balanço de gases etc.). O produto apresenta um custo mais elevado e os fatores limitantes descritos são baixa eficiência e controle preciso da expressão gênica. Sua aplicação é direcionada para área pesquisa médica, farmacêutica e testes de segurança, pecuária, xenotransplante de órgãos, entre outros. Exemplos de hospedeiros: células de rim de hamster, culturas celulares diversas e animais transgênicos biorreatores (55).
O sistema de expressão procariótico está sendo preferido em relação ao sistema de expressão eucariótico devido ao seu baixo custo, taxa de crescimento rápido, flexibilidade e facilidade de escala para produção de proteínas de nível mais alto que não requerem modificações pós-traducionais. As propriedades de recombinação são especialmente relevantes para a escolha de hospedeiros para propagação de bibliotecas a fim de evitar a deturpação devido ao crescimento desigual de clones (48,56). Sendo essencial localizar um “porto seguro” genômico para a inserção de genes, de modo que ele funcione corretamente e não cause risco ao hospedeiro, pois nem sempre estas células sobrevivem ao próprio gene ou à proteína expressada (48,56).
CONCLUSÃO
O sucesso da clonagem e expressão de genes que codificam celulases promove o desenvolvimento de uma estratégia de baixo custo para produzir este grupo enzimático em grande quantidade. O objetivo final de produzir uma proteína desejada em um hospedeiro heterólogo econômico é influenciado por uma variedade de fatores. No entanto, maximizar a produção de proteínas heterólogas para aplicação comercial ainda é uma arte. Uma expressão enzimática econômica e uma caracterização detalhada da enzima recombinante purificada pode ser um candidato valioso para uso em vários processos industriais, especialmente na indústria de biocombustíveis para conversão de biomassa vegetal em açúcar fermentável.
REFERENCIAS
1. Ladics GS, Sewalt V. Industrial microbial enzyme safety: What does the weight-of-evidence indicate? Regul Toxicol Pharmacol [Internet]. 2018;98(July):151–4. Available from: https://doi.org/10.1016/j.yrtph.2018.07.016
2. Liu ZL, Li HN, Song HT, Xiao WJ, Xia WC, Liu XP, et al. Construction of a trifunctional cellulase and expression in Saccharomyces cerevisiae using a fusion protein. BMC Biotechnol. 2018;18(1):1–7.
3. Wang BT, Hu S, Yu XY, Jin L, Zhu YJ, Jin FJ. Studies of cellulose and starch utilization and the regulatory mechanisms of related enzymes in Fungi. Polymers (Basel). 2020;12(3):1–17.
4. Fatma S, Hameed A, Noman M, Ahmed T, Sohail I, Shahid M, et al. Lignocellulosic Biomass: A Sustainable Bioenergy Source for Future. Protein Pept Lett. 2018;25:148–63.
5. Santos CA, Ferreira-Filho JA, O’Donovan A, Gupta VK, Tuohy MG, Souza AP. Production of a recombinant swollenin from Trichoderma harzianum in Escherichia coli and its potential synergistic role in biomass degradation. Microb Cell Fact. 2017;16(1):1–11.
6. Liu M, Yu H. Cocktail production of an endo-β-xylanase and a β-glucosidase from Trichoderma reesei QM 9414 in Escherichia coli. Biochem Eng J [Internet]. 2012;68:1–6. Available from: http://dx.doi.org/10.1016/j.bej.2012.07.003
7. Dadwal A, Sharma S, Satyanarayana T. Thermostable cellulose saccharifying microbial enzymes: Characteristics, recent advances and biotechnological applications. Int J Biol Macromol [Internet]. 2021;188(July):226–44. Available from: https://doi.org/10.1016/j.ijbiomac.2021.08.024
8. Olempska-Beer ZS, Merker RI, Ditto MD, DiNovi MJ. Food-processing enzymes from recombinant microorganisms-a review. Regul Toxicol Pharmacol. 2006;45(2):144–58.
9. Florencio C. Microrganismos Produtores de Celulases : Seleção de Isolados de Trichoderma spp. [Internet]. São Carlos: UFSCar. Universidade Federal de São Carlos; 2011. Available from: https://repositorio.ufscar.br/bitstream/handle/ufscar/6979/3718.pdf?sequence=1&isAllowed=y
10. da Silva R, Yim DK, Park YK. Application of thermostable xylanases from Humicola sp. for pulp improvement. J Ferment Bioeng. 1994;77(1):109–11.
11. Hölker U, Höfer M, Lenz J. Biotechnological advantages of laboratory-scale solid-state fermentation with fungi. Appl Microbiol Biotechnol. 2004;64(2):175–86.
12. Raimbault M. General and microbiological aspects of solid substrate fermentation. Electron J Biotechnol. 1998;1(3):114–40.
13. Hargreaves PI. Bioprospecção de novas celulases de fungos provenientes da floresta amazônica e otimização de sua produção sobre celulignina de bagaço de cana [Internet]. Rio de Janeiro. Universidade Federal do Rio de Janeiro; 2008. Available from: http://www.tpqb.eq.ufrj.br/download/bioprospeccao-de-novas-celulases-de-fungos-filamentosos.pdf
14. Singhania RR, Sukumaran RK, Patel AK, Larroche C, Pandey A. Advancement and comparative profiles in the production technologies using solid-state and submerged fermentation for microbial cellulases. Enzyme Microb Technol [Internet]. 2010;46(7):541–9. Available from: http://dx.doi.org/10.1016/j.enzmictec.2010.03.010
15. Tavares APM. Produção de lacase para potencial aplicação como oxidante na indústria papeleira [Internet]. Universidade de Aveiro; 2006. Available from: https://ria.ua.pt/bitstream/10773/3177/1/2007001374.pdf
16. Passos D de F, Pereira N, Castro AM de. A comparative review of recent advances in cellulases production by Aspergillus, Penicillium and Trichoderma strains and their use for lignocellulose deconstruction. Curr Opin Green Sustain Chem [Internet]. 2018;14:60–6. Available from: https://doi.org/10.1016/j.cogsc.2018.06.003
17. Zúñiga UFR. Desenvolvimento De Um Bioprocesso Para Produção De Celulases Específicas Na Cadeia Produtiva Do Etanol De Segunda Geração [Internet]. Escola de Engenharia de São Carlos, Universidade de São Paulo. Universidade de São Paulo; 2010. Available from: https://www.teses.usp.br/teses/disponiveis/18/18139/tde-15032011-133333/publico/TeseUrsulaFabiolaRodriguezZuniga.pdf
18. Santos ADS. Produção, concentração e caracterização parcial de extrato celulolítico produzido por Linhagem Fúngica Mutante [Internet]. Instituto De Tecnologia. Universidade federal rural do Rio de Janeiro; 2014. Available from: https://ainfo.cnptia.embrapa.br/digital/bitstream/item/48104/1/Dissertacao-Alex-Monica.pdf
19. Srivastava N, Rawat R, Singh Oberoi H, Ramteke PW. A review on fuel ethanol production from lignocellulosic biomass. Int J Green Energy. 2015;12(9):949–60.
20. Srivastava N, Srivastava M, Mishra PK, Gupta VK, Molina G, Rodriguez-Couto S, et al. Applications of fungal cellulases in biofuel production: Advances and limitations. Renew Sustain Energy Rev [Internet]. 2018;82(August 2017):2379–86. Available from: https://doi.org/10.1016/j.rser.2017.08.074
21. Kumar S, Sarma AK. Recent Advances in Bioenergy Edited by. Vol. I. 2013.
22. Kim D, Ku S. Bacillus cellulase molecular cloning, expression, and surface display on the outer membrane of Escherichia coli. Molecules. 2018;23(2).
23. Juturu V, Wu JC. Microbial cellulases: Engineering, production and applications. Renew Sustain Energy Rev. 2014;33:188–203.
24. Vadala BS, Deshpande S, Apte-Deshpande A. Soluble expression of recombinant active cellulase in E.coli using B.subtilis (natto strain) cellulase gene. J Genet Eng Biotechnol. 2021;19(1).
25. Lloberas J, Perez‐Pons JA, Querol E. Molecular cloning, expression and nucleotide sequence of the endo‐β‐1,3‐1,4‐d‐glucanase gene from Bacillus licheniformis Predictive structural analyses of the encoded polypeptide. Eur J Biochem. 1991;197(2):337–43.
26. Pandey A, Soccol CR, Nigam P, Soccol VT. Biotechnological potential of agro-industrial residues. I: Sugarcane bagasse. Bioresour Technol. 2000;74(1):69–80.
27. Wei KSC, Teoh TC, Koshy P, Salmah I, Zainudin A. Cloning, expression and characterization of the endoglucanase gene from bacillus subtilis UMC7 isolated from the gut of the indigenous termite macrotermes malaccensis in escherichia coli. Electron J Biotechnol. 2015;18(2):103–9.
28. Zafar M, Ahmed S, Imran M, Khan M, Jamil A. Recombinant expression and characterization of a novel endoglucanase from Bacillus subtilis in Escherichia coli. Mol Biol Rep. 2014;41(5):3295–302.
29. Li W, Zhang WW, Yang MM, Chen YL. Cloning of the thermostable cellulase gene from newly isolated Bacillus subtilis and its expression in Escherichia coli. Mol Biotechnol. 2008;40(2):195–201.
30. Baird SD, Johnson DA, Seligy VL. Molecular cloning, expression, and characterization of endo-β-1,4-glucanase genes from Bacillus polymyxa and Bacillus circulans. J Bacteriol. 1990;172(3):1576–86.
31. Gosalbes MJ, Perez-Gonzalez JA, Gonzalez R, Navarro A. Two β-glycanase genes are clustered in bacillus polymyxa: Molecular cloning, expression, and sequence analysis of genes encoding a xylanase and an endo-β-(1,3)-(1,4)-glucanase. J Bacteriol. 1991;173(23):7705–10.
32. Heng NCK, Jenkinson HF, Tannock GW. Cloning and expression of an endo-1,3-1,4-β-glucanase Gene from Bacillus macerans in Lactobacillus reuteri. Appl Environ Microbiol. 1997;63(8):3336–40.
33. Khan S, Ullah MW, Siddique R, Nabi G, Manan S, Yousaf M, et al. Role of recombinant DNA technology to improve life. Int J Genomics. 2016;2016.
34. Fauser BCJM, Mannaerts MJL, Devroey P, Leader A, Boime I, Baird DT. Advances in recombinant DNA technology: Corifollitropin alfa, a hybrid molecule with sustained follicle-stimulating activity and reduced injection frequency. Hum Reprod Update. 2009;15(3):309–21.
35. Alberto J, Candeias N. Novos aspectos da saúde pública/recent aspects of public health A Engenharia Genética. Rev Saúde Pública [Internet]. 1991;25(1):3–10. Available from: http://www.scielo.br/pdf/rsp/v25n1/02.pdf
36. Lira P de. Aplicação da tecnologia do DNA recombinante na saúde: riscos e benefícios [Internet]. Faculdade de ciências da educação e saúde; 2016. Available from: https://repositorio.uniceub.br/jspui/handle/235/9023
37. Griffin JA. Recombinant DNA — Potential for Gene Therapy. Am J Med Sci [Internet]. 1985;289(3):98–106. Available from: https://www.sciencedirect.com/science/article/pii/S0002962915371597
38. Roberts MAJ. Recombinant DNA technology and DNA sequencing. Essays Biochem. 2019;63(4):457–68.
39. Chen F, Li Y ya, Yu Y li, Dai J, Huang J ling, Lin J. Simplified plasmid cloning with a universal MCS design and bacterial in vivo assembly. BMC Biotechnol. 2021;21(1):1–13.
40. Green MR, Sambrook J. Dephosphorylation of DNA Fragments with Alkaline Phosphatase. Cold Spring Harb Protoc. 2020;2020(8):365–9.
41. SnapGene software [Internet]. San Diego; [cited 2022 Aug 29]. Available from: www.snapgene.com
42. Hoseini SS, Sauer MG. Molecular cloning using polymerase chain reaction, an educational guide for cellular engineering. J Biol Eng. 2015;9(1).
43. Chang JJ, Lin YJ, Lay CH, Thia C, Wu YC, Hou YH, et al. Constructing a cellulosic yeast host with an efficient cellulase cocktail. Biotechnol Bioeng. 2018;115(3):751–61.
44. Delaney S, Murphy R, Walsh F. A comparison of methods for the extraction of plasmids capable of conferring antibiotic resistance in a human pathogen from complex broiler cecal samples. Front Microbiol. 2018;9(AUG).
45. Sheva M, Hanania U, Ariel T, Turbovski A, Rathod VKR, Oz D, et al. Sequential Genome Editing and Induced Excision of the Transgene in N. tabacum BY2 Cells. Front Plant Sci. 2020;11(November):1–8.
46. Tan J, Wang Y, Chen S, Lin Z, Zhao Y, Xue Y, et al. An Efficient Marker Gene Excision Strategy Based on CRISPR/Cas9-Mediated Homology-Directed Repair in Rice. Int J Mol Sci. 2022;23(3).
47. Cronan JE. Escherichia colias an Experimental Organism [Internet]. John Wiley & Sons, Ltd; 2005. Available from: https://search.ebscohost.com/login.aspx?direct=true&db=edsair&AN=edsair.doi.dedup…..c4aa070d153c6cdee9b9aa5199766b4b&lang=pt-br&site=eds-live&scope=site
48. Rosano GL, Morales ES, Ceccarelli EA. New tools for recombinant protein production in Escherichia coli: A 5-year update. Protein Sci. 2019;28(8):1412–22.
49. Giovannoni M, Gramegna G, Benedetti M, Mattei B. Industrial Use of Cell Wall Degrading Enzymes: The Fine Line Between Production Strategy and Economic Feasibility. Front Bioeng Biotechnol. 2020;8(April):1–20.
50. Krüger A, Welsch N, Dürwald A, Brundiek H, Wardenga R. A host ‑ vector toolbox for improved secretory protein overproduction in Bacillus subtilis. Appl Microbiol Biotechnol [Internet]. 2022;9:5137–51. Available from: https://doi.org/10.1007/s00253-022-12062-2
51. Rozhkova AM, Kislitsin VY. CRISPR/Cas Genome Editing in Filamentous Fungi. Biochem. 2021; 86:S120–39.
52. Balabanova LA, Shkryl YN, Slepchenko L V., Yugay YA, Gorpenchenko TY, Kirichuk NN, et al. Development of host strains and vector system for an efficient genetic transformation of filamentous fungi. Plasmid [Internet]. 2019;101(August 2018):1–9. Available from: https://doi.org/10.1016/j.plasmid.2018.11.002
53. Ceasar SA, Maharajan T, Hillary VE, Ajeesh Krishna TP. Insights to improve the plant nutrient transport by CRISPR/Cas system. Biotechnol Adv [Internet]. 2022;59(April):107963. Available from: https://doi.org/10.1016/j.biotechadv.2022.107963
54. Li J, Scarano A, Gonzalez NM, D’Orso F, Yue Y, Nemeth K, et al. Biofortified tomatoes provide a new route to vitamin D sufficiency. Nat Plants. 2022;8(June).
55. Bihon Asfaw A, Assefa A. Animal transgenesis technology: A review. Cogent Food Agric [Internet]. 2019;5(1). Available from: https://doi.org/10.1080/23311932.2019.1686802
56. Sanal MG, Vats N, Venugopal SK, Taneja P, Sarin SK. Cloning of human ABCB11 gene in E. coli required the removal of an intragenic Pribnow-Schaller Box before it’s Insertion into genomic safe harbor AAVS1 site using CRISPR-Cas9. F1000Research. 2020;9:1–17.


