
Capítulo de livro publicado no livro do I CONGEB. Para acessa-lo clique aqui.
DOI: https://doi.org/10.53934/9786585062046-20
Este trabalho foi escrito por:
Nádia Koide Albuquerque*; Carine Fortes Aragão; Sandro Patroca da Silva; Ana Cecília Ribeiro Cruz
*Autor correspondente (Corresponding author) – Email:[email protected]
Resumo: Os morcegos têm sido associados a uma série de doenças virais zoonóticas, como as causadas pelas famílias Filoviridae, Paramyxoviridae, Rhabdoviridae e Coronaviridae, que exemplificam a fatalidade das doenças transmitidas. Com os avanços da biologia molecular, a metagenômica permite sequenciar o material genético de todos os microrganismos presentes em uma amostra, que podem compreender vírus, bactérias ou organismos eucariotos, sem necessidade de cultivo celular, isso amplia a caracterização de muitas espécies de microrganismos, inclusive aquelas abrigadas por morcegos. O objetivo deste trabalho foi investigar o viroma em amostras de morcegos da espécie Desmodus rotundus obtidos em um município no Pará. A detecção do viroma em importantes reservatórios auxilia na vigilância epidemiológica e são importantes para compreender as possíveis contribuições à resistência peculiar da espécie, seu desempenho como hospedeiro e questões evolutivas. Foram sequenciados fragmentos de pulmão no NextSeq 500 (Illumina) e os dados obtidos passaram por análises bioinformáticas. Os resultados mostram a presença de Desmodus rotundus endogenous retrovírus, que são de grande importância para estudos filogenéticos que abordam sobre a evolução das espécies e pode inferir, em estudos posteriores, como a presença desses elementos endógenos se relaciona com questões imunológicas peculiares dos morcegos.
Palavras–chave: metagenômica; morcegos; Desmodus rotundus; Desmodus rotundus endogenous retrovírus; bioinformática.
INTRODUÇÃO
Os morcegos são mamíferos silvestres da ordem Chiroptera e estão divididos em duas subordens: Megachiroptera que podem ser encontradas em regiões da África, Índia, Ásia e Oceania e Microchiroptera, encontrada em várias localidades ao longo do globo, excluindo zonas polares e algumas ilhas oceânicas. Ao todo possuem 21 famílias, 227 gêneros e 1.386 espécies. É a segunda ordem entre os mamíferos com maior diversidade de espécies, ficando atrás somente da ordem Rodentia (1,2).
O Norte e Nordeste do Brasil possuem grande diversidade de morcegos. O Pará é o estado do Norte com maior número de espécies registradas, sendo Desmodus rotundus e Carollia perspicillata os mais capturados em estudos de campo que retratam a quiropterofauna da região. A Amazônia brasileira possui em torno de 146 espécies distribuídas em 64 gêneros (3,4,5). São os únicos mamíferos com capacidade de voo, organizam-se em grupos diversificados e dispõe de características comportamentais particulares, entre elas o hábito noturno, além de contribuírem diretamente no equilíbrio do ecossistema, realizando predação de insetos, polinização e dispersão de sementes (1).
Entretanto, os morcegos têm sido associados a uma série de doenças virais zoonóticas. Essa relação ocorre por inúmeros fatores, entre eles a grande diversidade de espécies, o tamanho da colônia, longevidade, capacidade migratória e imunologia única que contribuem para a transmissão intraespecífica e interespecífica de patógenos (6,7). Estão entre as principais fontes de novas cepas virais, provavelmente, devido sua coevolução com tais vírus que forneceu oportunidade de manutenção (8,9).
Os morcegos abrigam a maior proporção de vírus zoonóticos, o que explica a maioria dos eventos de spillover para humanos. O spillover é o transbordamento de um vírus de uma espécie para outra que requer oportunidade ecológica para contato, compatibilidade celular entre vírus e o hospedeiro, além de resposta imune permissiva. Há eventos de spillover direto de morcegos para humanos e eventos indiretos, que envolvem hospedeiros intermediários (10,11).
As epidemias causadas por vírus das famílias Filoviridae, Paramyxoviridae, Rhabdoviridae e Coronaviridae exemplificam a fatalidade das doenças transmitidas, mas apesar de abrigarem grande diversidade viral, na maioria das vezes os morcegos não apresentam sintomatologia. Diante disso, algumas hipóteses imunológicas são propostas. A ausência de componentes do sistema imunológico que reconhecem o material genético de agentes estranhos, como a família de genes PYHIN, evitam a ativação do inflamassoma e os danos provocados pela infecção (12).
O estresse oxidativo é alto durante atividades metabólicas, entretanto, nos morcegos a liberação de espécies reativas de oxigênio (ROS) é limitada devido adaptações mitocondriais que minimizam o estresse durante o voo, o que pode diminuir a patogênese. Os morcegos ainda podem recrutar antioxidantes enzimáticos para eliminar o ROS eficientemente evitando assim danos oxidativos ao DNA mitocondrial e seus impactos negativos na longevidade (12,13). Em algumas espécies de morcegos a mutação no gene STING, estimulador de interferon (IFN), diminui a expressão do INF e pode permitir a replicação viral. Entretanto, apesar da replicação, a demora para recrutar linfócitos T e B após a infecção é uma estratégia evolutiva para impedir a resposta imunopatológica (13,14).
Além disso, a existência de elementos virais endógenos (EVE) é mais uma das hipóteses para a imunidade dos morcegos frente às infecções virais. Um EVE é integrado a uma célula germinativa após infecção, tornando-se parte do material genético do hospedeiro, portanto reconhece-o como próprio. Algumas regiões no genoma dos morcegos que contém esses elementos podem ter propriedades que inibem a produção de citocinas, fazendo com que sejam assintomáticos (13,14).
Atualmente, com os avanços da biologia molecular, algumas abordagens conseguem sequenciar o material genético de todos os microrganismos presentes em uma amostra, que podem compreender vírus, bactérias ou organismos eucariotos, sem necessidade de cultivo celular, permitindo ampliar a caracterização de muitas espécies de microrganismos, visto que a maioria não é cultivável. Tal vantagem proporcionou mais estudos acerca do microbioma de importantes hospedeiros (15,16).
Os vírus são capazes de infectar espécies de todos os domínios de vida existentes e através da metagenômica é possível detectar o viroma de diferentes táxons. A metagenômica possibilita uma visão mais ampla, tirando o foco dos principais causadores de doenças. Os ERVs estão presentes no material genético da maioria dos vertebrados devido infecções retrovirais acumuladas ao longo da sua história evolutiva, cerca de 10% do genoma é formado por ERVs (17).
O objetivo deste trabalho foi investigar o viroma em amostras de morcegos da espécie Desmodus rotundus obtidos em um município no Pará. A detecção do viroma em importantes reservatórios auxilia na vigilância epidemiológica e são importantes para compreender as possíveis contribuições à resistência peculiar da espécie, seu desempenho como hospedeiro e questões evolutivas. A região amazônica possui grande parte da diversidade de espécies de morcegos do Brasil, mas ainda necessita de mais relatos sobre o microbioma que eles carregam nessa área. A pandemia atual aumentou o alerta sobre a relevância de identificar novos vírus carregados por morcegos e relacioná-los com outros fatores (18).
MATERIAL E MÉTODOS
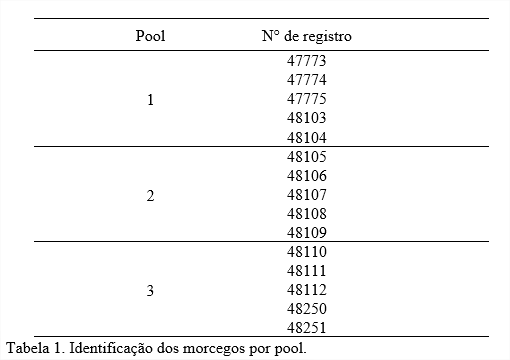
As amostras deste estudo foram obtidas de 15 morcegos, Desmodus rotundus, provenientes do município de Melgaço, localizado no arquipélago do Marajó, Pará (Figura 1), que chegaram ao Instituto Evandro Chagas em 2019 em status de óbito e foram submetidos ao teste de imunofluorescência direta para Raiva, porém todos foram negativos. Na necropsia foram coletados fragmentos de pulmão e separados em 3 pools, contendo 5 fragmentos em cada (Tabela 1) e armazenados em freezer -70 °C.
Os fragmentos das vísceras foram colocados em microtubos com capacidade de 2 mL, estéreis e livres de DNase e RNase e identificados por origem, víscera e número de registro dos animais. Foi adicionado 1 mL de Trizol e uma esfera de tungstênio de 5 mm de diâmetro para maceração no TissueLyser II (Qiagen) a 25 Hz por 1 minuto.
A extração e purificação do RNA total foi realizada pelo método Trizol combinado com o kit PureLink RNA Mini Kit (Thermo Fisher) e quantificado com Qubit® 2.0 Fluorometer (Invitrogen) utilizando o kit Qubit® RNA HS Assay. Posteriormente, foi verificada a qualidade dos fragmentos de RNA pelo Bioanalyzer 2100 (Agilent Technologies) com o kit Agilent RNA 6000 Pico, seguindo os protocolos dos fabricantes.
Para a síntese de DNA complementar (cDNA) foram utilizados 2 kits, para a 1° fita o SuperScript VILO MasterMix (Invitrogen) e para a 2° fita o NEBNext mRNA Second Strand Synthesis Module (New England Biolabs).
O cDNA foi purificado com o auxílio de esferas magnéticas AMPure bead e utilizado na preparação da biblioteca genômica pelo kit Nextera XT DNA Library Preparation (Illumina). A quantificação de moléculas de cDNA foi estimada com auxílio do equipamento Qubit® 2.0 Fluorometer e kit Qubit® dsDNA HS Assay. O tamanho dos fragmentos produzidos na biblioteca foi observado no Bioanalyzer 2100 (Agilent Technologies) através do kit High Sensitivity DNA Analysis (Agilent Technologies). A partir disso, foi realizado o sequenciamento pelo equipamento NextSeq 500 com o kit NextSeq 500/550 High Output v2.5 (300 ciclos) (Illumina) seguindo as instruções do fabricante.
Os dados obtidos do sequenciador foram transformados para extensão .fastq pelo programa bcl2fastq2 e resultou nos arquivos R1 e R2 para cada amostra. A qualidade dos dados brutos foi visualiza no Fastp e posteriormente filtrada para remover leituras curtas de baixa qualidade e bases indeterminadas, o Bowtie2 foi utilizado para mapear as leituras contra genoma do hospedeiro, Desmodus rotundus (genoma referência: ASM294091V2 Feb. 2018). O SortMeRNA v.2.1 realizou a remoção de RNA ribossomal. As sequências foram novamente visualizadas no Fastp para atestar a qualidade.
As leituras passaram por alinhamento no programa DIAMOND para comparação com o banco de sequências não redundantes (NR) do NCBI e o percentual correspondente a cada agente viral foi visualizado no Krona. O programa Kraken fez as classificações taxonômicas e Pavian permitiu visualizá-las em tabelas, ambos os programas oferecem resultados rápidos e de alta sensibilidade.
O SPAdes realizou a montagem De Novo das leituras através da seleção automática de k-mers21,33,55,77 baseado no comprimento de 150pb das sequências. Foi utilizado também o MegaHit, que faz montagem com k-mers 21,41,61,81,99. Após isso, os arquivos de contigs foram inspecionados no Megan v.6.23.2 para avaliar as famílias mais abundantes e os contigs selecionados passaram por edição, mapeamento por referência no Geneious v.9.1.8 e para alinhamento utilizamos ClustalW, ferramenta incluída no Geneious v.9.1.8.
RESULTADOS E DISCUSSÃO
A área de estudo foi selecionada em razão da diversidade de espécies quiropteros em sua extensão, como relatados por Marques-Aguiare et al. (25). Além do histórico de surtos de doenças reservadas em morcegos na região que podem ter transmissão facilitada devido à fatores socioeconômicos locais (26,27). Apesar de haver grande quantidade de morcegos dessa região analisados pelo IEC para vigilância epidemiológica da Raiva, a maioria apresenta diagnóstico negativo na imunofluorescência direta e prova biológica, o que pode indicar morte por outras causas, inclusive fatores virais. Isso mostra a necessidade de mais estudos em localidades específicas da Amazônia, além de investigação através de outras técnicas laboratoriais.
Foram obtidas 84.404.720 leituras brutas, sendo a menor quantidade para vírus, de acordo com os programas de classificação taxonômica (Tabela 1) e após passar por filtragem, o número de sequências reduziu significativamente. As sequências que apresentaram homologia com sequências do banco NR foram inspecionadas no MEGAN6 e o total de leituras e contigs obtidos foram 22.891 e 9.424 respectivamente. A redução das leituras já é algo esperado nas análises de bioinformática, a diminuição com do Bowtie2 elimina as sequências referentes ao hospedeiro, facilitando a montagem de contigs para vírus (19).
| Amostras | N° de leituras brutas | Leituras classificadas (%) | Leituras microbianas (%) | Leituras para vírus (%) |
| Pool 1 | 33.955.233 | 69.50% | 61.50% | 0.16% |
| Pool 2 | 22.816.122 | 63.10% | 53.70% | 0.17% |
| Pool 3 | 27.633.365 | 60.80% | 51.50% | 0.18% |
Tabela 2. Leituras brutas e percentuais obtidos antes da filtragem pelos sofwares Kraken e Pavian.
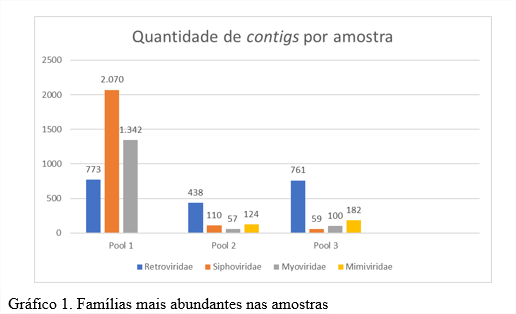
As famílias mais abundantes em leituras e contigs nos pools foram Siphoviridae, Myoviridae, Retroviridae e Mimiviridae (Gráfico 1), com destaque para Retroviridae que possui vírus que infectam vertebrados. A detecção do viroma em animais conhecidos como importantes reservatórios auxilia no monitoramento de vírus com potencial patogênico para humanos. De acordo com Van Brussel e Holmes (20), durante os últimos anos, foram detectados vários vírus em morcegos por meio do Sequenciamento de Nova Geração (NGS), cerca de 30% de todas as sequências virais relacionadas a morcegos existentes no GenBank foram obtidas através da metagenômica, sendo a maioria para vírus de RNA, que possuem maior facilidade de mutação, o que pode aumentar o grau de patogenicidade e produzir efeitos mais graves e fatais.
Os contigs montados para a família Retroviridae são referentes ao retrovírus endógeno (ERV) Desmodus rotundus endogenous retrovirus (DrERV). Os ERVs estão normalmente presentes no genoma dos vertebrados, provavelmente, devido a integração do provírus de um retrovírus exógeno no ancestral comum que se fixou no material genético antes da divergência das espécies. Portanto, são linhagens virais antigas em táxons de mamíferos e por meio delas é possível compreender sobre questões evolutivas da espécie (21).
Os retrovírus são transmitidos principalmente por contato com sangue, sendo assim, os retrovírus endógenos de morcegos hematófagos teriam facilmente saltado de uma espécie para outra. O DrERV é um provírus de baixo número de cópias que possui características estruturais betaretrovirais do tipo D. Os genes pol e env são elementos centrais que possuem vários códons de parada e espera-se que produzam produtos gênicos truncados (22).
Sequências relacionadas ao DrERV presentes na espécie Carollia perspicillata descrito por Baillie et al. (23) e a ausência em Diphylla ecaudata, outra espécie de morcego hematófago da família Phyllostomidae, sugere que o DrERV foi adquirido por infecções independentes após a divergência de espécies. Bergner et al. (24) também relata fatores ecológicos que podem influenciar a composição da comunidade viral em morcegos hematófagos, como diversidade de outras espécies animais para oferecer oportunidades de transmissão interespecífica e idade, visto que, os adultos acumulam mais infecções ao longo da vida e os juvenis desempenham papel importante na dinâmica viral devido à maior suscetibilidade.
Nos pools foram selecionados contigs do gênero Betaretrovirus já que apresentaram maior quantidade de nucleotídeos (nt) e maior identidade com uma região que codifica o gene pol de Desmodus rotundus endogenous retrovirus (DrERV) (GenBank ID AJR27940.1). Porém, no mapeamento e edição não foi possível estender de forma satisfatória as sequências. Para dar continuidade às análises, foi utilizada outra sequência completa como referência (MH648003) obtida em estudo anterior de Franco-Filho et al. (24) com morcegos da mesma espécie na Amazônia. O mapeamento contra os contigs de ambos os montadores resultou em sequências completas de DrERV, apesar de possuir algumas regiões com variabilidade genética (mutação de transição), são referentes a todos os genes deste retrovírus ou parte deles. O tamanho das sequências obtidas pode ser visualizado na Tabela 2.
| Amostras | Tamanho (nucleotídeos) |
| Pool 1 | 8.309 nt |
| Pool 2 | 10.713 nt |
| Pool 3 | 8.364 nt |
Tabela 2. Tamanho das sequências obtidas através do mapeamento dos contigs contra sequência de referência (n° de acesso MH648003) no NCBI.
As sequências de estudo alcançaram tamanho aproximado com a referência de Franco-Filho (24), o que pode inferir que DrEVR está disseminado nos D. rotundus de várias regiões amazônicas. O pulmão foi utilizado como amostra devido a possibilidade de infecções por via aerógena e visto que é um órgão altamente vascularizado, permitindo facilmente a replicação viral de cepas já conhecidas. Geralmente, amostras de urina, saliva e fezes são utilizadas com maior frequência nos estudos, pois minimizam o impacto nas colônias, além de representarem a possível via de transmissão de microrganismos, no entanto, trabalhar somente com essas amostras pode falhar na detecção de vírus associados à órgãos específicos ou sofrer interferência do ambiente nos resultados (11, 20).
CONCLUSÕES
A espécie D. rotundus está amplamente distribuída em áreas da zona rural com presença de animais domésticos de produção, devido à disponibilidade de alimento, uma vez que são hematófagos, este fator é um alerta para que a espécie seja mais investigada acerca dos microrganismos que pode armazenar. Porém, outros vírus não patogênicos, como os ERVs, são de grande importância para estudos filogenéticos que abordam sobre a evolução das espécies e pode inferir, em estudos posteriores, como a presença desses elementos endógenos se relaciona com questões imunológicas peculiares dos morcegos.
Os dados obtidos ainda podem ser profundamente analisados para avaliação de outros microrganismos presentes nos tecidos, como bactérias e fungos, complementando o presente estudo.
AGRADECIMENTOS
Este trabalho faz parte de um projeto maior envolvendo alunos e doutores do Instituto Evandro Chagas (IEC). Agradeço em especial a Seção de Arbovirologia e Febres Hemorrágicas (SAARB) que disponibilizou estrutura laboratorial, kits e equipamentos de excelência para construção destes resultados.
REFERÊNCIAS
- Reis NR, Peracchi AL, Pedro WA, Lima IP. Morcegos do Brasil. Universidade Estadual de Londrina. 2007.
- Burgin CJ, Colella JP, Kahn PL, Upham NS. How many species of mammals are there? Journal of Mammalogy. 2018; 99 (1): 1-14.
- Bernard E, Tavares VC, Sampaio E. Compilação atualizada das espécies de morcegos (Chiroptera) para a Amazônia Brasileira. Biota Neotropica. 2011; 11(1): 35-46.
- Cajaiba RL. Morcegos (Mammalia, Chiroptera) em cavernas no município de Uruará, Pará, norte do Brasil. Biota Amazônia. 2014; 4 (1): 81-86.
- Delgado‐Jaramillo M, Aguiar LM, Machado Rb, Bernard E. Assessing the distribution of a species‐rich group in a continental‐sized megadiverse country: Bats in Brazil. Diversity and Distributions. 2020; 26 (5): 632-643.
- Klimpel S, Mehlhorn H. Bats (Chiroptera) as vectors of diseases and parasites. Springer-Verlag Berlin Heidelberg.2016.
- Mandl JN, Schneider C, Schneider DS, Baker ML. Going to Bat(s) for Studies of Disease Tolerance. Frontiers in Immunology. 2018; 20(9):2112.
- Luis AD, Hayman DT, O’shea TJ, Cryan PM, Gilbert AT; Pulliam JR, Mills JN, Timonin ME, Willis CK, Cunningham AA, Fooks AR, Rupprecht CE, Wood JL, Webb CT. A comparison of bats and rodents as reservoirs of zoonotic viruses: are bats special? Proceedings of Royal Biology Society. 2013; 280 (1756): 20122753-20122753.
- Recht J, Schuenemann VJ, Sánchez-Villagra MR. Host Diversity and Origin of Zoonoses: The Ancient and the New. Animals. 2020; 10 (9):1672.
- Olival K, Hosseini P, Zambrana-Torrelio C. et al. Host and viral traits predict zoonotic spillover from mammals. Nature. 2017; 546 (7660): 646-650.
- Letko M, Seifert SN, Olival KJ, Plowright RK, Munster VJ. Bat-borne virus diversity, spillover and emergence. Natural Reviews of Microbiology. 2020; 18(8): 461- 471.
- Menachery VD, Graham RL, Baric RS. Jumping species – a mechanism for coronavirus persistence and survival. Current Opinion in Virology. 2017; 23: 1-7.
- Brook CE, Dobson AP. Bats as ‘special’ reservoirs for emerging zoonotic pathogens. Trends in Microbiology. 2015; 23 (3):172-180.
- Skirmuntt EC, Escalera-Zamudio M, Teeling EC, Smith A, Katzourakis A. The Potential Role of Endogenous Viral Elements in the Evolution of Bats as Reservoirs for Zoonotic Viruses. Annual Reviews of Virology. 2020; 7 (1):103-119.
- Radford AD, Chapman D, Dixon L, Chantrey J, Darby AC, Hall N. Application of next-generation sequencing technologies in virology. Journal of General Virology. 2012; 93(9): 1853-1868.
- Sudarikov K, Tyakht A, Alexeev D. Methods for the metagenomic data visualization and analysis. Current issues in molecular biology. 2017; 24(1): 37-58.
- Harvey E, Holmes EC. Diversity And Evolution Of The Animal Virome. Nature Reviews Microbiology. 2022; 20(6):321-334.
- Hayward JA, Tachedjian M, Cui J, Field H, Holmes EC, Wang LF, Tachedjian G. Identification of diverse full-length endogenous betaretroviruses in megabats and microbats. Retrovirology. 2013; 10 (35).
- Hardmeier I, Aeberhard N, Qi W, Schoenbaechler K, Kraettli H, Hatt J-M, et al. Metagenomic analysis of fecal and tissue samples from 18 endemic bat species in Switzerland revealed a diverse virus composition including potentially zoonotic viruses. PLoS ONE. 2021; 16(6): e0252534.
- Van Brussel K, Holmes EC. Zoonotic disease and virome diversity in bats. Current Opinion in Virology. 2021; 52:192-202.
- Hayward JA, Tachedjian M, Cui J, Field H, Holmes EC, Wang LF, Tachedjian G. Identification of diverse full-length endogenous betaretroviruses in megabats and microbats. 2013: Retrovirology 10: 35.
- Escalera-Zamudio M, Mendoza ML, Heeger F, Loza-Rubio E, Rojas-Anaya E, Méndez-Ojeda ML, Taboada B, Mazzoni CJ, Arias CF, Greenwood AD. A novel endogenous betaretrovirus in the common vampire bat (Desmodus rotundus) suggests multiple independent infection and cross-species transmission events. J Virology. 2015;89(9):5180-4.
- Baillie GJ, van de Lagemaat LN, Baust C, Mager DL. Multiple groups of endogenous betaretroviruses in mice, rats, and other mammals. Journal of Virology. 200478:5784–5798.
- Franco Filho LC, Barata RR, Cardoso JF, Massafra JMV, Lemos PDS, Casseb LMN, Cruz ACR, Nunes MRT. Metagenomic Analysis of Samples from Three Bat Species Collected in the Amazon Rain Forest. Microbiology Resourse Announcments. 2019; 8 (2):e01422- 18.
- Marques-Aguiare AS, Melo CCS, Aguiar GFS, Queiróz JAL. Levantamento preliminar da mastofauna da região de Anajás-Muaná, Ilha de Marajó, Pará, Brasil. Revista Brasileira de Zoologia. 2002; 13 (3): 841-854.
- Brasil. Ministério Da Saúde. Secretaria De Vigilância Em Saúde. Surto De Raiva Humana Transmitida Por Morcegos No Município De Portel-Pará. Boletim Eletrônico. 2004; 4 (6):1-5.
- Instituto Evandro Chagas (IEC). IEC Confirma 3 Mortes Por Raiva Humana Em Melgaço Em 2018. [acesso em 16 de Jul 2022]. Disponível em: https://www.iec.gov.br/wpcontent/uploads/2018/05/23maio2018_diario_do_para.pdf.
Previous

