OTIMIZAÇÃO, PADRONIZAÇÃO E CARACTERIZAÇÃO DA BIBLIOTECA DE ANTÍGENOS DE CORONAVÍRUS EXPRESSOS EM FAGOS (CORONASCAN)
- Home
- /
- Blog
- /
- Livros
- /
- Capítulo de Livro IV Mostra
- /
- OTIMIZAÇÃO, PADRONIZAÇÃO E CARACTERIZAÇÃO DA BIBLIOTECA DE ANTÍGENOS DE CORONAVÍRUS EXPRESSOS EM FAGOS (CORONASCAN)
Capítulo de livro publicado no Livro da IV Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
doi.org/10.53934/9786599965821-02
Este trabalho foi escrito por:
Gustavo Carvalho Amorim1; Carlos Roberto Prudêncio2
1Estudante do Curso de Especialização em Vigilância Laboratorial em Saúde Pública – Centro de Imunologia – Instituto Adolfo Lutz; E-mail: [email protected] 2Docente/pesquisador do Centro de Imunologia – Instituto Adolfo Lutz.
Resumo: A pandemia da COVID-19 trouxe inúmeros desafios para a sua mitigação ao redor do globo. A tecnologia de imunoprecipitação de fagos seguida pelo sequenciamento de alto rendimento (PhIP-Seq) propõe analisar o histórico imunológico individual e estudar interações antígeno-anticorpo por meio de fagos que expressam proteínas e peptídeos de interesse. Esse trabalho tem por objetivo otimizar e padronizar a técnica de titulação e expansão de uma biblioteca de antígenos de coronavírus expressos em fagos T7 (CoronaScan), bem como a quantificação de imunoglobulinas IgG (IgG) de amostras em soro humano por ELISA, que, combinadas, permitirão o aprimoramento de análises e estudos imunológicos e epidemiológicos. A cepa E. coli BLT5403 foi usada como hospedeira nos ensaios de titulação e expansão da biblioteca CoronaScan em placas ágar LB. Para a quantificação de IgG, uma curva padrão foi construída a partir de uma solução de IgG purificada e com concentração conhecida, juntamente a determinação de IgG de uma amostra de soro, que deverá ser utilizado como controle nos futuros ensaios. Após a expansão, biblioteca CoronaScan (1,5×105 ± 0,7 pfu/mL) passou a ter a titulação de 1,5×1010 ± 2,12 pfu/mL, enquanto a concentração de IgG adequado para o método se encontrou na diluição de 1/1.000.000. Apesar da otimização e a da padronização de tais técnicas terem sido realizadas, novos ensaios de quantificação de IgG pelo método de ELISA serão necessários para assegurar a reprodutibilidade. Além disso, recomenda-se a realização do controle da qualidade da biblioteca CoronaScan a fim de analisar a sua diversidade de peptídeos.
Palavras–chave: coronavírus; PhIP-Seq; T7; vigilância epidemiológica
INTRODUÇÃO
A pandemia da COVID-19 (Coronavirus disease 2019), doença causada pelo vírus SARS-CoV-2 (do inglês severe acute respiratory syndrome coronavirus 2), requereu um grande esforço mundial para o controle de sua disseminação. Em dezembro de 2022, dados da Organização Mundial da Saúde (OMS) confirmaram um pouco mais de 630 milhões de casos e 6,6 milhões de mortes ocasionados pela doença, que foi identificada pela primeira vez na cidade de Wuhan, na China (Herstein et al., 2021; Peng et al., 2021; Rahman et al., 2021).
Mutações aleatórias nas proteínas estruturais spike e do nucleocapsídeo podem não somente dificultar o diagnóstico como também abalar as estratégias da vigilância epidemiológica já instauradas. A tecnologia de imunoprecipitação de fagos seguida pelo sequenciamento de alto rendimento (NGS) (PhIP-Seq, do inglês Phage
Immunoprecipitation Sequencing) é uma ferramenta poderosa capaz de investigar respostas do sistema imune provocadas por infecções virais, doenças imunes e infecciosas que podem comprometer o sistema imunológico. A técnica, ao ser combinada com a biblioteca de fagos T7 coronavírus VirScan (CoronaScan) – que representa os peptídeos dos coronavírus humanos endêmicos (HCoV-NL63, HCoV-229E, HCoV-OC43 e HCoVHKU1), SARS- CoV-1, SARS-CoV-2 e MERS (Middle East Respiratory Syndrome), além dos peptídeos de coronavírus encontrados em morcegos (BatCoV-Rp3, BatCov-HKU3 e BatCov-279) -, permite a identificação de peptídeos reconhecidos por imunoglobulinas IgG (IgG) por meio da imunoprecipitação mediada por esferas magnéticas sensibilizadas com proteínas A e G, com posterior amplificação das sequências codificantes por reação em cadeia da polimerase (PCR) e sequenciamento de nova geração (NGS) (Mohan et al., 2018; Morgenlander et al., 2021; Wiegand et al., 2022; Xu et al., 2015).
A biblioteca de fagos tem como base o phage display desenvolvido a partir de fagos T7, que apresenta vantagens frente ao fago M13, como maior estabilidade na presença de inserto, alta taxa de replicação em Escherichia coli, resistência a condições extremas e entre outras. (Deng et al., 2018; Krumpe & Mori, 2014; Tan et al., 2016; Yue et al., 2022).
Portanto, este trabalho tem como objetivo a otimização e padronização a titulação e expansão da biblioteca de fagos CoronaScan, e a padronização do ensaio de ELISA para a quantificação de IgG presentes no soro dos pacientes que serão utilizados nos experimentos de imunoprecipitação com a biblioteca.
MATERIAL E MÉTODOS
Os ensaios foram realizados de acordo com os protocolos descritos no Manual T7Select® System da Novagen (2011) e por Mohan e colaboradores (2018). Os resultados foram expressos como média e desvio-padrão (±) de pelo menos dois ensaios independentes. A titulação requereu E. coli BLT5403 com OD600 = 1,0 u.a. cultivada em meio
M9LB (NOVAGEN, 2011). As bactérias foram crescidas neste meio suplementado com x (concentração) ampicilina a 37° C sob agitação constante de 200 RPM. As células hospedeiras foram incubadas por 8 min com 100 µL da biblioteca CoronaScan previamente diluída em meio LB nas diluições de 102 a 109. Em seguida, 3 mL de meio top agarose (20 g de meio LB e 0,6 de agarose low melting em volume final de 1 L) foi adicionado para cada diluição, sendo essa mistura plaqueada sob uma camada de LB ágar com ampicilina a 50 µg/mL em uma superfície plana e incubada overnight a temperatura ambiente (TA). A titulação (pfu/mL) foi determinada por meio da contagem das unidades formadoras de placas (pfu) na placa LB contendo a maior diluição em que as placas de fagos puderam ser contadas de modo isolado. A estimativa da concentração da biblioteca foi auxiliada por meio da seguinte fórmula: Titulação (pfu/mL) = PFU × Fator de diluição × 10.
Para a realização da expansão da biblioteca CoronaScan, foi necessitado uma concentração de 1×106 de fagos para cada 10 mL das células hospedeiras com OD600 = 0,6 a 1,0 u.a. Foi separado, em diferentes tubos, 1 mL da mistura fagos/hospedeiro e adicionado 10 mL de meio top agarose aquecido a 45° C, sendo plaqueada em placas de Petri contendo
uma camada de ágar LB com ampicilina a 50 µg/mL, seguido pela incubação em TA em uma superfície plana overnight. Após a conferência da quase confluência da placa, 10 mL do tampão de extração (20 mM Tris-HCl pH 8.0, 100 mM NaCl e 6 mM MgSO4) foi adicionado e as placas foram incubadas a 4° C em uma superfície plana overnight. Assim, o tampão de extração foi transferido para um único tubo estéril e 500 µL de clorofórmio foi adicionado para uma centrifugação de 3000 g por 5 minutos, sendo a nova titulação conferida após o término. A biblioteca foi aliquotada e armazenada a -80° C em DMSO 10% (vol/vol).
A quantificação de IgG foi realizada por um ELISA sanduíche, onde uma placa de 96 poços foi adsorvida com 2 µg/mL da porção Fab de anti-IgG humana (SouthernBiotech®, Birmingham, Alabama, EUA), produzido em cabra, diluído em tampão carbonato- bicarbonato (pH 9.5) em volume final de 50 µL/poço. A placa foi selada e incubada overnight a 4ºC e, então, o anticorpo de captura não adsorvido foi descartado. Logo, a placa foi lavada com tampão de lavagem (PBS 0,05% Tween-20) e bloqueada com solução de bloqueio (PBS 5% leite desnatado Molico®, Vevey, Suíça) por 1 hora a 37° C. A placa foi lavada novamente e 50 µL/poço da solução padrão de IgG (Human IgG Normal, Invitrogen®, Waltham, Massachusetts, EUA) diluída em solução de bloqueio foi adicionada em duplicata nas concentrações de 200, 100, 33.33, 11.11, 3.7, 1.23 e 0.41 ng/mL. Concomitantemente, um soro pertencente ao banco de soros do Laboratório de Imunobiotecnologia do Instituto Adolfo Lutz (autorizada, sob o n° 32264120.5.2001.0061, no Comitê de Ética do Instituto de Infectologia Emílio Ribas) foi diluído em solução bloqueio nas razões de 1/10.000, 1/100.000, 1/400.000, 1/800.000, 1/1.000.000, 1/2.000.000, 1/4.000.000 e 1/8.000.000 e
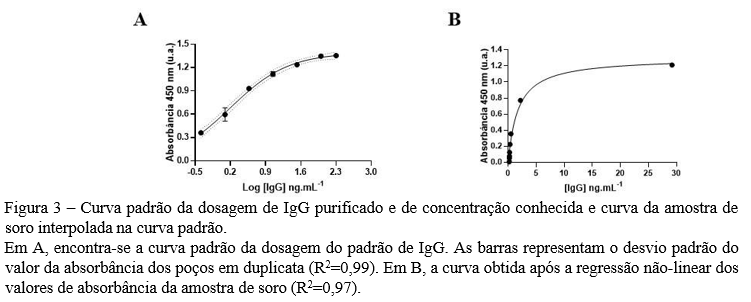
adicionada à placa em duplicata em volume de 50 µL/poço, sendo a placa selada e incubada por 1 hora a 37° C. Em seguida, a placa foi lavada 5x para remoção do material não ligado. Posteriormente, foi feita a incubação com 50 µL/poço de uma solução de anticorpo anti- human IgG -HRP (1:5000) em TA por 30 min. Após a lavagem, 50 µL/poço do substrato TMB (3,3′,5,5′-tetrametilbenzidina) foi adicionado em TA e a placa foi coberta em papel alumínio, sendo a intensidade de coloração monitorada a cada 2 minutos. A solução de (0,2 N de H2SO4) foi adicionada seguindo a ordem de adição de TMB (50 µL/poço). Por fim, a absorbância da placa foi lida no comprimento de onda (λ) de 450 nm. Os dados obtidos foram analisados no software GraphPrism versão 8.0.1 (GraphPad Software, San Diego, Califórnia EUA). A concentração da amostra de soro foi determinada pela interpolação dos valores de absorbância obtidos na diluição 1/1.000.000 com a curva padrão pelo modelo “sigmoidal, 4 PL and X is logX (concentration)”, sendo a curva padrão da amostra de soro construída por meio da análise de regressão não-linear pelo modelo “one-site binding model”.
RESULTADOS E DISCUSSÃO
Inicialmente, a biblioteca CoronaScan, que já se encontrava em posse do Laboratório de Imunobiotecnologia do IAL, obteve uma média de titulação (realizada em duplicata) de 1,5×105 (± 0,7) pfu/mL, como demonstrado na tabela 1.

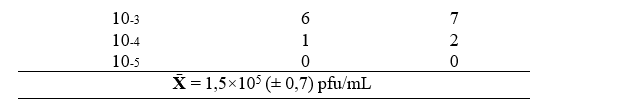
O processo morfogênico do fago T7 garante a expressão de uma maior diversidade de aminoácidos quando comparado ao fago M13, acarretando um maior número de peptídeos funcionais, a depender do quanto a nova molécula pode interferir na instabilidade da partícula viral bem como impedir a replicação do mesmo (Krumpe et al., 2007).
O método de escolha para a amplificação da biblioteca foi por ensaio de lise em placa, uma vez que o total de recombinantes primários era inferior a 5×106 pfu/mL, como demanda o protocolo da Novagen T7Select® System Manual (NOVAGEN, 2011). Após a expansão, foi possível observar pela grande confluência das placas utilizadas no procedimento que a titulação havia aumentado. Após a realização de uma nova titulação, determinou-se que a biblioteca foi aumentada cerca de 100.000 vezes, estando estimada em um título de 1,5×1010 (± 2,12) pfu/mL, conforme observado na tabela 2.

Embora o título da biblioteca tenha aumentado consideravelmente, é preocupante a qualidade da biblioteca, no que se diz a respeito à diversidade viral em que ela representa. O sequenciamento de Sanger e o NGS são duas alternativas que podem auxiliar na análise representativa dos fagos ali presentes. O sequenciamento de Sanger possibilita a investigação acerca da capacidade de síntese de oligonucleotídeos dos fagos, sendo limitado a 103 sequências, enquanto o NGS permite identificar a distribuição da frequência clonal da biblioteca e de sua integridade em uma janela de 106 a 108 ligantes, o que minimiza a subestimação da população de fagos e, consequentemente, na qualidade geral da biblioteca. Além disso, mutações e proteínas truncadas são características conhecidas por alterar a cinética de crescimento dos fagos que as apresentam, levando à proliferação vantajosa frente aos demais (Matochko & Derda, 2015; Mohan et al., 2018).
O PhIP-Seq exige, ao menos, 1 µL de soro por paciente, uma vez que apenas 2 µg de IgG para a técnica de imunoprecipitação é requisitado. Jollift e colaboradores (1982) observaram por nefelometria que os valores de referência de IgG em adultos permeiam entre 6,39 a 13,49 g/L, em uma média de 9,94 g/L. A curva padrão foi construída com os valores da concentração de IgG padrão conhecida e transformados em valores de logaritmo na base
10. A interpolação das amostras à curva padrão se mostrou ter um ajuste adequado para a curva (R2=0,99) de acordo com o software GraphPad. Assim sendo, a diluição do soro na razão de 1/1.000.000 recomendada por Mohan et al. (2018) foi adequada para a obtenção de 9 mg/mL de IgG a partir da amostra de soro, ambas as curvas podem ser observadas na figura 3.
Ao utilizarem a biblioteca VirScan, uma biblioteca similar que engloba todo o viroma humano, Xu et al. (2015) constataram uma sensibilidade e especificidade de 95% quando testada e comparada para o vírus da imunodeficiência humana (HIV) e vírus da hepatite C (HCV) e seus respectivos ensaios de ELISA, assim como em teste sorológico para Herpes Simplex Virus tipo 1 (HSV-1) e tipo 2 (HSV-2), possibilitando a avaliação desses parâmetros para os vírus dos mais diversos tamanhos. Shrock e colaboradores e (2020) com a mesma técnica, descreveram uma sensibilidade de 99% e especificidade de
98% para um modelo computacional capaz de identificar a exposição contra o SARS-CoV2. Chen e colaboradores (2022) descreveram um modelo computacional, BEER, capaz de aumentar a sensibilidade de peptídeos fracamente reativos para o uso do PhIP-Seq.
Os mecanismos que desencadeiam sequelas pós-agudas do SARS-CoV-2 (PASC, do inglês Post-Acute Sequelae of SARS-CoV-2) ainda não estão muito bem esclarecidos. Uma saída para elucidar os biomarcadores que conduzem a essa condição é o uso da tecnologia PhiP-Seq, que pode ser relevante para a obtenção de respostas e questionamentos científicos, principalmente dentro do âmbito de estudos epidemiológicos de soroprevalênca (Mantovani et al., 2022).
CONCLUSÕES
Com a otimização da titulação e da expansão da biblioteca CoronaScan, a manutenção, isso é, a constante renovação da biblioteca, poderá ser feita ao longo do tempo a medida em que os ensaios de PhIP-Seq forem sendo realizados, se tornando uma fonte inesgotável de reagente. No entanto, a quantificação de IgG pelo método de ELISA ainda precisa ser aprimorada para assegurar a reprodutibilidade em amostras de soro de pacientes de diferentes quadros clínicos. Sobretudo, recomenda-se a realização do controle de qualidade de biblioteca, devido ao fato da mesma ter sido expandida a partir de um estoque cuja titulação era abaixo do valor esperado.
AGRADECIMENTOS
Aos meus colegas de laboratório de Imunobiotecnologia, que contribuíram imensamente para a construção desse trabalho e para a minha formação e a coordenadoria e professores do Curso de Especialização em Vigilância Laboratorial em Saúde Pública do Instituto Adolfo Lutz, com os quais tornaram esse projeto possível.
REFERÊNCIAS
CHEN, Athena et al. Detecting and quantifying antibody reactivity in PhIP-Seq data with BEER. Bioinformatics, v. 38, n. 19, p. 4647-4649, 2022.
DENG, Xiangying et al. Advances in the T7 phage display system. Molecular medicine reports, v. 17, n. 1, p. 714-720, 2018.
HERSTEIN, Jocelyn J. et al. Emergency preparedness: What is the future?. Antimicrobial Stewardship & Healthcare Epidemiology, v. 1, n. 1, p. e29, 2021.
JOLLIFT, C. R. et al. Reference intervals for serum IgG, IgA, IgM, C3, and C4 as determined by rate nephelometry. Clinical chemistry, v. 28, n. 1, p. 126-128, 1982. KRUMPE, L. R. H.; MORI, T. T7 lytic phage-displayed peptide libraries: Construction and diversity characterization. Methods in Molecular Biology, v. 1088, p. 51–66, 2014.
KRUMPE, Lauren RH et al. Trinucleotide cassettes increase diversity of T7 phagedisplayed peptide library. BMC biotechnology, v. 7, n. 1, p. 1-8, 2007.
MANTOVANI, Alberto et al. Long Covid: where we stand and challenges ahead. Cell Death & Differentiation, v. 29, n. 10, p. 1891-1900, 2022.
MATOCHKO, Wadim L.; DERDA, Ratmir. Next-generation sequencing of phagedisplayed peptide libraries. Peptide Libraries, p. 249-266, 2015.
MOHAN, Divya et al. PhIP-Seq characterization of serum antibodies using oligonucleotide- encoded peptidomes. Nature protocols, v. 13, n. 9, p. 1958-1978, 2018. MORGENLANDER, William R. et al. Antibody responses to endemic coronaviruses modulate COVID-19 convalescent plasma functionality. The Journal of clinical investigation, v. 131, n. 7, 2021.
NOVAGEN. T7Select® System Manual. Disponível em:
www.merck4biosciences.com.
PENG, Xue-Liang et al. Advances in the design and development of SARS-CoV-2 vaccines.
Military Medical Research, v. 8, n. 1, p. 1-31, 2021.
RAHMAN, Sayeeda et al. Epidemiology, pathogenesis, clinical presentations, diagnosis and treatment of COVID-19: a review of current evidence. Expert review of clinical pharmacology, v. 14, n. 5, p. 601-621, 2021.
SHROCK, Ellen et al. Viral epitope profiling of COVID-19 patients reveals crossreactivity and correlates of severity. Science, v. 370, n. 6520, p. eabd4250, 2020. TAN, Yuyu et al. Advance in phage display technology for bioanalysis. Biotechnology journal, v. 11, n. 6, p. 732-745, 2016.
WHO COVID-19 Dashboard. Geneva: World Health Organization, 2020. Disponível em:
https://covid19.who.int/. Acessado em: 01/12/2022.
WIEGAND, Tanner et al. The Rise and Fall of SARS-CoV-2 Variants and Ongoing Diversification of Omicron. Viruses, v. 14, n. 9, p. 2009, 2022.
XU, George J. et al. Comprehensive serological profiling of human populations using a synthetic human virome. Science, v. 348, n. 6239, p. aaa0698, 2015.
YUE, Hui et al. T7 Phage as an Emerging Nanobiomaterial with Genetically Tunable Target Specificity. Advanced Science, v. 9, n. 4, p. 2103645, 2022.


