OTIMIZAÇÃO DO PROCESSO DE EXPRESSÃO DA PROTEÍNA RECOMBINANTE FUSIONADA (RBD + N) DO SARS-COV-2 EM E. COLI
- Home
- /
- Blog
- /
- Livros
- /
- Capítulo de Livro IV Mostra
- /
- OTIMIZAÇÃO DO PROCESSO DE EXPRESSÃO DA PROTEÍNA RECOMBINANTE FUSIONADA (RBD + N) DO SARS-COV-2 EM E. COLI
Capítulo de livro publicado no Livro da IV Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
doi.org/10.53934/9786599965821-24
Este trabalho foi escrito por:
Isabela Fernandes Rodrigues1; Carlos Roberto Prudêncio2.
1Estudante do Curso de Especialização em Vigilância Laboratorial em Saúde Pública – Centro de Imunologia – Instituto Adolfo Lutz; E-mail: [email protected]
2Docente/pesquisador do Centro de Imunologia – Instituto Adolfo Lutz
Resumo: A pandemia ocasionada pelo SARS-CoV-2 levou a comunidade científica a uma rápida busca por antígenos virais capazes de induzirem uma resposta imunológica eficiente para a produção de vacinas, bem como na aplicação em testes diagnósticos sorológicos. A obtenção e utilização de proteínas recombinantes por meio de técnicas biotecnológicas, tem se mostrado muito eficiente em razão do seu custo-benefício e da sua diversidade de aplicação, contribuindo para o avanço científico. Este trabalho teve como objetivo otimizar o processo de expressão de uma proteína recombinante fusionada (RBD+N) do SARS-CoV- 2 em bactérias E. coli, avaliando as melhores condições para a obtenção da proteína pura, que futuramente servirá para testes imunológicos in vitro e in vivo. O vetor de expressão pH6HTC His6 HaloTag® T7 Vector (Promega Corporation, USA) foi utilizado para a expressão da sequência da proteína fusionada. As cepas de E. coli BL21 (DE3) pLysS e Rosetta foram transformadas com o plasmídeo, para a realização da expressão. A extração da proteína foi realizada utilizando os tampões Bug Buster® e Sarkosyl e submetidas à purificação por cromatografia de afinidade. As amostras foram analisadas por gel de SDS- PAGE e Western Blotting. A cepa E. coli Rosetta no tempo de 4 horas apresentou maior rendimento de proteína expressa, enquanto a E. coli BL21 no tempo de 4 horas foi a que menos expressou. Ambas as cepas no tempo de 18 horas expressaram concentrações próximas. Ao avaliar o perfil de purificação, a BL21 apresentou baixa formação de corpo de inclusão possibilitando a liberação da proteína no primeiro estágio de extração proteica. Os resultados do Western blotting sugerem maiores investigações de possíveis problemas da sequência gênica no vetor de expressão que possam ter influenciado na purificação devido ao não reconhecimento da proteína pelo anticorpo anti-His tag.
Palavras–chave: E. coli, proteína recombinante fusionada, SARS-CoV-2.
INTRODUÇÃO
Em dezembro de 2019, o aparecimento de diversos casos de pneumonia na cidade de Wuhan, na China, acendeu um alerta para que o governo chinês buscasse a fonte do surto e comunicasse aos órgãos internacionais de vigilância. Posteriormente, em fevereiro, taxonomistas classificaram o vírus em SARS-CoV-2 (do inglês, severe acute respiratory syndrome coronavirus 2) e a OMS nomeou a nova doença como COVID-19 (Coronavirus disease 2019). Em meados de março já havia a circulação do vírus por diversos países ao redor do mundo, sendo caracterizada como pandemia, impactando fortemente na saúde pública global (SUN et al., 2020).
O vírus possui tropismo pelo trato respiratório, principalmente nas células pulmonares, causando uma série de complicações ao hospedeiro (HARRISON, LIN, WANG, 2020). Os casos variam de leves, moderados e graves. Entre os sintomas comumente relatados estão a tosse, febre e dor de garganta. Em casos mais graves, os pacientes podem apresentar dor no peito, falta de ar, podendo evoluir para uma síndrome respiratória aguda grave, necessitando de internação hospitalar (WHO, 2022).
Com o rápido aumento dos números de casos mundialmente, houve a necessidade da busca por marcadores virais capazes de auxiliarem na terapêutica e no diagnóstico da nova patologia (YÜCE, FILIZTEKIN, ÖZKAYA, 2021). A procura por sítios ativos e imunogênicos para produção de vacinas foi o foco de diversos grupos de pesquisa na tentativa de desenvolver antígenos recombinantes capazes de induzir a resposta imunológica humoral e/ou celular contra o novo vírus (AWADASSEID et al., 2021).
Este trabalho teve como objetivo otimizar o processo de expressão de uma proteína recombinante fusionada (RBD+N) do SARS-CoV-2 em bactérias Escherichia coli, avaliando as melhores condições para a obtenção da proteína pura, que futuramente servirá para testes imunológicos in vitro e in vivo.
MATERIAL E MÉTODOS
O vetor de expressão pH6HTC His6 HaloTag® T7 Vector (Promega Corporation, USA) foi utilizado para a expressão da sequência da proteína recombinante resultante da fusão das proteínas RBD (domínio de ligação ao receptor, uma subunidade de proteína Spike, de superfície do vírus) e N (proteína do nucleocapsídeo). As cepas E. coli BL21 (DE3) pLysS e Rosetta foram transformadas com o plasmídeo, para a realização da expressão. Foi realizado inicialmente um teste de indução após atingirem OD600nm 0,6 em 5 mL de cultura com concentração 0,4 mM de IPTG, para avaliar as características de cada cepa e posteriormente a expressão em 100 mL de cultura a 0,5 mM de IPTG em dois tempos diferentes (4 e 18 horas). A extração da proteína foi realizada utilizando os tampões Bug Buster® (Novagen®, USA), juntamente com a adição de lisozima (Invitrogen, USA; 1000 U/mL) e Benzonase® Nuclease (Sigma-Aldrich®, USA; 25 U/mL) e Sarkosyl (1,5% Sarkosyl, 25 mM Trietanolamina, 1mM de EDTA) e submetidas à purificação por cromatografia de afinidade. As amostras foram analisadas por SDS-PAGE e Western blotting.
RESULTADOS E DISCUSSÃO
A utilização de bactérias para a produção de proteínas heterólogas tem sido amplamente aplicada no ramo da biotecnologia. Muitas proteínas de interesse comercial têm-se utilizado esse modelo de vetor para estudos estruturais e funcionais devido ao seu baixo custo, fácil manipulação, rápido crescimento e alto rendimento (ROSANO, CECCARELLI, 2014). A espécie E. coli é a principal escolha de muitos pesquisadores devido a sua simplicidade genômica passível de transformação com DNA exógeno, ciclo de divisão celular entre 20 a 30 minutos em condições de crescimento favoráveis, além de possuir sua fisiologia bem caracterizada. (FRANCIS, PAGE, 2010).
Com os avanços da engenharia genética, a E. coli passou por diversas modificações a fim de melhorar seu desempenho na produção de insumos biotecnológicos, originando diversas cepas. Neste trabalho, a expressão heteróloga da proteína fusionada (RBD+N) foi avaliada nas cepas BL21(DE3) que possui uma cópia da RNA polimerase do fago T7 e possibilita a transcrição de sequências gênicas controladas pelo promotor T7 lac UV5(ROSANO, MORALES, CECCARELLI, 2019; RESENDE, 2015), e na cepa Rosetta que é recomendada para proteínas que contenham códons raros.
Dado tal contexto, as bactérias foram transformadas com o plasmídeo contendo a sequência da proteína recombinante fusionada nessas duas cepas. Para um teste inicial de expressão, foi realizada a indução durante um período de 3 horas, com uma concentração de IPTG de 0,4 mM. Durante o teste, foi retirada uma alíquota pré-indução (T0) e pós-indução a cada uma hora até completar 3 horas (T1, T2 e T3). As amostras foram submetidas à SDS- PAGE e o resultado está demonstrado na Figura 1. Após análise, foi possível observar que a cepa Rosetta forneceu um gel mais límpido em relação a BL21, tendo uma menor expressão de proteínas basais.
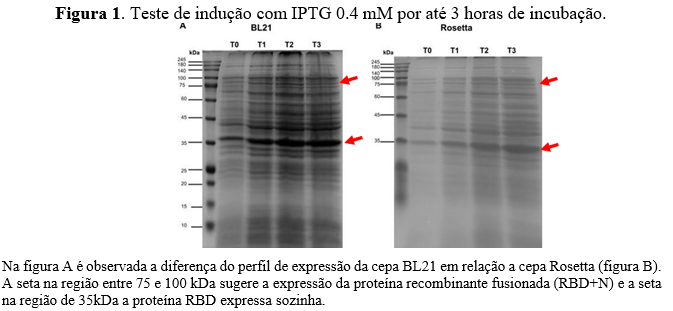
Para dar continuidade aos estudos, a expressão da proteína foi feita em uma escala maior, e decidiu-se alterar a concentração do indutor para 0,5 mM, que se baseou em estudos recém-publicados por Castro e colaboradores (2022), que trabalharam também com uma proteína fusionada (RBD+N), utilizando a concentração de 0,5 mM do indutor durante 18 horas. Portanto, as cepas foram induzidas por 4 e 18 horas de incubação a 37º C. Após a extração da proteína, utilizando dois tipos de detergentes para lisar a célula bacteriana, Bug Buster® e Sarkosyl, notou-se que os sobrenadantes obtidos dos cultivos de 18 horas apresentaram-se um aspecto viscoso, portando, essas amostras foram tratadas com DNAse (5 µg/mL), que após o tratamento ficaram fluídas, e essa característica auxilia na purificação por cromatografia de afinidade.
O vetor de expressão pH6HTC His6 HaloTag® T7 possui uma sequência de 6 resíduos de histidinas que são adicionadas na porção C-terminal da proteína alvo. Essa fusão de aminoácidos é responsável por auxiliar na purificação, através de ligações reversíveis ao níquel presente na coluna cromatográfica, permitindo que apenas as proteínas com essa cauda se liguem com maior afinidade, separando-as das demais produzidas (RESENDE, 2015). A reversão dessas ligações é realizada através da adição de imidazol no tampão de eluição, que por competição, se liga ao níquel liberando a proteína que estava imobilizada. A partir desta técnica, espera-se uma obtenção mais pura e sem contaminantes em uma única etapa (BORNHORST, FALKE, 2000). Para a purificação foi utilizado o sobrenadante após a lise celular com o tampão Sarkosyl, diluído no tampão de equilíbrio A (50 mM de Fosfato de sódio, 500 mM de Cloreto de sódio). A eluição da amostra ocorreu utilizando um gradiente de concentração de 5% a 100% do tampão B (50 mM de Fosfato de sódio, 500mM de Cloreto de sódio, 500 mM de Imidazol).
Ao analisar o perfil de purificação, as amostras da cepa Rosetta 4 e 18 horas e a BL21 18 horas apresentaram perfil de eluição próximas, entre 54-58% de imidazol (~ 280 mM). Entretanto, a amostra de BL21 de 4 horas apresentou eluição em 100% da concentração de imidazol (500 mM) presente no tampão (Figura 2). Logo, uma nova expressão dessa amostra foi realizada para observar se é perfil da cepa ou se havia algum erro durante o processo. Após a segunda purificação, observou-se que o perfil de eluição se manteve o mesmo (Figura 3) e dado tal cenário, surgiu a hipótese de que as proteínas estariam no sobrenadante do Bug Buster®, e que no tempo de 4 horas de indução nessa cepa, a formação de corpos de inclusão é baixa, sendo as proteínas liberadas no primeiro passo de extração.
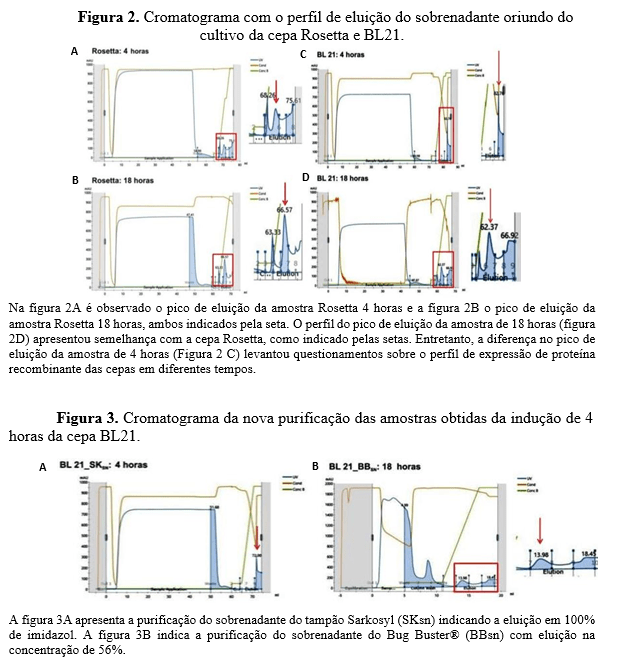
Para uma análise quantitativa das proteínas obtidas nas amostras, foi realizada a concentração das amostras para quantificação utilizando um kit comercial ((Pierce™ Rapid Gold BCA Protein Assay Kit – Thermo Fisher Scientific, USA). Os resultados da quantificação estão apresentados na Tabela 1. O eluido da purificação obtidos do cultivo da cepa Rosetta no tempo de 4 horas apresentou maior quantidade de proteína expressa dentre as amostras, enquanto os eluentes da BL21 4 horas produziu proteínas em menor concentração. As amostras de 18 horas apresentaram concentrações próximas.
Tabela 1. Quantificação da proteína expressa.

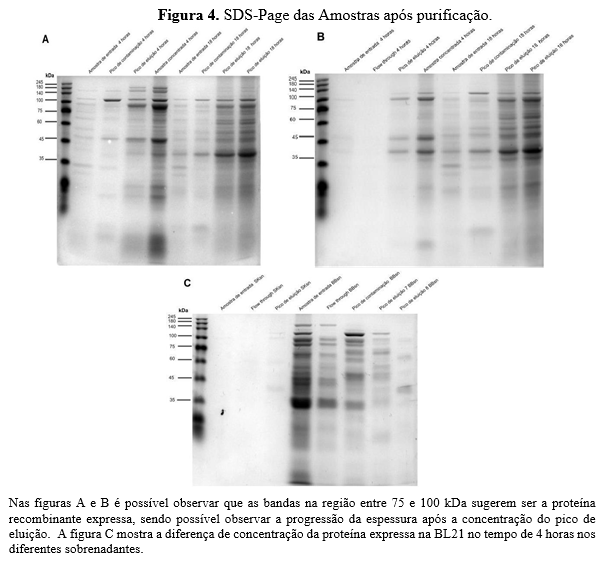
Ao final de todo o processo, as amostras foram submetidas à análise de SDS-PAGE e Western blotting. Os resultados da primeira análise revelaram que a amostras provenientes da cepa Rosetta do tempo de 4 horas apresentou uma banda de maior densidade em tempo de 18 horas (Fig. 4A), corroborando com os resultados da quantificação proteica. Observou- se também bandas entre 75 e 100 kDa, que sugere ser a proteína fusionada; porém notou-se uma banda da região de 35 kDa, característico da proteína RBD indicando que a purificação não foi eficiente, pois teoricamente a cauda de histidina estaria presente na proteína quimérica. Em relação à cepa BL21 (Fig. 4B) ocorreu a situação inversa, no tempo de 18 horas a bactéria produziu mais proteína em relação ao tempo de 4 horas. Também foi realizado um gel para analisar a segunda purificação do tempo de 4 horas da BL21 para comparar a eluição da proteína nos diferentes sobrenadantes (Fig. 4C). A partir desses resultados, concluiu-se que a maior concentração de eluição da proteína estava no sobrenadante do Bug Buster® e bem pouca no Sarkosyl. Os resultados do SDS-PAGE também indicaram a ineficiência da purificação, visto que apenas proteínas que possuem cauda de histidina deveriam ser eluída, apresentando-se em uma banda única no gel.
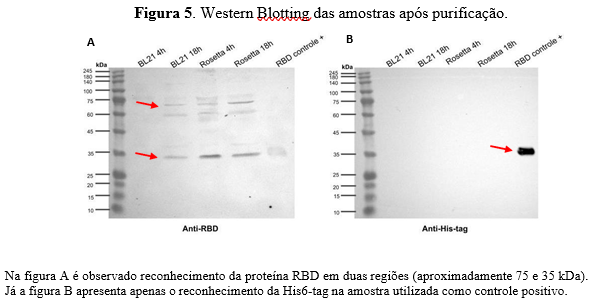
Em ambos os géis, ficou evidente uma banda espessa próxima a marcação de 35 kDa, que se assemelha ao perfil da proteína RBD sozinha e ainda há necessidade de otimizar o método de purificação. Para confirmar a expressão da proteína fusionada RBD+N, foi realizado um Western Blotting com imunorreação com anticorpo primário de coelho anti- His6-tag (Sigma-Aldrich®, USA; diluição 1:1000) e soro de coelho anti-RBD (produzido in-house pelo grupo de pesquisa). A membrana tratada com soro anti-RBD, apresentou marcação em duas regiões (~60 kDa e 35 kDa) (Figura 5A), indicando que além da proteína inteira, a bactéria possivelmente também expressou somente a porção da RBD. A membrana tratada com anticorpo anti-His6-tag para analisar o peso molecular e confirmar a expressão da proteína fusionada não apresentou imunorreação para o reconhecimento da cauda de histidina nas amostras pelo anticorpo, reconhecendo apenas o RBD com cauda de histidina (produzido pelo grupo de pesquisa) que foi utilizado como controle positivo (Figura 5B). Desta forma, supõe-se que a cauda da Histidina não está sendo produzida completa, pois está na sequência do vetor e não na sequência gênica da proteína, podendo haver algum problema na fusão entre as sequências. Fato que fica evidente durante a purificação, pois observou-se que um pico de eluição na concentração de aproximadamente 300 mM, sugerindo que apenas proteínas com poli-histidinas (mais de 3 resíduos de aminoácidos) ficariam mobilizadas nas colunas e eluiriam nessa concentração.
CONCLUSÕES
A utilização de duas cepas de E. coli, BL21(DE3) pLysS e Rosetta, permitiu avaliar o perfil em diferentes condições de tempo, mantendo a concentração do indutor e temperatura. A cepa Rosetta no tempo de 4 horas produziu maior quantidade de proteína recombinante fusionada (RBD+N), enquanto a cepa BL21 no mesmo tempo produziu menor quantidade.
Ao avaliar o perfil de purificação, a BL21 de 4 horas sugere a baixa formação de corpos de inclusão possibilitando a solubilização da proteína no citosol bacteriano e sendo liberado na primeira fase de lise celular.
Os resultados do Western Blotting sugerem a necessidade de maiores investigações quanto a possíveis interferentes na sequência gênica no vetor durante a expressão, para continuar otimizando a produção da proteína recombinante fusionada (RBD+N) a fim de obtê-la pura e sem contaminantes.
AGRADECIMENTOS
Gostaria de agradecer a todos os colaboradores do laboratório de Imunotecnologia do Instituto Adolfo Lutz que me auxiliaram durante o processo para a realização deste trabalho.
REFERÊNCIAS
AWADASSEID, Annoor et al. Current Advances in the development of SARS-CoV-2 vaccines. International Journal Of Biological Sciences, v. 17, n. 1, p. 8, 2021.
BORNHORST, Joshua A.; FALKE, Joseph J. [16] Purification of proteins using polyhistidine affinity tags. In: Methods in enzymology. Academic Press, 2000. p. 245-254.
CASTRO, Julia T. et al. Promotion of neutralizing antibody-independent immunity to wild- type and SARS-CoV-2 variants of concern using an RBD-Nucleocapsid fusion protein. Nature communications, v. 13, n. 1, p. 1-16, 2022.
FRANCIS, Dana M; PAGE, Rebecca. Strategies to optimize protein expression in E. coli. Current protocols in protein science, v. 61, n. 1, p. 5.24. 1-5.24. 29, 2010.
HARRISON, Andrew G.; LIN, Tao; WANG, Penghua. Mechanisms Of SARS-CoV-2 transmission and pathogenesis. Trends in immunology, v. 41, n. 12, p. 1100-1115, 2020.
RESENDE, Rodrigo R. Biotecnologia aplicada à saúde fundamentos e aplicações: Vol 2. Editora Blucher, 2015.
ROSANO, Germán L; CECCARELLI, Eduardo A. Recombinant protein expression in Escherichia coli: advances and challenges. Frontiers in microbiology, v. 5, p. 172, 2014.
ROSANO, Germán L; MORALES, Enrique S; CECCARELLI, Eduardo A. New tools for recombinant protein production in Escherichia coli: A 5‐year update. Protein science, v. 28, n. 8, p. 1412-1422, 2019.
SUN, Pengfei et al. Understanding Of COVID‐19 based on current evidence. Journal Of medical virology, v. 92, n. 6, p. 548-551, 2020.
TEGEL, Hanna et al. Increased levels of recombinant human proteins with the Escherichia coli strain Rosetta (DE3). Protein expression and purification, v. 69, n. 2, p. 159-167, 2010.
WANG, Mei-Yue et al. SARS-CoV-2: structure, biology, and structure-based therapeutics development. Frontiers in cellular and infection microbiology, v. 10, p. 587269, 2020.
WHO. World health organization, 2022. Coronavírus Disease (COVID-19) Symptoms. Disponível em: <https://www.who.int/health-topics/coronavirus#tab=tab_3>. Acesso em: 06 out. 2022.
WHO. World health organization, 2022. Coronavírus Disease (COVID-19) Prevention. Disponível em: <https://www.who.int/health-topics/coronavirus#tab=tab_2>. Acesso em: 06 out. 2022.
YANG, Haitao; RAO, Zihe. Structural biology Of SARS-CoV-2 and implications for therapeutic development. Nature Reviews Microbiology, v. 19, n. 11, p. 685-700, 2021.
YÜCE, Meral; FILIZTEKIN, Elif; ÖZKAYA, KorinGasia. COVID-19 diagnosis — A review of current methods. Biosensorsand Bioelectronics, v. 172, p. 112752, 2021.
