Resumo expandido TCC
- Home
- /
- Resumo expandido TCC
- /
- Page 4
Resumo expandido publicado nos Anais da III Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
Este trabalho foi escrito por:
Amilson da Silva Ribeiro
Estudante do Curso de Vigilância Laboratorial em Saúde Pública – NCB – IAL CLR III
E-mail: [email protected]
RESUMO
Introdução: A Escherichia coli (E. coli) é uma bactéria bacilar anaeróbia facultativa pertencente a família Enterobacteriaceae presente na flora intestinal de humanos. Geralmente não são patogênicas, porém existem alguns sorogrupos que possuem diversos fatores de virulência que são capazes de produzir toxinas provocando quadros diarreiogênicos e outras doenças extraintestinais. O diagnóstico laboratorial é realizado através de culturas de amostras biológicas, provas bioquímicas de identificação e testes de biologia molecular. Objetivo: Este trabalho propõe descrever as características das E. coli patogênicas e o seu perfil epidemiológico. Metodologia: Foi realizado uma revisão bibliográfica baseando-se em livros, dissertações de mestrado, teses de doutorado, monografias e artigos científicos. Resultados e discussão: A EHEC/STEC O157:H7 é uma das principais linhagens causadoras de surtos de origem hídrica e alimentar e também é responsável por causar a Síndrome Hemolítico Urêmica (SHU). Outras cepas também podem provocar a SHU como a O104:H4 causadora de um surto de enorme proporção na Alemanha em 2011. No Brasil nos anos de 2009 a 2018, a E. coli foi o patógeno de maior relevância em surtos de doenças de origem alimentar. A EPEC foi o sorotipo mais identificado em surtos no estado de São Paulo entre 2009 e 2018. Conclusão: As E. coli patogênicas são constantemente isoladas em surtos no Brasil e pelo mundo, desse modo, portanto é preciso fortalecer o sistema de vigilância epidemiológica com o objetivo de prevenir novos casos de contaminação e surtos subnotificados.
Palavras-chave: Doenças transmitidas por alimentos, Epidemiologia, Escherichia coli e Surtos.
INTRODUÇÃO
A Escherichia coli (E. coli) é uma bactéria bacilar anaeróbia facultativa pertencente a família Enterobacteriaceae que possue habitat no intestino de humanos e animais. Foi identificada pela primeira vez pelo médico pediatra Theodor Von Escherich em 1885 (GOMES et al, 2016).
A maior parte das cepas de E. coli não são patogênicas, porém as linhagens que possuem potencial patogênico são capazes de produzir toxinas acarretando doenças específicas provocando sintomas graves que necessitam hospitalização (MADIGAN et al, 2016). São bastonetes não esporulados que em sua maioria são móveis, pois possuem a presença de flagelos peritríquios (GARCIA, 2021).
Existem ao total seis tipos de E. coli patogênicas que provocam infecção entérica em humanos, sendo elas: E. coli enterotoxigênica (ETEC), E. coli enteropatogênica (EPEC), E. coli enterohemorrágica ou produtoras de toxina Shiga (EHEC/STEC), E. coli enteroinvasiva (EIEC), E. coli enteroagregativa (EAEC) e E. coli aderente difusa (DAEC) (SOUZA et al, 2016).
Algumas espécies de E. coli patogênicas podem provocar sintomas como diarreia, enquanto outros causam infecções do trato urinário, doenças respiratórias, pneumonia, e outras doenças (CDC, 2021).
Dentre os sorogrupos mais virulentos as ETEC e STEC possuem uma distribuição mundial afetando majoritariamente crianças menores que 5 anos de idade. Cepas de ETEC provocam em média anualmente 380.000 mortes, principalmente em regiões com saneamento básico precário (OLIVEIRA, 2012).
Vale ressaltar que algumas cepas de E. coli possuem duas ou mais características de diferentes patótipos e, dessa forma, são denominadas espécies híbridas tornando-as mais virulentas e agressivas (GOMES et al, 2016). Como exemplo tem-se a E. coli O104:H4 que possui características da E. coli enterohemorrágica (EHEC) e também da E. coli enteroagregativa (EAEC) (CVE, 2011).
As cepas de E coli patogênicas e comensais são similares fenotipicamente, dificultando sua identificação nos laboratórios de análise, assim o uso de métodos moleculares são importantes para o seu diagnóstico clínico (MOLINA et al, 2021).
Neste sentido este trabalho tem o objetivo de apresentar as principais características patológicas das E. coli patogênicas relatando os surtos e a frequência de infecção provocada por esses patótipos através de dados epidemiológicos. Apresentar a importância desse grupo de enterobactérias responsáveis por grande parte dos surtos alimentares e de outras infecções de notificação compulsória.
MATERIAL E MÉTODOS
Este trabalho foi elaborado através de uma revisão bibliográfica baseando-se em livros, manuais, monografias, sites de pesquisa, teses de mestrado e doutorado e artigos científicos. Foi utilizado como organizador de artigos e referências, o software Mendeley. Definiu-se trabalhar com as seguintes bases de dados Biblioteca virtual em saúde (BVS), MEDLINE, SciELO, EBESCO, LILACS e Google acadêmico. Assim foram consultadas informações referentes ao perfil epidemiológico das E. coli patogênicas nas esferas mundial, nacional e estadual, utilizando-se de descritores como Escherichia coli, Surtos, doenças transmitidas por alimentos e epidemiologia.
RESULTADOS E DISCUSSÃO
Situação epidemiológica das E. coli no mundo
Nos Estados Unidos em 1993, um surto de enorme proporção provocado por E. coli O157:H7 envolveu mais de 700 pessoas em 4 estados do país, com 51 casos de SHU e 4 mortes confirmadas.
Em 1996, no Japão, ocorreu o maior surto provocado por E. coli O157:H7 no qual mais de 8.000 pessoas foram infectadas,106 crianças desenvolveram SHU e três foram a óbito (PAULA et al, 2014).
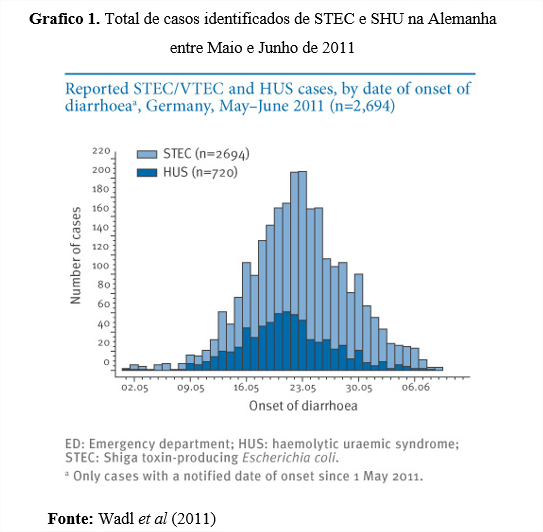
Em 2011, na Alemanha, iniciou-se um surto que se expandiu até outros países da Europa e América do Norte contaminando 4.000 pessoas, sendo que desse total houve 845 casos de SHU com 54 mortes.
De acordo com alguns estudos, o patógeno causador deste surto foi o sorotipo O104:H4 que possui fatores de aderência agregativa e produção de stx2, o que indica um perfil híbrido de EAEC e STEC (SANTOS et al, 2020).
O gráfico 1 demonstra o número de casos identificados no surto que ocorreu na Alemanha entre maio e junho de 2011. Nesse período foram registrados 2694 casos de STEC e 720 casos de SHU associados com quadros de diarreia aguda.
Situação epidemiológica das E. coli no Brasil
Na década de 1980, o patotipo EPEC foi responsável por causar aproximadamente 30% dos casos de diarreia infantil, sendo os sorotipos O111, O55 e O119 os principais causadores (GOMES et al, 1991).
De acordo com dados epidemiológicos, na década de 1990, os patotipos EPEC atípica e EAEC foram os sorotipos mais encontrados nos casos de diarreia aguda, tanto em crianças como em adultos (ORLANDI et al, 2006).
No ano de 2002, ocorreu o primeiro caso registrado de E. coli O157 em doenças em humanos, o patotipo foi isolado de três pacientes que apresentavam um quadro de diarreia sanguinolenta que é um dos principais sintomas da doença (IRINO et al. 2002).
Em 2019, ocorreu um surto em uma creche no estado do Espirito Santo com 22 pessoas atingindas e 1 óbito registrado. Segundo a Vigilância Epidemiológica, o patotipo identificado continha o gene stx2 demonstrando a presença da E. coli STEC/EHEC (SESA, 2019).
Os surtos provocados pelas E. coli patogênicas geralmente estão associados a doenças transmitidas por alimentos (DTA) que corresponde a um incidente com 2 ou mais pessoas que apresentam como principal sintoma a doença diarreica aguda que provoca diversas evaquações com fezes aquosas (CVE, 2011).
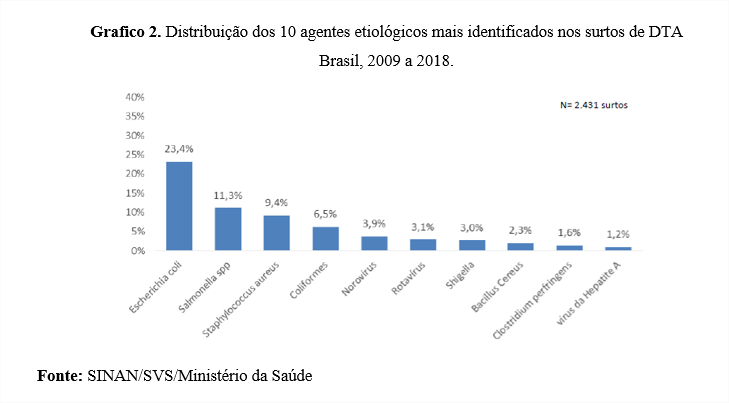
Observando o gráfico 2 fica evidente que a Escherichia coli é o agente etiológico mais identificado em surtos de DTA no período de 2009 a 2018 com cerca de 23,4% dos surtos , seguido de Salmonela spp com 11,3% confirmados.
Situação epidemiológica das E. coli no estado de São Paulo
Na cidade de São Paulo, as EPEC típicas foram os principais agentes etiológicos de diarreia infantil desde a década de 50, o que corresponde a 30% dos casos somente no primeiro ano de vida (OLIVEIRA, 2012).
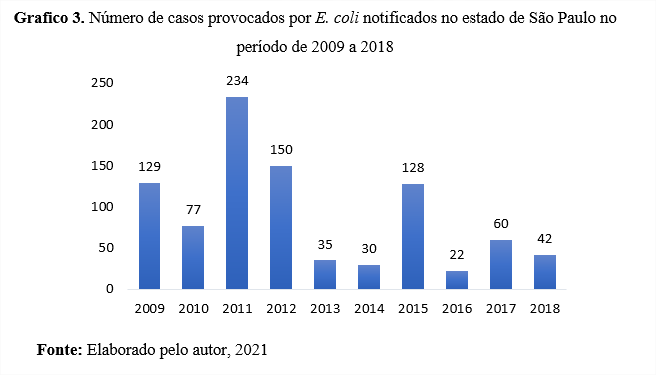
Em 2006, foram identificados 106 casos de EPECsorogrupo O128, sendo o patotipo EPEC o mais identificado nesse período. Nos anos 2006, 2013 e 2014 ocorreram três óbitos provocados por E coli patogênica (AGUIAR, 2019). Na cidade de São Paulo, sorotipos não O157:H7 foram identificados como causadores de SHU (Souza et al, 2007). De acordo com o gráfico 3, no período de 2009 a 2018, o número de casos provocados por E. coli no estado de São Paulo foi maior em 2011 com o total de 234 casos e o menor indice ocorreu em 2016 com 22 casos. Nesse mesmo período, o sorotipo de E. coli mais identificado foi a EPEC como o total de 210 casos.
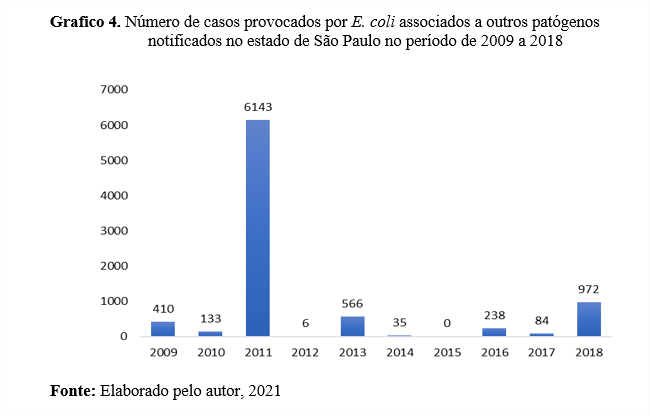
Observando o gráfico 4, o ano de 2011 obteve o maior número de casos ocasionados por E. coli associados a outros patógenos totalizando 6.143 casos, ao contrário do ano de 2015 no qual não houve registros.
CONCLUSÕES
As E. coli patogênicas podem representar um potencial risco para a saúde pública, pois é um microrganismo versátil com facilidade de adquirir vários fatores de virulência criando novos sorotipos com capacidade de causar novas doenças.
Entre 2009 a 2018, as E. coli foram os patógenos mais identificados em surtos transmitidos através de alimentos ressaltando a importância de realizar inquéritos epidemiológicos para identificar o patógeno causador de um surto.
Os números de surtos causados por DTHA são subnotificados no Brasil e a investigação da maioria acabam não chegando no agente causador. É preciso melhorar os sistemas de vigilância e bem como treinar melhor as equipes para as investigações dos surtos.
Dessa forma, espera-se que as E. coli patogênicas continuem aparecendo, sendo em casos esporádicos ou em surtos, pois ainda são frequentemente isoladas como contaminantes em amostras de alimentos e de reservatórios animais.
REFERÊNCIAS
AGUIAR, M. L. Ocorrência de diarreias associadas as Escherichia coli diarreiogênicas. 47f. Monografia (Especialização em Vigilância Laboratorial em Saúde Pública) – Secretaria de Estado da Saúde de São Paulo, Instituto Adolfo Lutz, São Paulo, 2019.
CDC, Centers for Disease Control and Prevention. E. coli (Escherichia coli). Disponível em: <https://www.cdc.gov/ecoli/>. Acesso em: 20 out. 2021.
CVE/SES-SP – Centro de Vigilância Epidemiológica. Secretaria de Estado da Saúde de São Paulo. Divisão de doenças de transmissão hídrica e alimentar. Síndrome Hemolítico-Urêmica e Escherichia coli O104:H4e o surto na Alemanha. São Paulo, 2011.
GARCIA, L. N. H. Identificação e caracterização de Escherichia coli diarreiogenica de abatedouro misto de bovinos e suínos. 69f. Dissertação (Mestrado em Medicina Veterinaria) – Universidade Federal de Viçosa, Viçosa, 2021.
GOMES, T. A. T. et al. Enteropathogens associated with acute diarrheal disease in urban infants in São Paulo, Brazil. Journal of Infectious Disease, v. 164, n. 2, p. 331-337, 1991.
GOMES, T. A. T. et al. Diarrheagenic Escherichia coli. Brazilian Journal of Microbiology, v. 47, n. 4, p. 3–30, 2016.
IRINO, K. et al. O157 Shiga toxin-producing Escherichia coli strains associated with sporadic cases of diarrhea in São Paulo, Brazil. Emerging Infectious Diseases, v. 8, p. 446-447, 2002.
MADIGAN, T. M.; MARTINKO, J. M.; BENDER, K. S.; BUCLEY, D. H.; DAVID, A. S.; Microbiologia de Brock. 14. ed., Porto Alegre: Artmed, 2016.
MOLINA, N. B. et al. Primer reporte de Escherichia coli diarreogénica en población pediátrica ambulatoria con diarrea atendida en la ciudad de La Plata, Argentina. Revista Argentina de Microbiologia, La Plata, abr. 2021.
OLIVEIRA, J. J. Surtos alimentares de origem bacteriana: uma revisão. 36f. Seminário (Disciplina Seminários aplicados do Programa de Mestrado em Ciência Animal). Escola de Veterinária e Zootecnia, Universidade Federal de Goiás, Goiânia, 2012.
ORLANDI, P. P. et al. Etiology of diarrheal infections in children of Porto Velho (Rondônia, Western Amazon region, Brazil). Brazilian Journal of Medical and Biological Research, v. 39, n. 4, p. 507- 517, 2006.
PAULA, C. M. D. et al. Escherichia coli O157:H7 — patógeno alimentar emergente. Vig Sanit Debate. v. 2, n. 4, p. 23-33, 2014.
SANTOS, A. C. M. et al. Diversity of Hybrid and Hetero Pathogenic Escherichia coli and their Potential Implication in More Severe Diseases. Frontiers in Cellular and Infection Microbiology. v. 10, n. 339, p. 131-141, 2020.
SESA, Secretaria do estado da Saúde do Espirito Santo. Investigação do Surto de doença diarreica aguda ocorrido no município de Vila Velha. Vila Velha, 2019.
SOUZA, C. O. et al. Escherichia coli enteropatogênica: uma categoria diarreiogênica versátil. Rev Pan-Amaz Saude, v. 7, n. 2, p. 79-91, 2016.
SOUZA, R. L.; NISHIMURA, L. S.; GUTH, B. E. C. Uncommon Shiga toxin-producing Escherichia coli serotype O165:HNM as cause of hemolytic uremic syndrome in São Paulo, Brazil. Diagnostic Microbiology and Infectious Disease. v. 59, n. 2, p. 223-225, 2007.
Resumo expandido publicado nos Anais da III Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
Este trabalho foi escrito por:
Juliana Lourenço da Silva Pereira1; Daniela Leite2.
1Estudante do Curso de Vigilância Laboratorial em Saúde- Centro de Bacteriologia– IAL. Email: [email protected]
2Docente/pesquisador do Núcleo de Doença Entéricas e Infecções por Patógenos Especiais – NDEI – IAL
RESUMO: A coqueluche é uma doença de transmissão respiratória, altamente contagiosa, causada pela Bordetella pertussis, que pode acometer indivíduos de todas as idades. O grupo de maior risco são os bebês menores de seis meses, não vacinados ou incompletamente imunizados. O diagnóstico laboratorial é realizado pelo isolamento por cultura e/ou pela identificação por qPCR de B. pertussis. Por ser um importante agravo em saúde pública, o objetivo do trabalho foi realizar uma análise epidemiológica dos dados secundários da doença no Estado de São Paulo entre os anos de 2010 a 2020. As informações foram obtidas a partir do banco de dados criado pelo NDEI. Os resultados demonstraram uma maior sensibilidade da qPCR em relação a cultura; um aumento gradual na positividade entre os anos de 2010 e 2014, e maior positividade entre indivíduos do sexo feminino. A faixa de crianças até seis meses foram os mais acometidos pela doença, e as unidades com maior número de exames e positividade foram o IAL Central, CLR de Campinas, Ribeirão Preto e Taubaté respectivamente. Conclui-se que, apesar da coqueluche ser uma doença imunoprevenível, ainda ocorre muitos casos da doença, inclusive epidemias como a de 2010 a 2014. A qPCR é uma técnica de maior sensibilidade em relação a cultura e esta é imprescindível para o diagnóstico, já que é fundamental para realização de estudos epidemiológicos.
Palavras–chave: coqueluche, epidemiologia; cultura; qPCR.
INTRODUÇÃO
Bordetella pertussis é o agente causal da coqueluche, doença infecciosa que apresenta altas taxas de transmissão (90%) e que pode acometer indivíduos de qualquer idade, sendo o grupo de maior risco os bebês menores de seis meses que ainda não completaram o esquema de imunização primária [1; 2; 3]. É um importante problema de saúde pública e apesar de uma redução significativa na carga global da doença com o advento da vacina, a doença permanece endêmica em todo o mundo, com surtos e picos epidêmicos ocorrendo a cada 3-5 anos [4]. Estimativas da Organização Mundial da Saúde (OMS) apontam que, em 2018, ocorreram, em todo o mundo, cerca de 151.000 casos de coqueluche, sendo 90% deles em países em desenvolvimento [5].
A doença é transmitida depessoa a pessoa através de gotículas de saliva de indivíduos contaminados. Possui uma duração média de quatro a 12 semanas, com três estágios: catarral, na qual os pacientes são mais contagiosos e os sintomas são confundidos com infecções respiratórias leves, apresentando febre baixa, coriza, tosse seca e mal-estar; paroxística, na qual a manifestação típica são os paroxismos caracterizados por uma crise súbita e incontrolável de tosse, seguiada de falta de ar e vômitos, ocorre estreitamento da glote que ao respirar profundamente pode emitir um som chamado de “guincho” e convalescença, onde ocorre a diminuição dos paroxismos e vômitos [4; 6;7;8].
O diagnóstico laboratorial para o agravo é realizado pelo isolamento da B. pertussis pela cultura e pela realização do diagnóstico rápido pelo método da PCR em Tempo Real-qPCR, ambos de material colhido de secreção nasofaríngea [9; 10].
Atualmente estão disponíveis dois tipos de vacinas (i) a vacina de células inteiras (wP) produzidas a partir da suspensão de bacilos inteiros mortos e a vacina acelular (aP) composta por um a cinco antígenos purificados [11]. No Brasil, a vacina pentavalente (DTP – toxóides diftérico e tetânico e pertussis + hepatite B e Haemophilus influenzae tipo b) é aplicada aos dois, quatro e seis meses de idade, com primeiro reforço com a DTP aos 15 meses e o segundo, entre os quatro e seis anos de idade. Em 2014, foi incluído no calendário vacinal a imunização de gestantes aplicada a cada gestação entre a 20ª e a 36ª semana (vacina dTpa) [12].
O Centro de Bacteriologia (CB) do Instituto Adolfo Lutz Central (IAL) é o Laboratório de Referência Nacional para Coqueluche, sendo responsável pelo diagnóstico laboratorial pelo método da cultura, e pelo método molecular da qPCR. O IAL também é responsável pelo desenvolvimento de pesquisas para caracterizar os isolados de B. pertussis circulantes no país.
Este trabalho teve como objetivo ampliar o conhecimento da epidemiologia da coqueluche em nosso meio por meio de análise de dados secundários dos casos suspeitos de coqueluche no Estado de São Paulo, cujos materiais biológicos foram encaminhados ao CB do IAL, para realização do diagnóstico laboratorial pelas técnicas de cultura e da qPCR.
MATERIAL E MÉTODOS
Amostras de swab nasofaríngeo, provenientes da assistência do Estado de São Paulo e encaminhadas ao IAL Central e aos Centros dos Laboratórios Regionais do IAL (CLR – IAL) entre janeiro/2010 a dezembro/2020, para a rotina diagnóstica de coqueluche. As informações obtidas foram organizadas em bancos de dados no Núcleo de Doenças Entéricas e Infecções por Patógenos Especiais – NDEI do CB/IAL. Não foram incluídas informações que permitissem identificar os pacientes, de forma a garantir a privacidade das informações e o anonimato dos sujeitos da pesquisa, utilizando-se os dados obtidos exclusivamente para os propósitos desta pesquisa. As variáveis que foram consideradas para a análise dos dados foram: ano calendário, área geográfica, faixa etária, sexo e resultados da cultura e qPCR.
RESULTADOS E DISCUSSÃO
Foram processadas 39.140 amostras de casos suspeitos de coqueluche no estado entre o período estudado.
Na figura 1, temos que a técnica de qPCR foi capaz de detectar um maior número de casos positivos em relação a cultura, 5.289 (13,51%) e 1.596 (4,1%) (somatória do período) para qPCR e cultura respectivamente, demonstrando a maior sensibilidade da qPCR entre as duas técnicas. Essa maior sensibilidade tem sido bastante evidenciada e relatada por diversos grupos ao redor do mundo. Um estudo realizado por LEE et al. (2018) [9] nos Estados Unidos, foi verificado a sensibilidade de diversas técnicas diagnósticas para a coqueluche dentre elas a qPCR e a cultura, os dados foram coletados de sete Estados entre os anos de 2007 e 2011, e os resultados demonstraram que a cultura apresentou 64% de sensibilidade enquanto a qPCR apresentou 90,6% do total de 868 amostras analisadas.
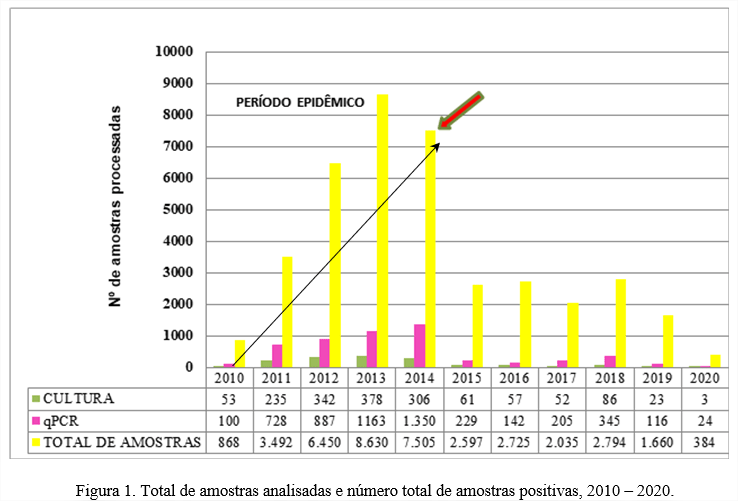
Observamos um aumento gradual da confirmação de casos suspeitos por qPCR, sendo o ano de 2014 o de maior positividade no Estado de São Paulo, correspondendo a 25,5% (1.350) do total de casos positivos por qPCR. Esses dados corroboram com os dados epidemiológicos do Brasil onde os anos de 2010-2014 foram considerados os anos do período epidêmico no país e no mundo.
O Estado da Califórnia, nos Estados Unidos, notificou um aumento no número de casos de coqueluche em 2014, tendo um aumento de cinco vezes mais registrado em anos anteriores, com um total de 9.935 casos reportados, descrevendo um caráter epidêmico cíclico (três a cinco anos) no aumento de número de casos confirmados [13]
Ainda podemos observar que o ano de 2018 deveria corresponder ao início do próximo período epidêmico, no entanto isso não se confirmou já que ocorreu uma queda nos casos confirmados e também no número de amostras processadas. Não se pode confirmar exatamente o porquê de não ter havido uma nova epidemia de coqueluche, mas o que se supõe é que a queda dos casos suspeitos e positivos seja um reflexo dos efeitos da vacinação de gestante, mas não se tem estudos suficientes para confirmar esta hipótese.
O ano de 2020 foi o período com menor número de amostras processas e também de resultados positivos, o que pode ser relacionado com a pandemia de Covid-19, já que nesse ano toda a atenção estava voltada para está doença o que fez com que o diagnóstico para outras doenças respiratórias tenha ficado em segundo plano e também o uso de máscara pode ter levado a diminuição da transmissão de outras doenças respiratórias influenciando a queda nas amostras processadas.
Na figura 2, observamos a distribuição das faixas etárias obtidas na análise dos resultados positivos da qPCR. O grupo etário de maior positividade corresponde ao das crianças com idade menor ou igual a dois meses, 2.411 (45,6%) amostras positivas. Essa faixa etária corresponde às crianças que ainda não receberam nenhuma dose de imunizante, sendo o grupo mais vulnerável, seguido pelo grupo de três a seis meses, com 1.235 (23,4%), ou seja, dos incompletamente vacinados.
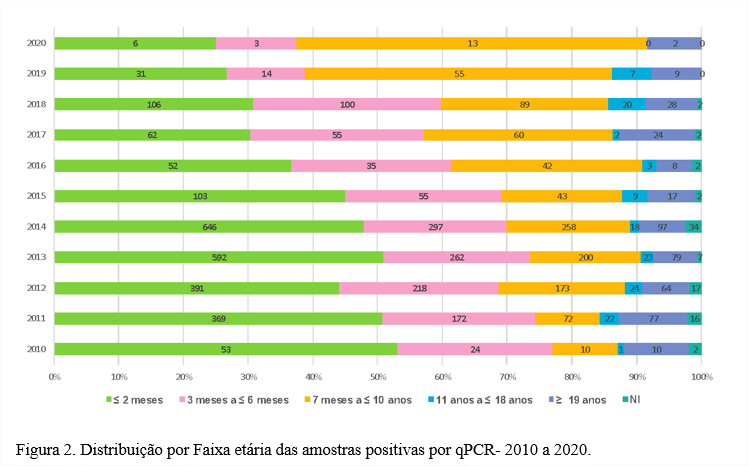
Entre o período de 2011 a 2014, o Ministério da Saúde (2020) registrou 22.772 casos positivos de coqueluche em todo o Brasil, onde 61% (13.935) desses casos em crianças menores de um ano de vida, sendo ainda 12.135 menores de seis meses, com esquema vacinal não iniciado ou incompleto, o que está de acordo com o observado em São Paulo [14].
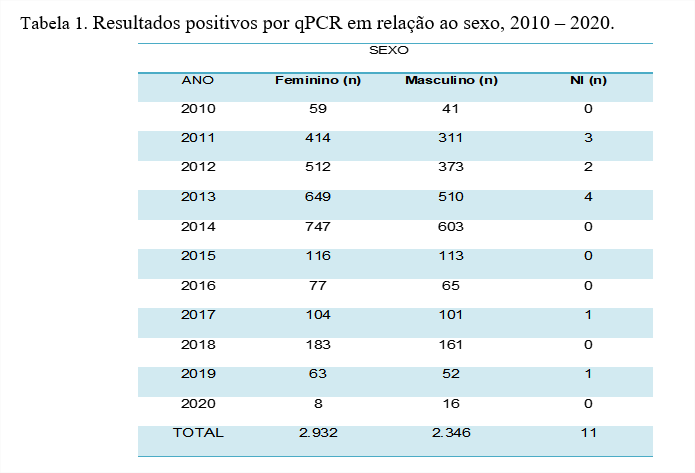
Na tabela 1, a análise do sexo, mostrou que 55,4% (2.932) dos resultados positivos correspondiam ao sexo feminino, enquanto 44,4% (2.346) ao masculino, 0,2% ao grupo de sexo não informado (ou seja, recém-nascidos). Apesar de pequena a diferença de positividade entre os sexos, existe um predomínio entre as mulheres. Essa proporção também foi registrada por PEER et al. (2020), no qual foi feito um levantamento da incidência do sexo feminino e masculino em relação aos casos positivos em nove países no período entre os anos de 1990- 2017, variando a positividade entre os países, 48,1% e 54,7% para sexo masculino e feminino, respectivamente [15].
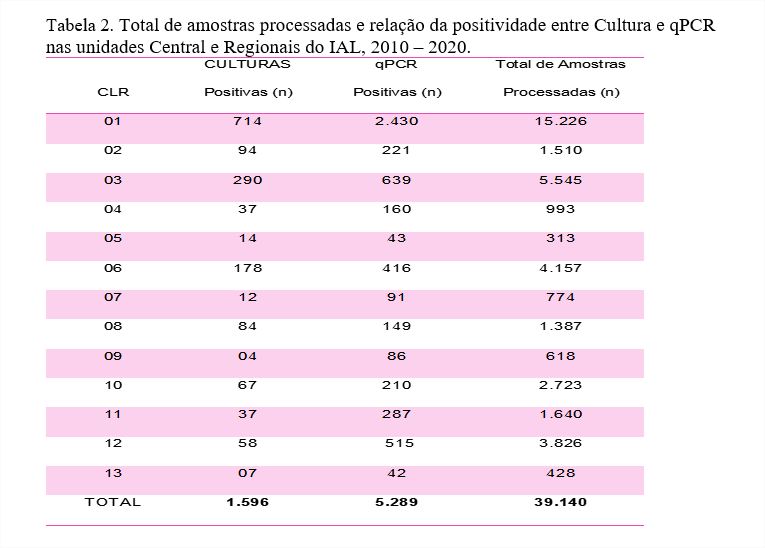
Na tabela 2 temos a análise da realização de exames processados em cada CLR – IAL nos 11 anos de estudo. O IAL Central (01) foi o que processou mais amostras e obteve o maior número de resultados positivos, correspondendo a 38,9% do total de amostras processadas entre 2010 e 2020, sendo seguido pelo CLR de Campinas (03), CLR de Ribeirão Preto (06) e CLR de Taubaté (12), com respectivamente 14,2%; 10,6% e 9,8% de amostras processadas. Presume-se que o alto número de amostras processadas seja atribuído ao intenso trabalho de vigilância epidemiológica e a presença de hospitais sentinelas nessas regiões o que permite um melhor rastreio de casos suspeitos e notificação dos casos positivos.
CONCLUSÕES
A análise dos dados epidemiológicos da coqueluche no Estado de São Paulo nos 11 anos estudados permitiu verificar que é um importante agravo em saúde pública. Apesar de ser uma doença imunoprevenível, ainda existem surtos e epidemias cíclicas, como a ocorrida entre 2010-2014. Assim como, verificou-se que a qPCR é uma ferramenta diagnóstica muito importante, quando comparada à cultura, pois possui maior sensibilidade e especificidade. Mas apesar de suas vantagens em relação à cultura, a qPCR é uma técnica auxiliar no diagnóstico, sendo a cultura necessária no diagnóstico pois sem ela seria impossível a realização de estudos epidemiológicos. E em relação à faixa etária, também se pode observar que a faixa mais atingida são as crianças menores de seis meses, ou seja, não imunizadas. Esse grupo é o mais vulnerável, mais exposto a adoecer e vir a óbito, sendo necessária a imunização materna para que os anticorpos produzidos ajudem a proteger esse grupo nos primeiros meses de vida.
REFERÊNCIAS
[1] MATTOO, S; CHERRY, J.D. Molecular pathogenesis, epidemiology, and clinical manifestations of respiratory infections due to Bordetella pertussis and other Bordetella subspecies. Clin Microbiol Rev, v. 18, n. 2, p. 326–382, 2005.
[2] CALVERT, A.; HEATH, P.T. Pertussis. Medicine, v. 45, n. 12, p. 735-738, 2017.
[3] MS. Ministério da Saúde. Coqueluche. 2020. Disponível em: https://www.gov.br/saude/pt-br/assuntos/saude-de-a-a-z/c/coqueluche Acesso em: 09 de dez. de 2021.
[4] NIEVES, D.J.; HEININGER, U. Bordetella pertussis. Microbiol Spectr, v. 4, n. 3, EI10, 2016.
[5] WHO.World Health Organization. Pertussis. 2019. Disponível em: who.int/health-topics/pertussis#tab=tab_1. Acesso em 05 de agosto de 2021.
[6] OLIVEIRA, D.R.; LEITE, D.; BEREZIN, E.; RENOINER, E.I.M.; ROSA, F.M.; LOBO F.C.B.; et al. Coqueluche. In: Ministério da Saúde, Guia de Vigilância em Saúde, Ministério da Saúde, 3°ed, v. único. Brasília, DF, 2019, p.70 -82.
[7] KILGORE, P.E; SALIM, A.M.; ZERVOS, M.J.; SCHMITT, H-J. Pertussis : Microbiology, Disease, Treatment, and Prevention. Clin Microbiol Rev, v. 29, n. 3, p. 449– 486, 2016.
[8] POLINORI, I.; ESPOSITO, S. Clinical Findings and Management of Pertussis. In: FEDELE, G.; AUSIELLO, C. (eds) Pertussis Infection and Vaccines. Advances in Experimental Medicine and Biology, v. 1183. Springer, Cham., 2019.
[9] LEE, AD.; CASSIDAY, P.K.; PAWLOSKI, L.C.; TATTI, K.M.; MARTIN, M.D.; BRIERE, E.C.;et al. Clinical evaluation and validation of laboratory methods for the diagnosis of Bordetella pertussis infection: Culture, polymerase chain reaction (PCR) and anti- pertussis toxin IgG serology (IgG-PT). PLoS ONE, v. 13, n. 4, e0195979, 2018.
[10] LEITE, D. Prevalência e caracterização de cepas de Bordetella pertussis deficientes em pertactina em um país com vacina de células inteiras contra coqueluche. 2019. 102f. Tese (Doutorado em Ciências). Coordenadoria de Controle de Doenças da Secretaria de Estado da Saúde de São Paulo.
[11] GUISO, N.; MEADE, B.D.; WIRSING VON KÖNIG, C-H. Pertussis vaccines: The first hundred years. Vaccine, v. 38, n. 5, p. 1271-1276, 2020.
[12] MS. Ministério da Saúde. Brasil. Nota informativa sobre mudanças no calendário nacional de vacinação para o ano de 2017. 2016
[13] WINTER, K.; GLASER, C.; WATT, J.; HARRIMAN, K. Pertussis Epidemic. MMWR, v.63, n. 48, p. 1129–1132, 2014.
[14] MS. Ministério da Saúde. Brasil. Nota informativa sobre mudanças no calendário nacional de vacinação para o ano de 2017. 2016
[15] PEER, V.; SCHWARTZ, N.; GREEN, M.S. A multi-country, multi-year, meta-analytic evaluation of the sex differences in agespecific pertussis incidence rates. PLoS ONE, v. 15, n. 4, e0231570,2020.
Resumo expandido publicado nos Anais da III Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
Este trabalho foi escrito por:
Jéssica Silva Santos¹; Elizabeth Natal de Gaspari²
¹Estudante do Curso de Vigilância Epidemiológica Laboratorial em Saúde Pública- CIM- Instituto Adolfo Lutz; E-mail: [email protected]
²Pesquisadora do Centro de Imunologia- CIM- Instituto Adolfo Lutz
Resumo: A resposta imune a vacinas, importante ferramenta em saúde pública, é amparada por ensaios laboratoriais, como a avaliação de anticorpos. Testes que indiquem a funcionalidade dos anticorpos são importantes, pois demonstram as propriedades dessas imunoglobulinas. Tratando-se da doença meningocócica invasiva, o teste funcional para correlato de proteção é o ensaio de atividade bactericida sérica, que revela a capacidade das imunoglobulinas de lisar o meningococo. Porém, este ensaio tem aplicação limitada devido ao elevado nível de biossegurança e refinamento técnico para manipulação da cepa viva, tempo de execução e custo. Em contrapartida, o ensaio de avidez indica a força de ligação entre o antígeno e os anticorpos, sendo capaz de sugerir a atividade bactericida do soro, e pode ser realizado a partir de uma técnica simples de ELISA modificado. Todavia, o protocolo utilizado pode interferir nos resultados finais. Neste trabalho, serão comparados os efeitos de agentes caotrópicos e diferentes temperaturas de incubação nos resultados de ensaio de avidez com soro de camundongos imunizados com antígenos de Neisseria meningitidis, a fim de se destacar a importância da padronização desse ensaio, que pode ser utilizado como ferramenta para estudo de vacinas.
Palavra-chave: agente caotrópico; avidez; doença meningocócica; vacinas
INTRODUÇÃO
Neisseria meningitidis é uma bactéria pertencente à família Neisseriaceae, gram-negativa, fastidiosa, em formato de diplococos, aeróbico e que pode ou não apresentar cápsula. Seu crescimento ideal necessita de uma temperatura entre 35 a 37ºC com 5-10% (v/v) de CO2 (dióxido de carbono), sua cultura pode ocorrer nos meios: ágar sangue, ágar triptona soja, ágar chocolate suplementado e ágar Mueller-Hinton (ROUPHAEL; STEPHENS, 2011).
A bactéria N. meningitidis apresenta alguns fatores de virulência que otimizam sua capacidade de invasão e aumentam sua sobrevivência no organismo hospedeiro, um dos fatores de virulência é a cápsula que auxilia na invasão em ambientes estéreis como o sangue e o líquido cefalorraquidiano protegendo a bactéria contra a resposta do sistema imunológico (SIDDIQUI; AMEER; GULICK, 2021). Além disso o meningococo é envolto por uma membrana externa que apresenta pili, proteínas de membrana externa e lipooligossacarídeos (LOS) que de acordo com sua constituição de antígenos expressos irá classificar a bactéria nos 12 sorogrupos: A, B, C, E, H, I, K, L, M, W, X, Y e Z, sendo que os de maior patogenicidade são o A, B, C, W e Y responsáveis por 90% dos casos da doença meningocócica invasiva (ROMANELLI, 2012; PIZZA; BEKKAT-BERKANI; RAPPUOLI, 2020).
A doença meningocócica invasiva (DMI) pode ser potencialmente mais grave e fatal na infância, seguida pela ocorrência na adolescência e por fim em jovens adultos, a infecção por Neisseria meningitidis pode resultar em choque séptico se manifestando como meningite, septicemia ou uma associação de ambas com acometimento súbito e potencialmente fatal entre 24 a 48 horas do início dos sintomas e com taxa de letalidade de 20%, mesmo em casos de recuperação pode permanecer sequelas como a perda auditiva, doenças no neurodesenvolvimento e amputações (SIDDIQUI; AMEER; GULICK, 2021) (PIZZA; BEKKAT-BERKANI; RAPPUOLI, 2020).
A vacinação é uma medida profilática que se fundamenta no reconhecimento dos polissacarídeos capsulares associados ou não adjuvantes, atualmente estão disponíveis contra os sorogrupos A, C, W e Y. O sorogrupo B é o objeto de intensas pesquisas no atual momento, uma vez que apresenta dificuldade na elaboração de uma vacina por possuir a estrutura do seu polissacarídeo semelhante ao ácido polisiálico presente nas células humanas, gerando o risco de estimular uma resposta autoimune através da vacinação para este sorogrupo (ANDREWS; POLLARD, 2014).
O Programa Nacional de Imunização (PNI) foi criado em 18 de setembro de 1973 por determinação do Ministério da Saúde, com o objetivo de coordenar as ações de imunização. Em 1975 o PNI é institucionalizado por fatores nacionais e internacionais que estimulam a utilização e expansão dos agentes imunizantes, com o objetivo de garantir a integridade das ações de imunização no país e é fortalecido pela legislação sobre imunizantes e vigilância epidemiológica Lei 6.259 de 30-10-1975 e Decreto 78.231 de 30-12-1976 (MINISTÉRIO DA SAÚDE, 2003)
A avidez é dada através da interação bivalente e multivalente entre um epítopo e um anticorpo. Sua análise é importante uma vez que pode ser empregada como critério auxiliar para avaliação da eficácia da resposta a vacinas, uma vez que anticorpos que apresentam alta afinidade podem gerar uma função otimizada e ser indicativos de anticorpos com maior potencial protetor (CORRÊA et al., 2021). O ensaio de ELISA é o método mais simples e rápido para análise do índice de avidez e por isso é a metodologia mais comum empregada, entretanto, outras técnicas também permitem a análise do índice de avidez como a precipitação com sulfato de amônio, radioimunoensaio em fase sólida e ressonância plasmônica de superfície (CORRÊA et al., 2021).
O tiocianato de potássio (KSCN) é um dos principais agentes caotrópicos empregado nos estudos de nosso laboratório, entretanto é descrito na literatura o uso de Ureia e Dietanolamina como agentes caotropicos para a análise do índice de avidez. Diante do exposto, este trabalho tem como objetivo analisar se há diferenças na efetividade de ação dos três agentes caotrópicos citados e analisar se a exposição em diferentes temperaturas poderia ser um fator de otimização da reação.
MATERIAIS E MÉTODOS
A cepa de N. meningitidis B:4:P1.9 empregada no ensaio se originou através da amostra clínica de um indivíduo brasileiro que desenvolveu a doença invasiva durante uma epidemia de Meningococo B e foi isolada pelo Centro de Bacteriologia do Instituto Adolfo Lutz. A partir desta cepa obteve-se as preparações antigênicas, vesículas de membrana externa (OMVs) empregadas na imunização, suspensões de células integras e ensaios laboratoriais. As cepas liofilizadas foram reativadas em ágar chocolate (Difco Laboratories) e então, cultivadas em caldo soja tripticaseína (TSB) (Difco Laboratories), suplementado com 1% de soro de cavalo (Sigma Chemical Company), a 37ºC, durante 24 horas. Para extrair as suspensões de células íntegras, as placas foram cobertas com tampão-fosfato salino (PBS) (pH 7,4)-0,02% azida sódica e homogeneizou-se as células gentilmente com alça de Drigalski. A suspensão foi transferida para tubos e inativada por calor, em banho-maria 56ºC por 30 minutos. Mensurou-se a densidade ótica (DO) a 600 nm e conservou-se a suspensão a 4ºC. A extração das OMVs seguiu a descrição de De Gaspari e Zollinger (2001).
O estudo utilizou amostras provenientes de ensaio que comparou a imunização de camundongos de meia-idade com OMVs complexadas a hidróxido de alumínio (HA) ou brometo de dioctadecildimetilamônio (DDA) como adjuvantes (TRZEWIKOSWKI DE LIMA et al., 2021), conforme aprovação pelo Comitê de Ética e Uso Animal (CEUA) IAL-Pasteur, nº 06/2012. Utilizou-se camundongos (Mus musculus), fêmeas, da linhagem heterogênea Swiss (não isogênicos), com 12 meses de vida (meia-idade), provenientes do Biotério de Criação do Instituto Adolfo Lutz (São Paulo, SP, Brasil). Imunizou-se os camundongos com 3 doses subcutâneas de 100 µL de preparações antigênicas descritas na tabela 1, nos dias 0, 20 e 35. As amostras utilizadas neste estudo foram coletadas por punção do plexo oftálmico nos dias 0 (controle pré-imune) e no dia 50, ou seja, 15 dias após a última dose. Após coleta, o sangue foi separado por centrifugação e armazenou-se o soro a -20ºC. A obtenção e caracterização das preparações antigênicas está descrita em Trzewikoswki de Lima et al. (2021).

Para determinação da avidez utilizou-se o ensaio de ELISA indireto empregando placas MaxiSorp Loose Nunc-immuno module. A placa foi sensibilizada com antígeno Whole Cells diluídas em tampão fosfato-salino (PBS) até a densidade óptica de 0,1nm. As lavagens foram realizadas com solução de PBS-tween 0,05% e o bloqueio realizado com diluição 5% de leite-PBS-tween 0,05%. Como amostra, empregou-se soro de camundongos da linhagem swiss imunizados com a terceira dose para OMV de N. meningitidis diluídos 1:3200. Utilizou-se como agentes caotrópico o Tiocianato de potássio (KSCN), ureia e Dietanolamina (DEA) incubando em câmara úmida e expondo nas temperaturas 4ºC, temperatura ambiente (TA) e 37ºC. Como anticorpo conjugado utilizou-se Anti-IgG de camundongos marcado com peroxidase cadeia pesada específico (γ) da KPL (Lote: 10219636) diluído 1:20.000 em PBS-tween em leite 2,5%. Empregou-se como substrato cromogênico a tetrametilbenzidina (TMB) e para solução stop ácido sulfúrico (H2SO4).
RESULTADOS E DISCUSSÃO
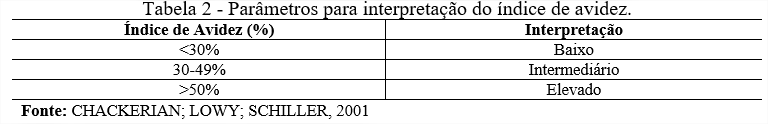
Para determinação do índice de avidez foi medido a resistência dos complexos de antígeno-anticorpo em exposição aos agentes KSCN, ureia e DEA em condições de temperatura de 4°C, temperatura ambiente e 37ºC por 20 minutos. O índice de avidez foi calculado realizando a média das duplicatas das densidades óptica (DO) das amostras e posteriormente realizando o cálculo para o índice de avidez que é dado pela divisão da DO do soro tratado com o agente caotrópico dividido pela DO do soro sem tratamento e o resultado multiplicado por 100. A interpretação do resultado obtido é representada pela tabela 2.
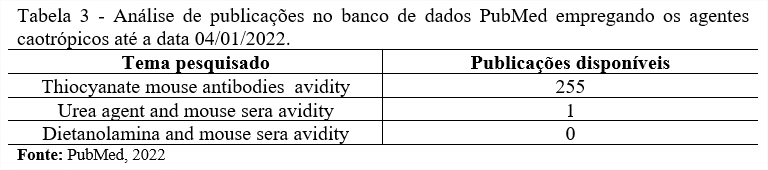
O levantamento das publicações disponíveis na base de dados do PubMed até a data de 04/01/2022 demonstrou uma prevalência no uso do Tiocianato de Potássio, enquanto outros agentes caotrópicos como Ureia e Dietanolamina não se apresentam tão expressivos para o uso no modelo camundongo, sugerindo assim a prevalência do uso do KSCN nos ensaios de avidez, corroborando com nossos atuais protocolos de laboratório. Sendo assim, podemos sugerir que há uma maior utilização do uso deste agente caotrópico na comunidade científica, permitindo a abertura de questões a respeito da possível obtenção de resultados mais satisfatórios e reprodutíveis em relação a outros agentes caotrópico existentes para camundongos (Tabela 3).
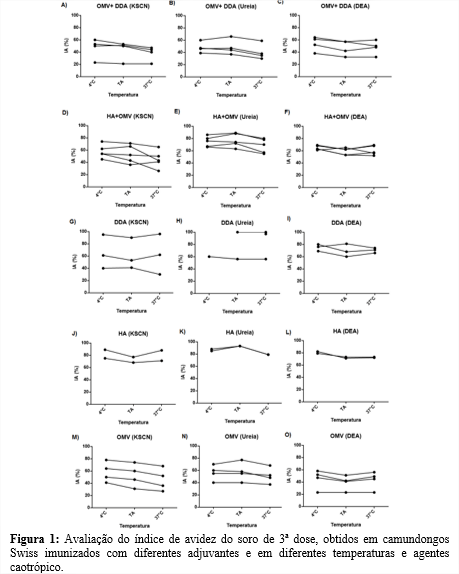
Os grupos experimentais foram delimitados de acordo com o tipo de adjuvante utilizado ou sua ausência compondo os grupos: OMV+ DDA (N=4), HA+OMV (N=5), DDA (N=3), HA (N=2), OMV (N=4). Os dados obtidos com esses ensaios preliminares demonstraram a utilização do KSCN como agente caotrópico não apresentou diferenças significativas ao uso dos diferentes agentes caotrópico utilizados. A alteração mais significativa ocorreu na amostra de soro de camundongos quando imunizados com HA+OMV, onde se obtêm um índice de avidez elevado (54%) na temperatura de 4ºC, um índice intermediário (43%) na temperatura ambiente e um índice baixo (26%) na temperatura de 37°C, podendo assim se atribuir o uso da temperatura de 37ºC a protocolos já estabelecidos. Entretanto essas condições não se reproduzem nos outros grupos imunizados com outros adjuvantes, podendo ser uma possível característica da heterogeneidade do adjuvante utilizado e da espécie dos animais Swiss, tão bem quanto pode estar vinculado a quantidade de anticorpos presente na amostra (CORRÊA, 2021).
Diferentemente dos outros dois agentes caotrópico, mesmo com a exposição a três temperaturas distintas a ureia não apresentou em nenhuma das amostras índices de avidez baixos, ou seja, inferiores a 30%. Também foi observado que algumas respostas a imunização apresentaram índices de avidez extremamente altos, atingindo 100%, mas tal resultado pode ser justificado em virtude do tipo de cálculo utilizado, pois os valores com as amostras sem tratamento ficaram próximos aos das amostras tratadas. Apesar das análises realizadas ainda se trata de observações iniciais sem cálculo estatístico e passível das limitações cabíveis da análise de índice de avidez utilizado. Em relação ao dietanolamina apenas um animal imunizado com OMV apresentou índice de avidez baixo e sem alteração na exposição nas diferentes temperaturas. Os resultados obtidos com a avaliação da avidez dos grupos supracitados são expressos na figura 1.
CONCLUSÃO
Com base nos resultados que obtivemos podemos observar a possibilidade de expandir os estudos empregando a primeira e segunda dose dos soros utilizados para avaliar se ocorreu diferença de avidez em períodos diferentes de imunização. Os agentes caotrópico inicialmente não apresentam grandes diferença entre si, mesmo aplicando temperaturas diversas. Os levantamentos da literatura indicam uma prevalência no uso de tiocianato de potássio para avaliação de soro de camundongos, possibilitando um questionamento a respeito de uma maior afinidade deste agente caotrópico com o tipo de amostra utilizada em relação aos demais estudados. A aplicação de apenas uma metodologia de análise de afinidade aparenta não ser suficiente para gerar resultados conclusivos a respeito das questões geradas no decorrer deste trabalho, necessitando assim de mais estudos acerca do tema.
REFERÊNCIAS
ANDREWS, Sophie M.; POLLARD, Andrew J. A vaccine against serogroup B Neisseria meningitidis: dealing with uncertainty. The Lancet Infectious Diseases, v.14. n. 5, p.426-434, 2014.
CORRÊA, Victor Araujo et al. Modified ELISA for antibody avidity evaluation: The need for standardization. Biomedical Journal, v. 44, n. 4, p.433-438, 2021.
De GASPARI, E.; ZOLLINGER, W. Expression of class 5 antigens by meningococcal strains obtained from patients in Brazil and evaluation of two new monoclonal antibodies. Brazilian Journal of Infectious Diseases, v. 5, n. 3, p. 143-153, 2001.
MINISTÉRIO DA SAÚDE. Programa Nacional de Imunizações. Brasilia: Secretaria de Vigilância em Saúde, 2003
PIZZA, Mariagrazia; BEKKAT-BERKANI, Rafik; RAPPUOLI, Rino. Vaccines against Meningococcal Diseases. Microorganisms. V.8, n.10, 2020.
ROMANELLI, C. S. S. Prevalência e variabilidade genética de antígenos candidatos vacinais em isolados clínicos de N. meningitidis circulantes no Brasil. 2012. 95 f. Dissertação (Mestrado em Vigilância Sanitária). Instituto Nacional de Controle de Qualidade em Saúde, Fundação Oswaldo Cruz, Rio de Janeiro, 2012.
ROUPHAEL, Nadine G; STEPHENS, David S. Neisseria meningitidis: biology, microbiology, and epidemiology. Methods in molecular biology, v. 799, p. 1-20, 2011
SIDDIQUI, J. A; AMEER, M.A; GULICK, Peter G. Meningococcemia. StatPearls, 2021. TRZEWIKOSWKI DE LIMA, G.; RODRIGUES, T.S.; PORTILHO, A.I.; et al. Immune responses of meningococcal B outer membrane vesicles in middle-aged mice. Pathogens and Disease, v. 78, n. 5, fta 0028, 2021.
Resumo expandido publicado nos Anais da III Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
Este trabalho foi escrito por:
Rafaela Nascimento Cont¹; Gabriela Bastos Cabral²
¹Estudante do Curso de Especialização em Vigilância Laboratorial em Saúde Pública – CLR IAL; E-mail: [email protected]
² Núcleo de Ciências Biomédicas do Instituto Adolfo Lutz Regional de Santos, SP
Resumo: A febre Chikungunya, é a doença causada pela infecção do vírus Chikungunya, um alphavirus, da família Togaviridae, transmitida por mosquitos Aedes aegypti e Aedes albopictus, que circula no Brasil desde 2014.No ano vigente, até setembro, ocorreram 87.748 casos prováveis, e 10 óbitos confirmados por critérios laboratoriais. A co-circulação de arboviroses, como Dengue e Zika, é uma realidade em muitas regiões, o que dificulta o diagnóstico clínico, devido a semelhança dos sintomas entre essas doenças. Para um diagnóstico mais específico, utiliza-se os testes laboratoriais. Entretanto, um dos desafios primários na interpretação dos testes sorológicos de infecções agudas por arboviroses, são os resultados falsos-positivos devido a reatividade-cruzada. Através de uma revisão bibliográfica, demonstrou-se a ocorrência de reação-cruzada nos testes sorológicos de Chikungunya em diversos estudos, variando de 1% a 37%, dependendo do kit comercial e da quantidade de amostras utilizados. Além disso, apresentamos estudos para o desenvolvimento de testes capazes de minimizar essa ocorrência.
Palavras-chave: arboviroses; chikungunya; ELISA; reação-cruzada
INTRODUÇÃO
A febre Chikungunya (CHIKF), é a doença causada pela infecção do vírus Chikungunya (CHIKV), um alphavirus, da família Togaviridae, transmitida por mosquitos Aedes aegypti e Aedes albopictus principalmente. O CHIKV foi isolado pela primeira vez na década de 50, na Tanzânia, onde originou-se o termo Chikungunya, derivada da língua Makonde, que significa “encurvado”, referindo-se a forma que o indivíduo infectado fica devido as dores nas articulações(MORCERF et al., 2015).
No Brasil, os anos de maior incidência de casos de CHIKF até o ano vigente, foram 2016 e 2017, com mais de 151.000 casos/ano, e mais de 190 óbitos (BRASIL, 2018). Em 2021, foi emitido um alerta sobre a ocorrência de surto de Chikungunya no estado de São Paulo, localizado mais especificamente na região da Baixada Santista, com aumento de 380% nas notificações e 33 vezes mais casos confirmados comparado ao ano anterior (SÃO PAULO, 2021).
Assim como a CHIKF, outras arboviroses, como Dengue, Zika e Febre amarela, existem há séculos, e são encontrados frequentemente, porém não exclusivamente, em regiões equatoriais e subtropicais do planeta. Ao longo da história, observa-se o aumento dos casos de surtos e epidemias ao redor do mundo, justificados pela globalização, adaptação dos vetores e falhas no controle das doenças, tornando-se assim uma problemática de saúde pública (FERREIRA et al., 2021; ZAID et al., 2021).
Uma realidade em diversos países, é a co-circulação dessas doenças, o que traz alguns desafios para um diagnóstico adequado. Por apresentarem sintomas muito similares, o diagnóstico clínico é dificultoso, exigindo assim a confiabilidade diagnóstica dos testes laboratoriais (NATRAJAN; ROJAS; WAGGONER, 2019; LIMA et al., 2021).
Por ser uma região endêmica para outras arboviroses, principalmente a dengue que causa epidemias no país há mais de 40 anos, se faz necessário a compreensão sobre a capacidade dos testes diagnósticos em diferenciar essas doenças. O objetivo deste estudo foi realizar um levantamento bibliográfico sobre os principais intercorrentes e desafios encontrados no diagnóstico sorológico da infecção pelo Chikungunya.
MATERIAL E MÉTODO
Foi realizada uma revisão bibliográfica priorizando literaturas publicadas nos últimos 5 anos, utilizando bases de dados da National Center for Biotechnology Information (NCBI – PubMed). Artigos que abordavam o tema da febre Chikungunya, como: diagnóstico sorológico, reação-cruzada e co-circulação de arboviroses foram incluídos no estudo.
RESULTADO E DISCUSSÃO
Para um diagnóstico mais específicos, utiliza-se os testes laboratoriais. Atualmente, as duas principais formas de diagnóstico de CHIKV e diferenciação com outras doenças são os testes moleculares e exames sorológicos. A partir dos testes moleculares temos a detecção do RNA viral por RT-PCR (Reação em Cadeira da Polimerase com Transcriptase Reversa), até o quinto dia após o início dos sintomas. Já os testes sorológicos, detecta-se a presença de anticorpos IgM/IgG após o sexto dia do início dos sintomas (BRASIL, 2017).
Sabe-se que a detecção de anticorpos específicos é de suma importância na infecção por arboviroses. A curta duração da viremia, torna os testes sorológicos a melhor opção para o diagnóstico, visto que dificilmente os pacientes procuram o auxílio médico no início dos sintomas. Assim, algumas das técnicas de diagnóstico sorológico que podem ser utilizados são ELISA, MAC-ELISA, testes imunocromatográficos (teste rápido) e o teste de neutralização por redução de placas (PRNT) por exemplo (FISCHER et al., 2021).
O teste padrão ouro para confirmação pela sorologia é o teste de neutralização (PRNT). Neste método, podemos observar o grau de infecção celular, após a incubação do vírus com anticorpos neutralizantes presentes no soro. O resultado fornece uma estimativa direta da quantidade de anticorpos funcionais. Seus dados podem ser úteis principalmente na detecção de casos subclínicos, auxiliando na avaliação do impacto da doença, na compreensão do comportamento do vírus e no desenvolvimento de vacina, entretanto, é uma metodologia dispendiosa, tanto de custos como de tempo, é de difícil acesso e interpretação subjetiva (MARQUES et al., 2017; PIANTADOSI; KANJILAL, 2020).
O IgM Antibody Capture Enzyme-linked immunosorbent assay (MAC-ELISA) é usado para uma detecção mais específica de IgM. Comparado ao ELISA, a competição de IgG é menor, reduzindo o risco de ligações não-específicas. Em uma avaliação do Center for Disease Control and Prevention (CDC), os resultados positivos no MAC-ELISA, tiveram 90% de concordância com PRNT (JOHNSON; RUSSELL; GOODMAN, 2016; PIANTADOSI; KANJILAL, 2020).
A partir dessas considerações, os testes mais avaliados, utilizados e disponíveis são, portanto, ELISA e o teste rápido. Segundo recomendações do Ministério da Saúde, os testes imunocromatográficos devem ser utilizados como técnicas de triagem, dessa forma, quando um resultado é positivo, a confirmação por ELISA é necessária (BRASIL, 2017).
Segundo Piantadosi e Kanjilal (2020), um dos desafios primários na interpretação dos testes sorológicos de infecções agudas por arboviroses, são os resultados falsos-positivos devido a reatividade-cruzada.
A reação-cruzada é definida como a habilidade de elementos estruturalmente diferentes realizarem a mesma função. Agentes infecciosos frequentemente compartilham similaridade molecular com antígenos-próprios, os quais os auxiliam no escape do sistema imune do hospedeiro. Assim como também pode ocorrer reação-cruzada entre epítopos de patógenos diferentes, possibilitando a neutralização de ambos, o que pode ser uma boa escolha para o desenvolvimento de vacinas contra essas duas ou mais doenças (ROJAS et al., 2018; JAIN; SALUNKE, 2019).
A provável razão da ocorrência dessa multi-especificidade nos anticorpos, é a existência de epítopos comuns em diferentes antígenos. O mimetismo molecular é a semelhança sequencial, estrutural, química ou imunológica compartilhada entre dois epítopos que os leva a interagir com o mesmo paratopo, ademais os anticorpos podem utilizar diferentes paratopos para a ligação com epítopos semelhantes. Essa promiscuidade na ligação de anticorpos contra antígenos diferentes não está diretamente relacionada a especificidade e nem com a interação de baixa afinidade desses anticorpos segundo Jain e Salunke (2019).
A reação-cruzada entre vírus de famílias diferentes, como o caso do CHIKV com o DENV ou ZIKV, é muitas vezes reportado como um evento improvável, devido suas diferenças estruturais e antigênicas. Entretanto, há alguns estudos que demonstram essa ocorrência.
Lima et al. (2021) reportou em seu estudo, a reatividade-cruzada com amostras positivas para Dengue em um kit comercial ELISA CHIKV IgM (Euroimmun). As amostras para Dengue, foram coletadas antes da entrada do CHIKV no Brasil, excluindo a possibilidade de co-infecção, além da retestagem dessas amostras com testes moleculares e sorológicos. No geral, 37% (58 de 155 amostras) dos pacientes positivos para DENV também positivaram no ELISA CHIKV IgM.
Em uma pesquisa realizada na Tailândia por Suwanmanee et al. (2018), 6 amostras positivas para DENV (RT-PCR), no total de 129 amostras (4,6%), apresentaram reação-cruzada com CHIKV, mais especificamente amostras do sorotipo DENV-2 e DENV-3.
Na análise para o desenvolvimento de um teste rápido diferencial para DENV e CHIKV, Wang et al. (2019) observou a presença de alta reatividade-cruzada entre proteínas do envelope do sorotipo DENV-3 e DENV-4, com anticorpos anti-CHIKV. Essas proteínas então, não foram utilizadas para compor o teste, limitando um pouco os resultados, porém evitando ligações equivocadas.
No estudo de Mendoza et al. (2019), uma análise de reação-cruzada foi realizada entre diferentes kits de ELISA para CHIKV. Em amostras positivas para flavivírus, confirmados por PRNT, a porcentagem de falsos-positivos para os teste da Euroimmun IgM, Euroimmun IgG e Abcam IgG foram 10,7%, 1,2% e 1,2% respectivamente. Neste mesmo estudo, reação-cruzada intrafamiliar (4%) para os kits da Euroimmun (IgM e IgG) também foram observados.
Quando se utiliza epítopos mais específicos como base do ELISA, como utilizou Kam et al. (2015), a porcentagem de falsos-positivos para flavivírus é relativamente baixa (7% – 4/60 amostras), porém para vírus do mesmo gênero, no caso Alphavirus, devido à proximidade filogenética, mais da metade dos pacientes com alphaviroses tiveram ELISA positivo, e 50% dessas amostras também apresentaram PRNT reagente.
No cenário epidemiológico atual do Brasil, não há outro alfavírus de grande circulação no país, como o CHIKV. Entretanto, o virus Mayaro (MAYV), do mesmo gênero, é endêmico na região Amazônica, e a ocorrência de reatividade cruzada foi demostrada por Fischer et al. (2020) e Fumagalli (2018). Portanto, é importante a constante atenção a essa doença também, sendo essenciais medidas de prevenção e controle, através de notificações e desenvolvimento de diagnósticos diferenciais(BARROS et al., 2021).
A proposta de Damle et al. (2016) foi desenvolver um anticorpo anti-proteina C, para sua utilização no MAC-ELISA. Durante a execução do teste com o anticorpo, observou-se 3% de reação-cruzada com Dengue (1/35 amostras), 10% com leptospirose (1/10 amostras) e 10% para Fator-reumatóide (1/10).
É necessário o desenvolvimento de testes laboratoriais capazes de diferenciar os casos de arboviroses de forma segura e rápida. Isso será possível, através de exames sensíveis e específicos, que possam ser facilmente performados, e que viabilizem tempo e custo para sua realização.
Damle et al. (2016) desenvolveu um MAC ELISA, utilizando anticorpos contra a proteína C (capsídeo) do CHIKV. Comparando com o MAC ELIZA CHIKV desenvolvido no Instituto Nacional de Virologia de Pune (NIV MAC ELISA) a sensibilidade do teste foi de 87% e 100% de especificidade.
A possibilidade de uma detecção simultânea, de forma sensível e específica seria o ideal para regiões de co-circulação de arboviroses. Wang et al., (2019), desenvolveu um teste imunocromatográfico colorimétrico, capaz de detectar IgM e IgG contra DENV e CHIKV simultaneamente.
CONCLUSÕES
A CHIKF é uma doença debilitante, e desde sua introdução no país, vem gerando preocupações na saúde pública. A eminência de surtos e epidemias em diferentes regiões, chama atenção para certos cuidados no manejo da doença.
A co-circulação de arboviroses, dificulta o diagnóstico baseado nos sintomas, exigindo assim, a confiabilidade dos testes laboratoriais para um diagnóstico correto. Apesar da principal medida de prevenção dessas doenças ser a mesma, ou seja, controle dos vetores, um diagnóstico incorreto dificulta a escolha do tratamento, e o monitoramento do cenário epidemiológico.
Os métodos sorológicos são muito utilizados para o diagnóstico, devido sua praticidade e custo-efetividade, porém, está sujeito a interferentes, como a reação-cruzada. Por meio de estudos, pode-se observar a ocorrência de resultados falsos-positivos, mesmo entre vírus de famílias diferentes.
Para contornar esse problema, é importante a combinação de testes, aumentando a acurácia diagnóstica, por meio da detecção de antígeno e anticorpo, como a associação dos testes moleculares com os sorológicos. Além disso, temos diversos estudos para o desenvolvimento de novos testes, capazes de diferenciar essas doenças, ainda que seja um desafio obter um teste com alta sensibilidade e especificidade.
REFERÊNCIAS
BARROS, Elizabeth Regina de et al. Epidemiologia do vírus mayaro na américa latina entre 2009 a 2019. 2021. Disponível em: http://www.recima21.com.br/index.php/recima21/article/view/327. Acesso em: 05 dez. 2021.
BRASIL. MINISTÉRIO DA SAÚDE. Chykungunya: manejo clínico. 2017. Disponível em: https://bvsms.saude.gov.br/bvs/publicacoes/chikungunya_manejo_clinico.pdf. Acesso em: 11 nov. 2021.
BRASIL. Ministério da Saúde. Secretaria de Vigilância em Saúde. Boletim Epidemiológico: monitoramento dos casos de dengue, febre de chikungunya e febre pelo vírus zika até a semana epidemiológica 52, 2017. 2018. Disponível em: https://antigo.saude.gov.br/images/pdf/2018/janeiro/23/Boletim-2018-001-Dengue.pdf. Acesso em: 06 nov. 2021
DAMLE, R G et al. Diagnostic potential of monoclonal antibodies against the capsid protein of chikungunya virus for detection of recent infection. 2016. Disponível em: https://pubmed.ncbi.nlm.nih.gov/27016930/. Acesso em: 02 dez. 2021.
EISEN, Herman N.. Affinity Enhancement of Antibodies: How Low-Affinity Antibodies Produced Early in Immune Responses Are Followed by High-Affinity Antibodies Later and in Memory B-Cell Responses. 2021. Disponível em: https://cancerimmunolres.aacrjournals.org/content/canimm/2/5/381.full.pdf. Acesso em: 24 out. 2021.
FERREIRA, Andreia Silva et al. Biomarkers of severity and chronification in chikungunya fever: a systematic review and meta-analysis. 2021. Disponível em: https://www.scielo.br/j/rimtsp/a/WqLJ3gCt3gkH5T7Tjxprhps/. Acesso em: 10 out. 2021.
FISCHER, Carlo et al. Challenges towards serologic diagnostics of emerging arboviruses. 2021. Disponível em: https://pubmed.ncbi.nlm.nih.gov/34111589/. Acesso em: 23 out. 2021.
FISCHER, Carlo et al. Robustness of Serologic Investigations for Chikungunya and Mayaro Viruses following Coemergence. 2020. Disponível em: https://pubmed.ncbi.nlm.nih.gov/32024703/. Acesso em: 01 dez. 2021.
FUMAGALLI, Marcilio Jorge. Desenvolvimento de métodos sorológicos para diagnóstico de infecções pelos vírus Chikungunya e Mayaro. 2018. Disponível em: https://teses.usp.br/teses/disponiveis/17/17147/tde-15102018-115825/pt-br.php. Acesso em: 03 dez. 2021.
JAIN, Deepti; SALUNKE, Dinakar M. Antibody specificity and promiscuity. 2019. Disponível em: https://pubmed.ncbi.nlm.nih.gov/30723137/. Acesso em: 08 nov. 2021.
JOHNSON, Barbara W; RUSSELL, Brandy J; GOODMAN, Christin H. Laboratory Diagnosis of Chikungunya Virus Infections and Commercial Sources for Diagnostic Assays. 2016. Disponível em: https://pubmed.ncbi.nlm.nih.gov/27920176/. Acesso em: 10 nov. 2021.
KAM, Yiu-Wing et al. Sero-Prevalence and Cross-Reactivity of Chikungunya Virus Specific Anti-E2EP3 Antibodies in Arbovirus-Infected Patients. 2015. Disponível em: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4287563/. Acesso em: 15 nov. 2021
LIMA, Monique da Rocha Queiroz et al. Analysis of a Routinely Used Commercial Anti-Chikungunya IgM ELISA Reveals Cross-Reactivities with Dengue in Brazil: a new challenge for differential diagnosis?. A New Challenge for Differential Diagnosis?. 2021. Disponível em: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC8147240/pdf/diagnostics-11-00819.pdf. Acesso em: 18 set. 2021.
MALE, David; PEEBLES JUNIOR, R. Stokes; MALE, Victoria. Immunology. 9. ed. Polônia: Elsevier, 2020. Disponível em: https://books.google.com.br/books?id=GIDRDwAAQBAJ&lpg=PP1&hl=pt-BR&pg=PA154&redir_esc=y#v=onepage&q&f=false. Acesso em: 24 out. 2021.
MARQUES, Claudia Diniz Lopes et al. Recomendac¸ões da Sociedade Brasileira de Reumatologia para diagnóstico e tratamento da febre chikungunya. Parte 1 – Diagnóstico e situac¸ões especiais. 2017. Disponível em: https://www.scielo.br/j/rbr/a/8w8g4dCBcTYg5zR4TGvRWhD/?lang=pt&format=pdf. Acesso em: 11 nov. 2021.
MENDOZA, Emelissa J et al. Combining anti-IgM and IgG immunoassays for comprehensive chikungunya virus diagnostic testing. 2019. Disponível em: https://pubmed.ncbi.nlm.nih.gov/31449360/. Acesso em: 30 nov. 2021.
MORCERF, Cely Carolyne Pontes et al. CHIKUNGUNYA: ARBOVIROSE COMO PROBLEMA DE SAÚDE EM EXPANSÃO – UMA REVISÃO BIBLIOGRÁFICA. 2015. Disponível em: http://publicacoes.unigranrio.edu.br/index.php/rcs/article/view/2688. Acesso em: 23 nov. 2021.
NATRAJAN, Muktha s; ROJAS, Alejandra; WAGGONER, Jesse J. Beyond Fever and Pain: Diagnostic Methods for Chikungunya Virus. 2019. Disponível em: https://pubmed.ncbi.nlm.nih.gov/30995993/. Acesso em: 20 set. 2021.
PIANTADOSI, Anne; KANJILAL, Sanjat. Diagnostic Approach for Arboviral Infections in the United States. 2020. Disponível em: https://pubmed.ncbi.nlm.nih.gov/32938736/. Acesso em: 11 nov. 2021.
ROJAS, Manuel et al. Molecular mimicry and autoimmunity. 2018. Disponível em: https://pubmed.ncbi.nlm.nih.gov/30509385/. Acesso em: 14 nov. 2021.
SÃO PAULO. SECRETARIA DE SAÚDE DO ESTADO DE SÃO PAULO. ALERTA 01 SURTO DE CHIKUNGUNYA ESTADO DE SÃO PAULO Sala de Situação Estadual de Enfrentamento às Arboviroses 2021. 2021. Disponível em: https://www.saude.sp.gov.br/resources/cve-centro-de-vigilancia-epidemiologica/areas-de-vigilancia/doencas-de-transmissao-por-vetores-e-zoonoses/doc/chikung/chikung21_alerta01_surto.pdf. Acesso em: 06 nov. 2021.
SUWANMANEE, San et al. Monitoring arbovirus in Thailand: Surveillance of dengue, chikungunya and zika virus, with a focus on coinfections. 2018. Disponível em: https://pubmed.ncbi.nlm.nih.gov/30248317/. Acesso em: 05 dez. 2021.
WANG, Ruisheng et al. Rapid Diagnostic Platform for Colorimetric Differential Detection of Dengue and Chikungunya Viral Infections. 2019. Disponível em: https://pubmed.ncbi.nlm.nih.gov/30896928/. Acesso em: 02 dez. 2021.
ZAID, Ali et al. Arthritogenic alphaviruses: epidemiological and clinical perspective on emerging arboviruses. 2021. Disponível em: https://pubmed.ncbi.nlm.nih.gov/33160445/. Acesso em: 21 out. 2021.
Resumo expandido publicado nos Anais da III Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
Este trabalho foi escrito por:
Nathália Namie Asito1; Luciana da Silva Ruiz Menezes2
1Estudante do Curso de Vigilância Epidemiológica em Saúde Pública – IAL CLR II Bauru-SP; E-mail: [email protected]
2Docente/pesquisador do Núcleo de Ciências Biomédicas – IAL CLR II Bauru-SP.
O Mycobacterium tuberculosis é o principal agente causador da tuberculose, doença milenar, que persiste como um grande problema de saúde pública, acometendo milhões de pessoas anualmente, sendo considerada uma das doenças infecciosas mais letais do mundo. Agravando este cenário, a pandemia de COVID-19 tem tido um impacto significativo no diagnóstico e tratamento da tuberculose, ocasionando uma queda significativa nas notificações e diagnósticos de tuberculose, principalmente nos primeiros meses. O objetivo deste estudo foi avaliar o impacto da COVID-19 na rotina dos exames para diagnóstico de tuberculose no Laboratório de Micobactérias do Instituto Adolfo Lutz CLR II Bauru/SP, bem como realizar levantamento dos casos de coinfecção tuberculose/COVID-19 entre os pacientes com exames enviados aos Laboratórios de Doenças Respiratórias e de Micobactérias do Instituto Adolfo Lutz CLR II Bauru/SP. Os dados obtidos foram coletados no sistema Gerenciador de Ambiente Laboratorial (GAL) para tuberculose e COVID-19. Não foram observadas alterações relevantes em relação ao número de exames e casos diagnosticados de tuberculose quando comparados os anos de 2019, 2020 e 2021. Tal achado pode estar relacionado a população atendida, representada em sua maioria por indivíduos privados de liberdade. No período, de um total de 112 amostras com solicitação de exames para diagnóstico de TB e COVID-19, concomitantemente, foram identificados quatro casos de coinfecção tuberculose/COVID-19, salientando a importância de diagnóstico adequado e rápido para ambas as doenças, tanto para uma coinfecção tuberculose/COVID-19 quanto para o diagnóstico de infecção por tuberculose ou por COVID-19, separadamente.
Palavras–chave: COVID-19. Mycobacterium tuberculosis. SARS-CoV-2. Tuberculose. Coinfecção.
INTRODUÇÃO
A tuberculose (TB) é uma doença infectocontagiosa causada principalmente pelo Mycobacterium tuberculosis stricto senso (BRASIL, 2019). Acomete ambos os sexos e atinge várias faixas etárias. A forma pulmonar é a mais frequente, mas há também, a tuberculose extrapulmonar. A transmissão ocorre principalmente de pessoa para pessoa, por via respiratória em que há a inalação de aerossóis produzidos pela tosse, fala ou espirro de um doente com tuberculose ativa pulmonar ou laríngea (WHO, 2020).
Desde os anos 90, a TB é considerada uma emergência global de saúde pública, acometendo milhões de pessoas anualmente, sendo considerada uma das doenças infecciosas mais letais do mundo, além de ser a principal causa de morte entre pessoas com diagnóstico por HIV (Human Immunodeficiency Virus – Vírus da imunodeficiência humana) (BRASIL, 2021; BERRA, et al.; 2021; WHO, 2020).
É uma infecção que pode ser prevenida e curada, no entanto, ainda permanece como um grande problema de saúde pública, acometendo prioritariamente os países de baixa e média renda, e responsável pela morte de aproximadamente 1,5 milhão de pessoas todos os anos (BRASIL, 2019). Está associada a pessoas em situação de pobreza, dificuldades econômicas, em vulnerabilidade e marginalização (WHO, 2020). A coinfecção com HIV e o surgimento de isolados resistentes às drogas (tratamento inadequado e uso indiscriminado de antibióticos) tornam a situação ainda mais alarmantes (WHO, 2015). Além disso, a TB é uma doença negligenciada, sem muito investimento da indústria e do governo na descoberta de novos fármacos ou novos métodos diagnósticos, e diante do advento da pandemia, este cenário foi ainda mais agravado, o que futuramente poderá ocasionar graves consequências.
No Brasil, preconizado pelo Ministério da Saúde, o diagnóstico de TB é feito a partir da baciloscopia e cultura de escarro (TB pulmonar) ou de outros espécimes clínicos (TB extrapulmonar). A cultura é considerada a técnica padrão ouro para identificação da doença (BERRA et al., 2021). A OMS, em 2010, aprovou o uso do sistema GeneXpert MTB/RIF para realizar testes moleculares rápidos para TB, com resultados liberados em menos de 2 horas (LIMA, et al., 2017).
A chegada da pandemia de COVID-19 ameaça a sustentabilidade das ações de controle da TB bem como a manutenção dos avanços já obtidos (BRASIL, 2021). Tal cenário apresenta um impacto significativo no diagnóstico e tratamento da TB, em que se observa a piora dos indicadores laboratoriais, de tratamento e busca de novos casos da doença, que poderá refletir nas futuras taxas de incidência e mortalidade desta doença (MIGLIORI, et al., 2020). Essa redução poderá sobrecarregar a rede de atenção à saúde para suprir as demandas de COVID-19, a baixa procura da população aos serviços de saúde e erros diagnósticos pela similaridade de alguns sintomas entre TB e COVID-19, acarretando implicações significativas no diagnóstico laboratorial e tratamento da infecção.
Além disso, desde o início da pandemia, têm sido notificados casos de tuberculose e COVID-19 concomitantes (TADOLINI, et al., 2020) A associação apresenta grande potencial de morbidade e mortalidade. Pacientes que apresentam coinfecção tuberculose/COVID-19 apresentam maiores riscos de desfechos negativos e óbito do que pacientes que apresentam somente a infecção por COVID-19. Devido a isso, a detecção prévia de uma infecção conjunta, é muito necessária, para que sejam realizados seus manejos adequados, avaliando as individualidades de ambas as doenças (SILVA, et al., 2021).
O objetivo deste estudo foi avaliar o impacto da COVID-19 na rotina de exames para tuberculose no Laboratório de Micobactérias do Instituto Adolfo Lutz CLR II Bauru/SP. Foram pesquisados e comparados, o número de exames de baciloscopia, cultura e teste rápido molecular para TB, o número de casos diagnosticados e realizado um levantamento dos casos de coinfecção tuberculose/COVID-19, no período de 2019 a 2021.
MATERIAL E MÉTODOS
Trata-se de um estudo descritivo e quantitativo baseado na coleta de dados. Os dados obtidos no estudo foram provenientes de demanda de exames para diagnóstico de TB do Laboratório de Micobactérias do Núcleo de Ciências Biomédicas do Instituto Adolfo Lutz CLR II Bauru/SP, e coletados por meio de pesquisa no sistema Gerenciador de Ambiente Laboratorial (GAL) para TB e COVID-19. Foram pesquisados dados referentes ao quantitativo de exames de baciloscopia, cultura e gene Xpert realizados, além, do número de casos diagnosticados com entrada para diagnóstico da tuberculose. Os dados coletados foram referentes ao período de janeiro a outubro de 2019 a 2021, e a presença da coinfecção tuberculose/COVID-19, no período de abril de 2020 a novembro de 2021.
RESULTADOS E DISCUSSÃO
Em primeiro de abril do ano de 2020, a COVID-19 superou a TB e se tornou a doença infecciosa que mais mata a cada dia (SAUNDERS e EVANS, 2020). Durante a pandemia muitas ações foram implementadas em resposta à COVID-19, principalmente o reaproveitamento de equipamentos de diagnóstico, profissionais e instalações de saúde, orçamentos e lacunas na comunicação de dados, impactando assim, nos programas de monitoramento da TB (SARINOGLU, et al., 2020).
Jain et al. (2020), identificaram no início da pandemia de COVID-19, consideráveis interrupções nos serviços de saúde relacionados à TB, tanto na atenção primária quanto em hospitais. Outro estudo que incluiu 37 centros de TB do mundo inteiro, comparou o primeiro trimestre de 2019 com o de 2020 e concluiu que houve redução do número de novos casos de tuberculose, e também, do número de consultas ambulatoriais (MIGLIORI, et al., 2020).
Na tabela 01, é observado o total de exames realizados (TRM, baciloscopia e cultura) para diagnóstico de TB nos meses de janeiro a outubro nos anos de 2019, 2020 e 2021 no IAL CLR II Bauru. De janeiro a outubro de 2020 houve um aumento de 10,32% nas solicitações de exames para diagnóstico de TB quando comparado a 2019. E de janeiro a outubro de 2021 houve um aumento de 17,6% e 9% em relação a janeiro a outubro de 2019 e 2020, respectivamente.

O ano de 2020 (janeiro a outubro) apresentou um aumento de 9,83% no número de baciloscopias realizadas em relação ao mesmo período de 2019. Em 2021, entre os meses de janeiro a outubro, foi observada uma redução de 22% em relação as baciloscopias realizados nos mesmos meses de 2020 (Figura 01).
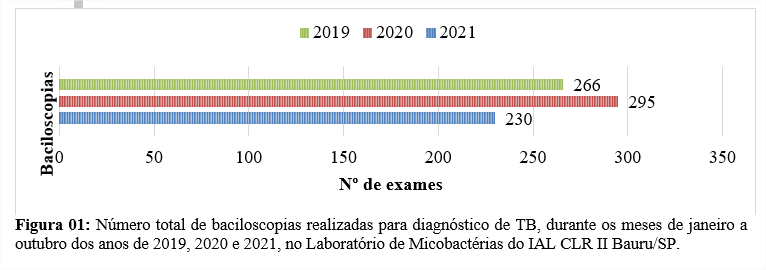
Em relação à baciloscopia, os casos controles foram excluídos, assim, o número total de baciloscopias no ano de 2020, são para diagnóstico de casos novos. Neste ano, foi observado um número maior de amostras de escarro sendo enviadas com volume abaixo do recomendado. Possivelmente este exame não mostrou uma queda em função do baixo acesso ao serviço, mas talvez, por questões relacionadas a quantidade de material coletado, o que justificaria também, a diminuição de TRM no ano de 2020, que durante os meses avaliados no estudo apresentou um quantitativo de exames de 5% e 4,7% menor que os anos de 2019 e 2021, respectivamente (Figura 02). Amostras com volume abaixo do recomendado não são recomendadas para TRM, sendo assim a baciloscopia é realizada.
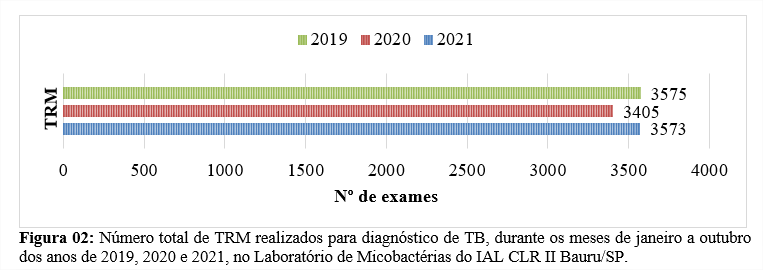
O total de exames de cultura para diagnóstico de TB, de janeiro a outubro de 2020, foi 26,9% maior comparado a 2019, e 13,9% menor em relação a 2021 (Figura 03).
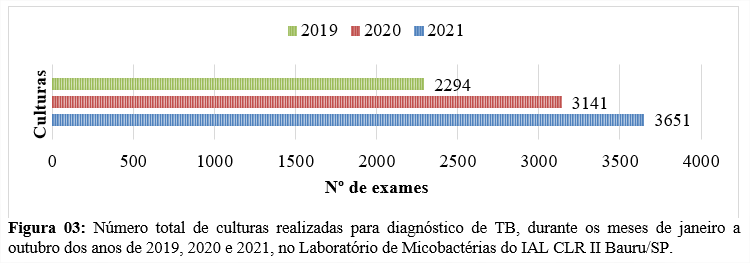
Em relação ao quantitativo de casos diagnosticados por meio da cultura (padrão ouro), os anos de 2020 e 2021 apresentaram 7,3% menos casos positivos comparados ao ano de 2019. Um número maior de casos negativos foi observado nos anos de 2020 (29%) e 2021 (40%), em relação ao ano de 2019 (Tabela 02).

Diferentemente dos estudos relatados, quando comparamos 2019 e 2020, neste estudo, não foi observada diminuição de diagnóstico de novos casos de TB no Laboratório de Micobactérias do IAL CLR II Bauru/SP. No ano de 2021 foi observado um aumento de 9% no diagnóstico de TB em relação ao ano de início da pandemia (2020). Talvez, pelo fato de que a maioria da população atendida, é composta por indivíduos privados de liberdade, provenientes de 13 diferentes unidades prisionais do Estado de São Paulo, não tenha sido observado queda no diagnóstico de casos novos entre o ano de 2019 e os anos de surgimento e estabelecimento da pandemia, já que é uma população confinada, suas condições são conhecidas e existe um vínculo estabelecido entre o Sistema Penitenciário e o Serviço de Saúde, além de uma logística determinada para exame de escarro.
Em 2020, He et al., demostraram a ocorrência de três casos de coinfecção tuberculose/COVID-19, dentre os 139 casos estudados. Dois pacientes já haviam tido TB pulmonar e recebido terapia, enquanto um paciente apresentava TB não tratado por 50 anos. Dois dos pacientes eram mais idosos e assim, um desenvolveu um tipo crítico e o outro um tipo grave de COVID-19. Os três pacientes apresentaram uma longa recuperação antes de receberem alta hospitalar. Neste estudo, para um total de 112 pacientes com exames para diagnóstico de TB e COVID-19, concomitantemente, foi observada a coinfecção tuberculose/COVID-19 em quatro pacientes (Figura 04).

He et al. (2020) e Jain et al. (2020), observaram que doenças pulmonares prévias (TB tratada ou não) e a idade avançada do paciente, são fatores de risco para um pior prognóstico de pessoas infectadas com COVID-19, podendo apresentar danos pulmonares prévios, agravando com a infecção por SARS-CoV-2. Algumas características comuns dos pacientes: comorbidades crônicas, hipertensão crônica, doenças crônicas cardíacas, diabetes, pacientes imunossuprimidos e doença crônica longa (SARINOGLU, et al., 2020). Jain et al. (2020), sugeriram que pacientes com TB latente e doença estabelecida apresentam risco aumentado de infecção por SARS-CoV-2 e maior predisposição aos sintomas da COVID-19.
A pandemia de COVID-19, teve impacto significativo na execução de vários programas de prevenção, tratamento e vigilância da tuberculose. As novas diretrizes de saúde implementadas com a chegada da pandemia, fizeram com que outras áreas da saúde ficassem sem a devida atenção, resultando em desafios no tratamento tradicional da tuberculose e exigiram a reorganização da metodologia para dar suporte aos pacientes, incluindo até, a utilização das consultas remotas (JAIN et al., 2020). A importância do diagnóstico rápido e adequado em ambas as doenças é um fator crucial, pois o isolamento do paciente infectado é a medida preventiva mais importante para que seja limitada a transmissão das duas doenças (HE, et al., 2020; SARINOGLU, et al., 2020). A prevenção é o caminho, reaproveitando as metodologias disponíveis para combater novas pandemias, e também, garantir a continuidade no monitoramento e controle das pessoas que buscam tratamento para doenças previamente existentes (CHOPRA, ARORA e SINGH, 2020).
CONCLUSÕES
Com este estudo pode-se observar que no laboratório de Micobactérias do CLR II Bauru/SP, não foram verificadas alterações relevantes que confirmem as informações encontradas nos demais estudos citados, em relação ao diagnóstico de TB no período da pandemia por COVID-19, o que pode estar relacionado com a população atendida pelo serviço do laboratório de Micobactérias do CLR II Bauru/SP, representada em sua maioria por indivíduos privados de liberdade.A identificação de casos de coinfecção tuberculose/COVID-19 salienta a necessidade de uma identificação diagnóstica adequada e rápida tanto para uma coinfecção tuberculose/COVID-19 quanto para o diagnóstico de infecção por tuberculose ou por COVID-19, separadamente, para que sejam realizados o tratamento e controle adequados de ambas as doenças, principalmente por serem doenças que se disseminam rapidamente na população e juntas podem ocasionar a evolução para quadros mais graves.
REFERÊNCIAS
BERRA, T. Z.; et al. Effectiveness and Trend Forecasting of Tuberculosis Diagnosis After Introduction of GeneXpert in a City in South-eastern Brazil. PLoS One, v. 16, n. 5, 2021.
BRASIL. Ministério da Saúde. Secretaria de Vigilância em Saúde – Departamento de Vigilância das Doenças Transmissíveis. Manual de Recomendações Para o Controle da Tuberculose no Brasil. Brasília – DF, 2019.
BRASIL, Ministério da Saúde. Secretaria de Vigilância em Saúde. Indicadores Operacionais da Tuberculose no Brasil e a COVID-19: análise comparativa dos anos de 2019 e 2020. Boletim Epidemiológico, v. 52, n. 22, 2021.
CHOPRA, K. K.; ARORA, V. K.; SINGH, S. COVID 19 and tuberculosis. Indian J Tuberc, v. 67, n. 2, p. 149-151, 2020.
HE, G.; et al. COVID-19 in Tuberculosis patients: a report of three cases. J Med Virol, 2020.
JAIN, V. K.; et al. Tuberculosis in the era of COVID-19 in India. Diabetes Metab Syndr, v. 15, n. 5, p. 1439-1443, 2020.
LIMA, T. M.; et al. GeneXpert MTB/RIF assay for diagnosis of tuberculosis. Rev Pan-Amaz Saude, v. 8, n. 2, p. 67–78, 2017.
MIGLIORI, G. B.; et al. Worldwide Effects of Coronavirus Disease Pandemic on Tuberculosis Services, January-April 2020. Emerg Infect Dis, v. 26, n. 11, p. 2709-2712, 2020.
SARINOGLU, R. C.; et al. Tuberculosis and COVID-19: Na overlapping situation during pandemic. J Infect Dev Ctries, v. 14, n. 7, p. 721-725, 2020.
SAUNDERS, M. J.; EVANS, C. A. COVID-19, tuberculosis and poverty: preventing a perfect storm. Eur Respir J, v. 56, n. 1, p. 1-5, 2020.
SILVA, D. R.; et al. Tuberculose e COVID-19, o Novo Dueto Maldito: quais as diferenças entre Brasil e Europa. J Bras Pneumol, v. 47, n. 2, 2021.
TADOLINI, M.; et al. Active tuberculosis, sequelae and COVID-19 coinfection: first cohort of 49 cases. Eur Respir J, v. 56, n. 1, p. 1-5, 2020.
WHO, World Health Organization. Global Tuberculosis Report: Global Tuberculosis Control. Geneva. 2015.
WHO, World Health Organization. Global Tuberculosis Report. Geneva, 2020.
Resumo expandido publicado nos Anais da III Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
Este trabalho foi escrito por:
Leonora Feitosa De Carvalho1; Juliana Maira Watanabe Pinhata2
1Estudante do Curso de Vigilância Laboratorial em Saúde Pública – CB – IAL; E-mail: [email protected]
2Pesquisadora Científicado Núcleo de Tuberculose e Micobacterioses – CB – IAL.
RESUMO
O Núcleo de Tuberculose e Micobacterioses (NTM) é o laboratório de referência estadual para tuberculose, e recebe cepas de micobactérias enviadas pelos laboratórios públicos do estado de São Paulo. No NTM, as cepas sugestivas de Mycobacterium tuberculosis com critérios para teste de sensibilidade são submetidas ao teste GenoType MTBDRplus (fita Hain). As cepas sugestivas de micobactérias não tuberculosas (MNTs) são enviadas para identificação da espécie. Este trabalho avaliou as cepas identificadas presuntivamente como M. tuberculosis encaminhadas para a fita Hain, mas que tiveram resultado negativo para M. tuberculosis, caracterizando-as quanto aos seus aspectos macroscópicos e ao resultado de identificação. Entre fevereiro/2019 a agosto/2021, 13.144 cepas foram encaminhadas para a fita Hain. Destas, 424 (3,2%) foram negativas para M. tuberculosis, sendo que 377 (88,9%) foram encaminhadas para identificação de espécie. Das culturas encaminhadas para identificação, 235 (62,3%) possuíam características de crescimento de M. tuberculosis, 113 (30%) apresentavam características de contaminação, 27 (7,2%) tinham pouco crescimento e 2 (0,5%) foram identificadas visualmente como mistura de M. tuberculosis+MNT (0,5%). As espécies de MNT mais prevalentes foram M. fortuitum (n=71, 18,8%), M. abscessus (n=28, 7,4%),complexo M. avium (n=26, 6,9%),complexo M. kansasii (n=21, 5,6%) e M. intracellulare/M. chimaera (n=20, 5,3%). A análise presuntiva das culturas e a posterior identificação da espécie de micobactéria são muito importantes, pois permitem a diferenciação entre M. tuberculosis e MNTs. Algumas espécies de MNTs podem apresentar floculação com ou sem turbidez em meio líquido, características comuns a M. tuberculosis, confudindo o observador no momento da triagem das culturas.
Palavras-chave: complexo Mycobacterium tuberculosis; fita Hain; GenoType MTBDRplus; micobactérias não tuberculosas
INTRODUÇÃO
A tuberculose (TB) é uma doença contagiosa e crônica que atinge principalmente os pulmões. A forma clássica de transmissão da TB pulmonar acontece por via respiratória, de pessoa a pessoa, quando o doente com a forma ativa da doença tosse, fala ou espirra, eliminando os bacilos (DUCATI et al., 2015).
O principal agente etiológico da TB é o Mycobacterium tuberculosis. Outras espécies de micobactérias que causam TB são M. bovis (M. bovis subsp. bovis e M. bovis BCG), M. microti, M. africanum, M. caprae, M. pinnipedi, M. canettii, M. suricattae, M. dassie, M. orygis e M. mungi. Essas espécies de micobactérias, juntamente com o M. tuberculosis, são estritamente patogênicas e fazem parte do complexo M. tuberculosis, infectando tanto o homem como alguns animais. O M. leprae, agente etiológico da hanseníase, é a outra espécie estritamente patogênica dentro do gênero Mycobacterium (COSCOLLA E GAGNEUX, 2014; TORTORA et al., 2017).
As micobactérias não tuberculosas (MNTs) são espécies de micobactérias encontradas amplamente distribuídas no ambiente, sendo consideradas patógenos oportunistas principalmente em imunodeprimidos, causando infecções que são conhecidas como micobacterioses (BRASIL, 2021).
O Núcleo de Tuberculose e Micobacterioses (NTM) do Instituto Adolfo Lutz é o laboratório de referência estadual para TB, e recebe culturas de micobactérias enviadas pelos laboratórios públicos do estado de São Paulo. No NTM são analisadas as culturas em meio líquido e sólido e dependendo de suas características macroscópicas, estas são identificadas presuntivamente como M. tuberculosis ou MNT. As cepas sugestivas de M. tuberculosis cujos pacientes possuam critérios para o teste de sensibilidade seguem para a identificação e detecção de mutações associadas à resistência aos fármacos de 1ª linha pelo kit GenoType MTBDRplus (Hain Lifescience, Alemanha), aqui denominado fita Hain, implantado na rotina do NTM em fevereiro de 2019. As culturas sugestivas de MNT são enviadas para identificação da espécie.
O objetivo deste trabalho é descrever as características das cepas identificadas presuntivamente como M. tuberculosis que apresentaram resultado negativo no teste de fita Hain para o complexo M. tuberculosis. Pretende-se, assim, avaliar os fatores e as características dessas culturas que interferem diretamente nos erros do processo, buscando corrigir e melhorar a identificação presuntiva das espécies de micobactérias e seu direcionamento para os testes, garantindo um resultado eficaz no método de triagem e economia de tempo e insumos no laboratório, além de diminuir as perdas e atrasos nos exames.
MATERIAL E MÉTODOS
O NTM recebe culturas de micobactérias isoladas em meio líquido ou meio sólido dos laboratórios públicos do estado de São Paulo, para que seja realizada a identificação de espécie e o teste de sensibilidade pelo método de fita Hain (HAIN LIFESCIENCE, 2015). A triagem para este teste é feita através da análise das características de crescimento das culturas para que estas possam ser identificadas presuntivamente como sendo do complexo M. tuberculosis ou MNTs.
As bactérias do complexo M. tuberculosis isoladas em meio sólido apresentam colônias de cor creme, rugosas e de aspecto seco. O crescimento em meio líquido deve estar límpido e com presença de floculação (PINHATA et al., 2018). Uma vez identificadas presuntivamente como sendo do complexo M. tuberculosis, as culturas são enviadas para o teste de fita Hain.
Como a fita Hain é um teste molecular e detecta apenas sequências específicas de DNA do M. tuberculosis, as culturas visualmente contaminadas e com pouco crescimento, mas com aspecto das colônias do complexo M. tuberculosis, são também enviadas para o teste.
As colônias das MNT em meio sólido apresentam-se de cor creme ou pigmentada, lisas, úmidas ou rugosas. Em meio líquido, elas apresentam crescimento turvo e/ou pigmentado, mas também podem estar floculadas. Culturas com essas características são enviadas para a identificação molecular da espécie pela técnica Speed-Oligo Mycobacteria (Vircell, Espanha). As MNTs que não puderem ser identificadas por esta técnica são encaminhadas para a metodologia in house de PRA-hsp65, onde uma PCR é realizada para a amplificação de uma sequência de 440 pb do gene hsp65, presente em todas as espécies de micobactérias (CHIMARA et al., 2008).
Para a análise dos dados, criou-se uma planilha no programa Excel para onde foram repassadas todas as informações constantes nas listas de realização do teste de fita Hain, contendo os seguintes dados das cepas encaminhadas para o teste: tipo de meio de cultura onde foram isoladas, número da amostra, número da cepa, resultados do teste e possíveis observações.
Os resultados tanto dos testes de fita Hain como da identificação da espécie de micobactéria liberados no sistema GAL (Gerenciamento de Ambiente Laboratorial) foram obtidos por meio da geração de relatórios epidemiológicos, em formato compatível com o Excel. Estes dados foram agrupados aos dados levantados a partir das listas do teste de fita Hain.
A partir do banco de dados criado, foram avaliados: (i) o número e frequência de cepas com resultado negativo para o complexo M. tuberculosis no período de estudo, (ii) os anos de recebimento das cepas, (iii) o tipo de meio de cultura (sólido ou líquido) no qual a cepa foi isolada, (iv) as características macroscópicas das culturas e (v) o número e frequência das espécies de MNT identificadas.
RESULTADOS E DISCUSSÃO
No período de fevereiro de 2019 a agosto de 2021, 13.144 cepas foram submetidas ao teste de fita Hain, sendo que 424 (3,2%) tiveram resultado negativo para o complexo M. tuberculosis. Esta baixa taxa de isolados negativos para o complexo M. tuberculosis encaminhados para a fita Hain demonstra que a triagem das culturas através da observação visual é bastante acurada.
A Tabela 1 mostra os critérios avaliados referentes às 424 cepas negativas no teste de fita Hain. As maiores frequências de cepas negativas no teste foram observadas em 2019 (n=172, 40,6%) e em 2020 (n=183, 43,2%). Dentre as 424 cepas, 345 (81,4%) estavam semeadas em meio de MGIT e 75 (17,7%) em meio sólido (Tabela 1).
Foram enviadas para identificação de espécies 377 (88,9%) cepas, sendo que 113 (30,0%) estavam visualmente contaminadas, 235 (62,3%) estavam visualmente puras e com características de M. tuberculosis, 27 (7,2%) tinham pouco crescimento e 2 (0,5%) foram identificadas visualmente como mistura de M. tuberculosis + MNT (0,5%) (Tabela 1).
Das 47 (11,1%) cepas que tiveram o resultado liberado direto da fita Hain, ou seja, não foram encaminhadas para identificação, 2 (4,3%) foram liberadas como culturas contaminadas, 5 (10,6%) como complexo M. tuberculosis após a realização do teste imunocromatográfico TBAg MPT64 e 40 (85,1%) como não houve crescimento de BAAR (bacilos álcool-ácido resistentes), o que significa que havia pouco ou nenhum crescimento na cultura, impedindo a realização da identificação (Tabela 1).
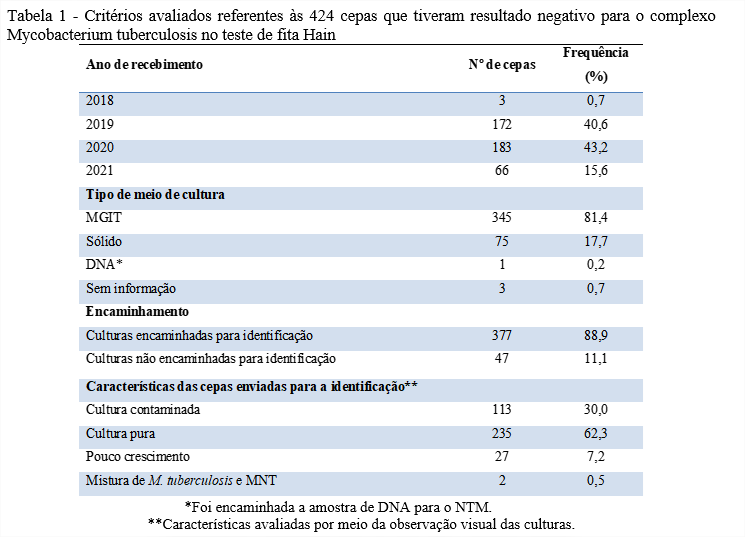
A Tabela 2 mostra as espécies de micobactérias identificadas a partir das culturas visualmente puras e das culturas visualmente contaminadas. Dentre as 113 (30,0%) culturas que estavam visualmente contaminadas, mas foram encaminhadas para a fita Hain por possuírem também características visuais de M. tuberculosis, as espécies mais frequentes isoladas foram M. fortuitum (n=29, 25,7%), M. intracellulare/M. chimaera (n=9, 8,0%), complexo M. avium (n=8, 7,1%), M. peregrinum (n=7, 6,2%), M. abscessus (n=6, 5,3%) e M. gordonae (n=6, 5,3%). Em 16 culturas (14,2%) foi liberado o resultado de não houve crescimento de BAAR. Pinhata e col. (2018) observaram que espécies de MNT como M. intracellulare/M. chimaera, M. nebraskense e micobactérias de crescimento rápido podem apresentar crescimento floculado e turvo em meio líquido de MGIT, levando o observador a interpretar essas características como sendo de M. tuberculosis com contaminação.
Entre as culturas puras, ou seja, com características de M. tuberculosis, as espécies mais frequentes isoladas foram M. fortuitum (n=41, 17,4%), M. abscessus (n=21, 8,9%), M. kansasii (n=15, 6,3%), M. avium (n=14, 6,0%) e M. peregrinum (n=12, 5,1%). Em 28 culturas (11,9%) foi liberado o resultado de Não houve crescimento de BAAR (Tabela 2). Pinhata e col. (2018) observaram crescimento característico de M. tuberculosis (floculado em MGIT) em espécies como M. fortuitum, M. intracellulare/M. chimaera, M. gordonae, M. avium, M. chelonae e M. abscessus, semelhante ao encontrado neste trabalho.
Tabela 2 – Número e freqüência das espécies identificadas a partir das culturas contaminadas e puras após análise visual e que tiveram resultado negativo para o complexo Mycobacterium tuberculosis no teste de fita Hain
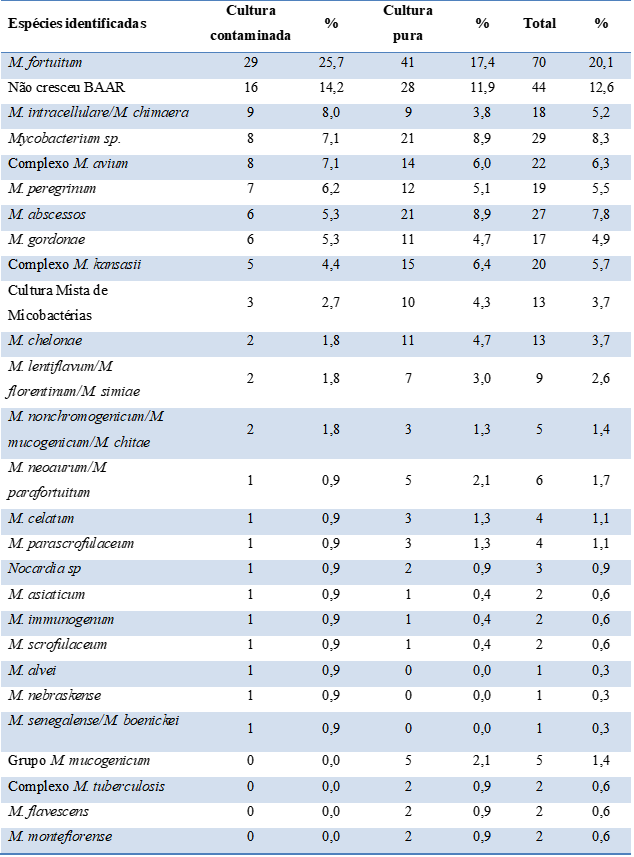
Entre as 27 culturas com pouco crescimento negativas para o complexo M. tuberculosis na fita Hain, 9 não puderam ser identificadas devido ao pouco crescimento e 2 foram identificadas apenas como sendo do gênero Mycobacterium. O restante das espécies identificadas foram: complexo M. avium (n=4), M.lentiflavum/M. florentinum/M. simiae (n=3), M. gordonae (n=2), M. intracellulare/M.chimaera (n=2), complexo M. kansasii, M. abscessus, M. fortuitum, M.lentiflavum/M. florentinum/M. simiae e M. neoaurum/M. parafortuitum, com 1 isolado cada.
Das 2 culturas identificadas visualmente como sendo mistura de M. tuberculosis + MNT e negativas na fita Hain, 1 foi identificada como sendo mistura de micobactérias (cultura mista) e a outra como M. lentiflavum/M. florentinum/M. simiae.
De acordo com o Manual de Recomendações para o diagnóstico e tratamento das doenças causadas por micobactérias não tuberculosas no Brasil (BRASIL, 2021), entre 2013 a 2019, as espécies mais prevalentes causadoras de micobacterioses em São Paulo foram as do complexo M. avium, complexo M. abscessus, complexo M. fortuitum e M. kansasii, corroborando os resultados do nosso estudo.
CONCLUSÕES
Podemos concluir que a análise presuntiva das culturas e a posterior identificação da espécie de micobactéria são muito importantes na rotina do laboratório, pois permitem a diferenciação entre o complexo M. tuberculosis e as MNTs. Como os sinais e sintomas da TB pulmonar são semelhantes aos das micobacterioses pulmonares, identificar a espécie causadora de doença é primordial para se indicar o tratamento correto.
Algumas espécies de MNTs apresentam floculação com ou sem turbidez em meio líquido de MGIT, aspectos que confundem o observador no momento da triagem das culturas, pois são características comuns ao complexo M. tuberculosis, causando erros no direcionamento das culturas. Portanto, otreinamento dos técnicos que realizam a triagem macroscópica e microscópica das culturas é de extrema importânciapara o laboratório, visto que um direcionamento incorreto das culturas gera gastos, acúmulo de trabalho e atraso no laudo do paciente.
AGRADECIMENTOS
A Aparecida A. Pereira pelo recepcionamento das amostras no laboratório; a Andreia R. Souza, Erica Chimara, Fernanda C. S. Simeão, Juliana F. Gallo e Romilda A. Lemes pela realização da identificação da espécie; a Fabiane M. A. Ferreira, Sonia M. Costa, Vera L. M. Silva e Rosângela S. Oliveira pela realização dos testes de fita Hain; a Flavia F. Mendes e Gabriela M. Moraes pela contribuição no levantamento dos dados; e a Rosângela S. Oliveira, Angela P. Brandão e Lucilaine Ferrazoli pelas contribuições científicas.
REFERÊNCIAS
1. BRASIL. Ministério da Saúde. Recomendações para o diagnóstico e tratamento das doenças causadas por micobactérias não tuberculosas no Brasil. P95. Brasília: 2021.
2. CHIMARA, E. et al. Reliable identification of mycobacterial species by PCR-restriction enzyme analysis (PRA)-hsp65 in a reference laboratory and elaboration of a sequence-based extended algorithm of PRA-hsp65 patterns. BMC Microbiol. 2008; 8, 48.
3. COSCOLLA, M.; GAGNEUX, S. Consequences of genomic diversity in Mycobacterium tuberculosis. Semin Immunol. 2014; 26:431–44.
4. DUCATI, R. G. et al. Micobactérias. Microbiologia Médica. 6ed. São Paulo: Editora, Atheneu: 2015.
5. HAIN LIFESCIENCE. GenoType MTBDRplus. Hain Lifescience. Versão 2.0; 06/2015. Disponível em:
https://www.hainlifescience.de/include_datei/kundenmodule/packungsbeilage/download.php?id=936. Acesso em: 18 out. 2021.
6. PINHATA, J. M. W. et al. Growth characteristics of liquid cultures increase the reliability of presumptive identification of Mycobacterium tuberculosis complex. J Med Microbiol. 2018; 67(6):828-33.
7. TORTORA, G. J.; FUNKE, B.R.; CASE, C. Microbiologia. 12ª ed. Porto Alegre: Atheneu, 2017.
8. VIRCELL. Instrução de uso do produto. Disponível em: https://vircell.com.br. Acesso em: 20 set. 2021.
Resumo expandido publicado nos Anais da III Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
Este trabalho foi escrito por:
Juliana Yi Dan Wu1; Márcia Lombardo2
1Estudante do Curso de Especialização em Vigilância Laboratorial em Saúde Pública/Unidade didática Instituto Adolfo Lutz – CEFOR/SUS – IAL. E-mail: [email protected]
2Docente/pesquisador do Núcleo de Ensaios Físicos e Químicos em Medicamentos/Instituto Adolfo Lutz – NFQM – IAL. E-mail: [email protected]
Resumo: Nitrosaminas (NAs) são compostos orgânicos caracterizados pela ligação de um grupo nitroso a um grupo amina e potencial carcinogênico em exposições por longos períodos. Recentemente, a presença de NAs foi identificada em medicamentos e insumos farmacêuticos ativos, acarretando no recolhimento de produtos disponíveis no mercado. O objetivo deste trabalho foi elaborar uma revisão narrativa sobre NAs em medicamentos. O levantamento bibliográfico foi realizado em sítios eletrônicos de órgãos nacionais e internacionais de saúde e nas bases de dados Google acadêmico e Pubmed. Verificou-se que a causa mais discutida para a ocorrência de NAs em medicamentos é o emprego de agentes nitrosantes durante a síntese de fármacos e a principal estratégia de controle consiste no gerenciamento de risco quanto à formação e toxicidade. As análises laboratoriais para identificação e quantificação das NAs incluem técnicas de cromatografia gasosa ou líquida acoplada à espectroscopia de massas. Na área farmacêutica, o controle de NAs é um tema que ainda se encontra em ampla discussão quanto aos impactos na saúde, limites de aceitação, métodos de eliminação, detecção e monitoramento.
Palavras–chave: impurezas em medicamentos; insumos farmacêuticos; nitrosaminas; vigilância sanitária
INTRODUÇÃO
As nitrosaminas (NAs) são compostos orgânicos frequentemente encontrados como contaminantes de produtos para uso humano e que podem apresentar carcinogenicidade em exposições crônicas. O termo N-nitrosamina é usado para caracterizar um amplo grupo de compostos N-nitrosos alifáticos ou aromáticos que possuem o grupo funcional nitroso ligado a uma amina, como mostra a Figura 1 (ANVISA, 2021a).
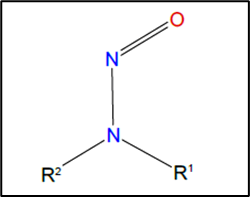
Fonte: ANVISA (2021b)
De maneira geral, as NAs são formadas a partir da nitrosação de aminas, principalmente aminas secundárias, com agentes nitrosantes (Figura 2), e tal reação ocorre em pH ácido (RATH; CANAES, 2009).
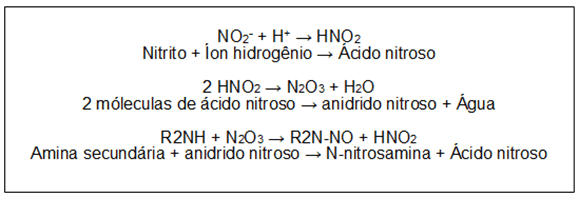
Fonte: Adaptado de Rath e Canaes (2009)
Primeiramente identificadas como importantes poluentes ambientais, as NAs são frequentemente encontradas em águas cloradas e cloraminadas, como também em alimentos curados ou fertilizados, produtos derivados do tabaco e produtos cosméticos e de higiene pessoal. Recentemente, estas impurezas foram encontradas em diversos produtos farmacêuticos comercializados (PASCHOAL, 2015; GUSHGARI, HALDEN, 2018; THRESHER et al., 2020).
A descoberta de NAs em medicamentos iniciou-se em meados de 2018, quando a Agência Europeia de Medicamentos (EMA) informou a presença de dimetilnitrosamina (NDMA) no insumo farmacêutico ativo (IFA) de valsartana (anti-hipertensivo) fornecido por uma empresa chinesa. Posteriormente, diferentes NAs foram detectadas em vários fármacos (EMA, 2019; ANVISA, 2021a).
Diante disso, as agências reguladoras e indústrias farmacêuticas iniciaram investigações sobre a ocorrência de NAs em medicamentos e insumos farmacêuticos, e priorizaram o desenvolvimento de métodos para controle e monitoramento destas impurezas (ANVISA, 2021a; FDA, 2021a).
Considerando os impactos negativos das NAs para a saúde da população e que ainda existem poucos estudos acerca de NAs na área farmacêutica, o objetivo deste trabalho foi elaborar uma revisão bibliográfica sobre o tema, abordando aspectos regulatórios, analíticos e toxicológicos de impurezas do tipo NA em medicamentos e IFAs.
MATERIAL E MÉTODOS
Uma revisão bibliográfica narrativa foi realizada por meio de buscas on-line, divididas em três etapas: (i) consulta de endereços eletrônicos oficiais da Agência Nacional de Vigilância Sanitária (ANVISA), European Medicines Agency (EMA) e Food and Drug Administration (FDA), para a seleção de notícias, informes técnicos, guias e normas relacionadas com NAs em medicamentos e IFAs; (ii) busca ampla empregando-se a ferramenta de pesquisa Google Acadêmico e a palavra-chave nitrosaminas, para a seleção de publicações relacionadas com NAs em produtos para uso humano; (iii) busca direcionada empregando-se a ferramenta de pesquisa Pubmed e os descritores nitrosamines pharmaceutical products, nitrosamines valsartane, nitrosamines ranitidine e nitrosamines metformine, para a seleção de artigos científicos com abordagem direta do tema, publicados nos últimos cinco anos.
RESULTADOS E DISCUSSÃO
Neste estudo, foi possível verificar que existem muitas causas para a ocorrência de NAs em medicamentos, como: uso de agentes nitrosantes durante a síntese de fármacos, sendo o nitrito de sódio o mais utilizado; uso de matérias-primas, materiais recuperados ou reciclados (solventes, reagentes e catalisadores) contaminados com NAs; fornecedores que utilizam matérias-primas ou processos contendo agentes nitrosantes ou NAs residuais; degradação de matérias-primas durante a fabricação ou armazenamento do produto, como é o caso dos solventes dimetilformamida e metilpirrolidona, que podem sofrer hidrólise ácida ou básica, formando um substrato vulnerável à nitrosação; transferência de NAs durante o processo de fabricação; embalagens contendo película de nitrocelulose, que pode reagir com aminas de baixo peso molecular durante a selagem por calor; presença de grupos funcionais nitrosáveis em IFAs ou suas impurezas, que podem reagir durante a fabricação ou armazenamento do produto (ANVISA, 2021a; EMA, 2021a).
Para garantir a segurança do paciente, as agências reguladoras do Brasil e do mundo determinaram o recolhimento voluntário e a suspensão de manipulação, comercialização, distribuição e uso de inúmeros lotes de medicamentos e/ou IFAs que continham impurezas do tipo NA (Tabela 1). Atualmente, a produção de IFAs e produtos acabados de cloridrato de ranitidina estão proibidos, visto que durante o armazenamento ocorre a degradação espontânea da molécula e a formação de NDMA, não havendo ainda uma possibilidade de estabilização deste fármaco (ANVISA, 2020; EMA, 2021b).

Como os compostos N-nitrosos geralmente apresentam elevada atividade mutagênica, os limites aceitáveis de ingestão diária devem ser determinados caso a caso, com base na literatura e em estudos de relação estrutura-atividade e carcinogenicidade. As NAs mais discutidas na área farmacêutica estão apresentadas na Tabela 2 (ICH, 2017; ANVISA, 2021a; FDA, 2021a).
Os limites de ingestão diária de NAs dependem da dose máxima diária do medicamento em questão. No caso de NAs novas ou desconhecidas, pode ser adotado um valor conservador de 18 ng/dia. Ressalta-se que os limites atualmente disponíveis são passíveis de alteração diante do surgimento de novas evidências científicas (ANVISA, 2021a).
A ação carcinogênica das NAs requer ativação enzimática no organismo, isto é, a hidroxilação do carbono ɑ do grupo alquila, catalisada pela monoxigenase do citocromo P450, com formação de aldeído ou cetona e uma NA primária instável. A tautomerização para um alquildiazoidróxido dá origem ao íon diazônio, que é responsável pela alquilação dos sítios nucleofílicos do material genético, causando lesões citotóxicas e mutações (RATH; CANAES, 2009, ADAMSON; CHABNER, 2020).
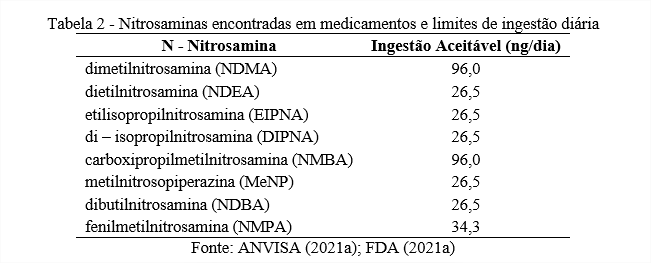
A ferramenta mais importante a ser utilizada na investigação de NAs em medicamentos e IFAs é o gerenciamento de risco, que abrange três etapas: (i) avaliação do risco relacionado a qualquer elemento envolvido na obtenção do IFA e do produto acabado, incluindo a revisão de processos, meios para a eliminação (purga) e grau de carcinogenicidade de possíveis NAs; (ii) testes confirmatórios utilizando métodos analíticos sensíveis para a quantificação; (iii) alterações pós-registro caso seja necessário adequar especificações e propor medidas de monitoramento (ICH, 2005; ANVISA, 2021a).
Estudos de carcinogenicidade e mutagenicidade podem ser necessários às investigações. O ensaio Salmonella/microssoma, conhecido como teste de Ames, é adotado por muitos laboratórios para avaliação de mutagenicidade, visto que é um ensaio in vitro reprodutível e de curta duração, que utiliza linhagens bacterianas (ANVISA, 2013; CETESB, 2021).
O desenvolvimento de métodos analíticos aplicáveis ao controle de impurezas do tipo NA em medicamentos e IFAs é uma necessidade emergente e um grande desafio. As NAs podem ser voláteis ou não voláteis, o que influencia no desenvolvimento de metodologias. A maioria das NAs encontradas em medicamentos são de natureza volátil, tais como NDMA, NDEA, NDPA e NDBA (RATH; CANAES, 2009).
Diferentes metodologias são descritas para a determinação de NAs nas mais variadas matrizes. Os métodos de pré-tratamento mais comuns são extração em fase sólida, extração líquido-líquido e procedimentos de microextração. Já os métodos de quantificação incluem cromatografia líquida (CLAE) ou gasosa (CG), cromatografia de fluido supercrítico, métodos eletroquímicos, entre outros (Parr; Joseph, 2019; Alshehri; Alghamdi; Aldawsari, 2020; Bian et al., 2020).
Segundo Tuesuwan e Vongsutilers (2021), há atualmente mais de 40 métodos analíticos disponíveis para a determinação de NAs em medicamentos e IFAs, porém apenas alguns deles fornecem o limite de quantificação recomendado de 0,03 ppm. Dentre estes métodos se destacam a CLAE em fase reversa acoplada à espectroscopia de massas (EM) de alta resolução ou EM em tandem, utilizando fontes de ionização por spray de elétrons ou ionização química à pressão atmosférica e analisadores do tipo quadrupolo ion trap ou triplo quadrupolo (QqQ). No caso de CG acoplada à espectrometria de massas, destaca-se a injeção direta ou headspace, com fonte de ionização eletrônica e analisadores QqQ ou ion trap.
Neste contexto, opções de métodos para a detecção e quantificação de impurezas do tipo NA em alguns IFAs e produtos acabados já foram publicados pelo FDA. Estes métodos são úteis para apoiar avaliações de qualidade ou submissões regulamentares e devem ser previamente validados pelo usuário (FDA, 2021b).
CONCLUSÕES
O controle de impurezas do tipo NA em medicamentos e insumos farmacêuticos ativos é um tema recente e que ainda se encontra em ampla discussão entre os órgãos reguladores, o setor regulado e a comunidade científica. O gerenciamento de risco é a principal estratégia adotada, necessitando da atuação multiprofissional de farmacêuticos, químicos e toxicologistas. As análises laboratoriais de identificação e quantificação requerem técnicas de elevada sensibilidade, como a cromatografia gasosa ou líquida acoplada à espectroscopia de massas. Apesar das NAs se apresentarem em concentrações reduzidas, as ações de vigilância são relevantes, especialmente quando envolve medicamentos de uso contínuo e prolongado para tratar doenças crônicas.
REFERÊNCIAS
ADAMSON, R.H.; CHABNER, B.A. The finding of N-nitrosodimethylamine in common medicines. The Oncologist, v.25, n.6, p.460–462, 2020.
ALSHEHRI, Y.M.; ALGHAMDI, T.S.; ALDAWSARI, F.S. HS-SPME-GC-MS as an alternative method for NDMA analysis in ranitidine products. Journal of Pharmaceutical And Biomedical Analysis, v.191, p.1-6, 2020.
ANVISA. Guia para a condução de estudos não clínicos de toxicologia e segurança farmacológica necessários ao desenvolvimento de medicamentos. Brasília, 2013.
ANVISA. Parecer nº 3/2020/Sei/Gimed/Ggfis/Dire4/Anvisa: proposta de RDC sobre avaliação de risco e controle de nitrosaminas potencialmente carcinogênicas em medicamentos de uso humano. Brasília, 2020.
ANVISA. Guia sobre o controle de nitrosaminas em insumos farmacêuticos ativos e medicamentos: guia nº 50/2021, versão 1. Brasília, 2021a.
ANVISA. RDC nº 494, de 15 de abril de 2021. Altera a RDC nº 283, de 17 de maio de 2019, que dispõe sobre investigação, controle e eliminação de nitrosaminas potencialmente carcinogênicas em antagonistas de receptor de angiotensina II. Brasília, 2021b.
BIAN, Y.; et al. Progress in the pretreatment and analysis of N-nitrosamines: an update since 2010. Critical Reviews in Food Science and Nutrition, v.61, n.21, p.1-35, 2020.
BRUNTON, L.L.; CHABNER, B.A.; KNOLLMANN, B.C. (org.). As Bases Farmacológicas da Terapêutica de Goodman & Gilman. 12. ed. Brasil: Amgh Editora, 2012.
CETESB. Ensaios de Genotoxicidade. Disponível em: https://cetesb.sp.gov.br/laboratorios/wp-content/uploads/sites/24/2013/11/ensaios-genotoxicidade-saiba-mais.pdf. Acesso em: 21 dez. 2021.
EMA. Referral under article 31 of directive 2001/83/EC angiotensin-II-receptor antagonists (sartans) containing a tetrazole group. Assessment report EMA 217823, p.1-41, 2019.
EMA. Questions and answers for marketing authorisation holders/applicants on the CHMP Opinion for the Article 5(3) of Regulation (EC) Nº 726/2004 referral on nitrosamine impurities in human medicinal products. EMA/409815/2020 Rev.6, p.1-17, 2021a.
EMA. Ranitidine-containing medicinal products. Disponível em: https://www.ema.europa.eu/en/medicines/human/referrals/ranitidine-containing-medicinal-products. Acesso em: 07 dez. 2021b.
FDA. Control of nitrosamine impurities in human drugs: guidance for industry, Revision 1. Silver Spring, 2021a.
FDA. Information about nitrosamine impurities in medications. Disponível em: https://www.fda.gov/drugs/drug-safety-and-availability/information-about-nitrosamine-impurities-medications. Acesso em: 06 dez. 2021b.
GUSHGARI, A.J.; HALDEN, R.U. Critical review of major sources of human exposure to N-nitrosamines. Chemosphere, v. 210, p.1124–1136, 2018.
ICH. Manual tripartite harmonizado do ICH Gestão do Risco à Qualidade Q9, 2005.
ICH. Guia harmonizado do ICH Avaliação e controle de impurezas reativas ao DNA (mutagênicas) em produtos farmacêuticos para limitar o risco carcinogênico em potencial M7 (R1), 2017.
PARR, M.K.; JOSEPH, J.F. NDMA impurity in valsartan and other pharmaceutical products: Analytical methods for the determination of N-nitrosamines. Journal of Pharmaceutical and Biomedical Analysis, v.164, p.536-549, 2019.
PASCHOAL, A.D. O orgânico nosso de cada dia. Rede Câncer, v.31, p. 20–21, 2015.
RATH, S.; CANAES, L.S. Contaminação de produtos de higiene e cosméticos por N-nitrosaminas. Química Nova, v.32, n.8, p.2159–2168, 2009.
THRESHER, A.; et al. Are all nitrosamines concerning? A review of mutagenicity and carcinogenicity data. Regulatory Toxicology and Pharmacology, v.116, article 104749, 2020.
TUESUWAN, B.; VONGSUTILERS, V. Nitrosamine contamination in pharmaceuticals: threat, impact, and control. Journal of Pharmaceutical Sciences, v.110, n.9, p.3118-3128, 2021.
Resumo expandido publicado nos Anais da III Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
Este trabalho foi escrito por:
Gabrielle Yuriko Batista de Moraes1; Ana Cristina Scarparo de Miranda2; Tamiko Ichikawa Ikeda2; Aurea Silveira Cruz Garçon2
1Estudante do Curso de Especialização em Vigilância Laboratorial em Saúde Pública – IAL; E-mail: [email protected]
2Docente/Pesquisadora do Núcleo de Cultura de Células – NCC – IAL.
Resumo: As culturas de células são amplamente utilizadas em diversas áreas, na pesquisa ou diagnóstico, além de serem uma excelente alternativa ao uso de testes com animais. Essas culturas devem ser autenticadas, tendo as suas características conhecidas, a espécie de origem comprovada e estarem livres de contaminação cruzada e microbiológica, especialmente por micoplasmas, o que pode evitar a perda de recursos financeiros, conhecimento científico e a credibilidade dos trabalhos publicados. Esta revisão tem como objetivo abordar e discorrer sobre algumas técnicas recomendadas para autenticação celular, assim como suas vantagens e limitações, colaborando com a conscientização da comunidade científica sobre a necessidade de utilizar linhagens celulares obtidas de coleções reconhecidas e a importância do constante monitoramento das mesmas. Sendo assim, foi realizada uma vasta busca na literatura sobre este tema e, de acordo com o levantamento, a comprovação da ausência de micoplasmas pode ser feita por cultura microbiológica, coloração fluorescente, técnica de ELISA, PCR, microscopia eletrônica ou método bioquímico (MycoAlertTM). Com relação à verificação de espécie, as metodologias disponíveis são eletroforese de isoenzimas, análise cromossômica, marcação imunológica e técnicas de biologia molecular para análise de regiões conservadas do genoma. Como toda técnica possui vantagens e limitações, deve-se empregar mais de uma para assegurar a identidade e ausência de contaminantes da linhagem utilizada. Além do uso destes ensaios para controle é essencial a utilização de técnicas assépticas em todas as etapas da manutenção das culturas, evitando a introdução de possíveis contaminantes. Todos estes cuidados são fundamentais para o sucesso dos estudos realizados com culturas celulares.
Palavras–chave: autenticação de linhagens celulares; contaminação; contaminação cruzada; cultura de células.
INTRODUÇÃO
Cultura de células é definida como um modelo in vitro que permite manter as células vivas, não mais organizadas em tecidos e órgãos. Essa técnica apresenta muitas vantagens em comparação aos modelos in vivo que utilizam animais em ensaios experimentais, tais como homogeneidade das linhagens, são mais econômicos, de fácil manipulação e controle, além de ser uma alternativa para substituir e/ou reduzir a utilização de animais em pesquisa. Suas aplicações vão da área de pesquisa ao diagnóstico, principalmente em Biologia Celular e Molecular, Bioquímica, Virologia, Farmacologia, Toxicologia e Oncologia, entre outras (PERES; CURI, 2005; ALVES; GUIMARÃES, 2010; MIGITA, 2012; GONÇALVES; SOBRAL, 2020).
Para que o conhecimento produzido com a utilização de células seja válido e reprodutível, é essencial que as linhagens celulares sejam autenticadas, ou seja, que sua identidade seja confirmada, espécie de origem comprovada e estejam livres de contaminantes, seja micro-organismos e/ou outras linhagens celulares. O uso de culturas contaminadas ou erroneamente identificadas gera resultados inválidos e consequentemente compromete a credibilidade da pesquisa realizada, além de disseminar informações equivocadas (CAPES-DAVIS et al., 2010; MARX, 2014; FREEDMAN et al., 2015; HORBACH; HALFFMAN, 2017; COSME et al., 2017).
Os principais micro-organismos que podem contaminar as culturas celulares são as bactérias, fungos e vírus. Dentre esses, os micoplasmas são as menores bactérias de vida livre, não possuem parede celular e pertencem à classe Mollicutes. São os contaminantes mais comuns em culturas celulares e um dos mais difíceis de identificar, podendo permanecer indetectáveis por várias passagens e alterar diversas características e respostas celulares (DREXLER; UPHOFF, 2002; MAHMOOD; ALI, 2017). Outro tipo de contaminação que pode ocorrer durante a cultura de células é a cruzada, quando diferentes linhagens celulares são misturadas ou identificadas erroneamente, resultando em uma cultura que não é mais condizente ao seu doador, tecido ou espécie de origem (ICLAC, 2022). As estimativas são de que cerca de 20 a 30 % das linhagens celulares possam estar contaminadas com outras ou erroneamente identificadas e de 5 a 35 % por micoplasmas, demonstrando a extensão desse problema (DREXLER; UPHOFF, 2002; CAPES-DAVIS et al., 2010; YOUNG; SUNG; MASTERS, 2010).
Por isso, antes de iniciar qualquer estudo é importante buscar maiores informações sobre as células que serão utilizadas em sites especializados como do Comitê Internacional de Autenticação de Linhagens Celulares – ICLAC e The Cellosaurus, entre outros (BAIROCH, 2018). Além disso, as linhagens celulares devem ser adquiridas, preferencialmente, de bancos de células reconhecidos, que possuem um controle de qualidade rigoroso (CAPES-DAVIS et al., 2010; GERAGHTY et al., 2014; MARX, 2014).
Para reduzir essa problemática, Nardone (2007) também sugere que as revistas e instituições de fomento à pesquisa exijam a comprovação da autenticação das linhagens para publicação de resultados e liberação de recursos financeiros, o que vem sendo adotado por algumas organizações, entre outras ações.
Este trabalho tem como objetivos abordar a importância da autenticação de linhagens celulares, discorrer sobre as principais técnicas disponíveis, assim como suas vantagens e limitações, o que colabora com a conscientização da comunidade científica sobre a necessidade de utilizar células obtidas de coleções reconhecidas e necessidade do constante monitoramento das mesmas.
METODOLOGIA
Esta revisão de literatura aborda o tema de autenticação celular e as principais técnicas recomendadas. Sendo assim, foi realizada uma consulta a base de dados do PubMed (https://www.ncbi.nlm.nih.gov/pubmed), utilizando os descritores: “cell culture”, “authentication”, “contamination”, “cross-contamination”, “misidentification”, “authentication techniques”; “authentication methods of cell culture”. As publicações foram avaliadas de acordo com título e resumo pertinentes ao tema e importância histórica e científica. Informações complementares foram obtidas no site do Comitê Internacional de Autenticação de Linhagens Celulares – ICLAC (https://iclac.org/).
RESULTADOS E DISCUSSÃO
Existem diversas técnicas que podem ser empregadas para autenticar as linhagens celulares, tanto na comprovação da ausência de contaminação por micoplasmas, quanto na verificação da espécie de origem para garantir que as mesmas não foram contaminadas com outra linhagem celular ou erroneamente identificadas.
Para a detecção de micoplasmas pode-se utilizar ensaios de cultivo microbiológico, coloração fluorescente, imunocoloração, PCR (Polymerase Chain Reaction), microscopia eletrônica e detecção bioquímica. O cultivo microbiológico foi uma das primeiras técnicas padronizadas, muito utilizado e considerado como “padrão-ouro”. Esse método possui alta sensibilidade e especificidade, permitindo o reconhecimento visual das colônias em ágar, com aspecto típico de “ovo frito”. No entanto, o crescimento desses micro-organismos é um processo demorado (três a quatro semanas) e nem todas as espécies de micoplasmas são cultiváveis (ATCC, 1992; DREXLER; UPHOFF, 2002; PERES; CURI, 2005; YOUNG; SUNG; MASTERS, 2010; GERAGHTY et al., 2014; WRIGLEY et al., 2014).
O método de coloração fluorescente, utilizando DAPI (4′,6-Diamidine-2′-phenylindole dihydrochloride) ou Hoechst 33258 (2′-(4-hydroxyphenyl)-5-(4-methyl-1-piperazinyl)-2,5′-bi-1H-benzimidazole trihydrochloride hydrate), é uma opção barata e relativamente rápida de ser realizada. Essas substâncias coram diretamente o DNA de forma inespecífica e, ao ser visualizado em microscópio com luz ultravioleta, permite observar pontos fluorescentes sobre e ao redor dos núcleos celulares, indicando contaminação (DREXLER; UPHOFF, 2002; PERES; CURI, 2005; GERAGHTY et al., 2014; ATCC, 2022).
A técnica de PCR também pode ser empregada na detecção de micoplasmas, é rápida e com elevada sensibilidade, geralmente empregando primers (oligonucleotídeos iniciadores) universais que identificam regiões conservadas entre as diferentes espécies. Há muitos protocolos disponíveis, seja por PCR convencional ou em tempo real, além de opções de kits comerciais de diversos fornecedores (DREXLER; UPHOFF, 2002; WRIGLEY et al., 2014).
Além dessas, é possível realizar a detecção bioquímica por meio de kits comerciais, como o MycoAlertTM, baseado na detecção de enzimas desses micro-organismos, avaliando os níveis de adenosina trifosfato (ATP) por luminescência. Embora tenha uma sensibilidade mediana, apresenta alta especificidade, permite a obtenção do resultado rapidamente, além de possuir controle positivo interno sem necessitar manipular culturas de micoplasmas (PERES; CURI, 2005; GERAGHTY et al., 2014; MAHMOOD; ALI, 2017; ATCC, 2022).
Para a confirmação da espécie de origem existem várias técnicas disponíveis, como eletroforese de isoenzimas, cariótipo, marcação imunológica, DNA fingerprint para análises das regiões de Short Tandem Repeat (STR), Variable Number of Tandem Repeat (VNTR), Single Nucleotide Polymorphism (SNP) e DNA barcode (CAPES-DAVIS et al., 2010; NIMS; REID, 2017; CAPES-DAVIS, 2018; ATCC, 2022).
Dentre essas, a eletroforese de isoenzimas é um método bioquímico que se baseia na diferença dos padrões estruturais apresentados por algumas enzimas entre as espécies, sem alterar sua função. Essas variações estruturais podem ser identificadas pela mobilidade eletroforética das isoenzimas presentes no extrato celular. A revelação do gel é feita utilizando substratos e cofatores específicos para a isoenzima desejada. Após a coloração das bandas, as distâncias de migração podem ser mensuradas e devem ser comparadas às linhagens usadas como controle e padrão do ensaio, NCTC clone 929 e HeLa, respectivamente (KAPLAN; HUKKU, 1998; NIMS et al., 1998; NIMS et al., 2010; ATCC, 2022).
Mesmo com o surgimento de metodologias baseadas em biologia molecular, a técnica de isoenzimas é de grande valia na comprovação da espécie de origem de linhagens celulares, pois a mesma é acessível, rápida, apresenta custo relativamente baixo e é capaz de detectar contaminação cruzada quando as células contaminantes correspondem a cerca de 10 % do número de células totais (NIMS et al., 1998; MACHADO et al., 2016; NIMS; REID, 2017).
Outra técnica disponível é a análise cromossômica, uma das primeiras técnicas utilizadas para identificação interespecífica, capaz de detectar rearranjos cromossômicos que são característicos de cada espécie. Para tal, devem ser analisadas ao menos 50 metáfases, o que permite a identificação do número de cromossomos, presença de possíveis marcadores, variações na ploidia e diferenciação de espécies (KAPLAN; HUKKU, 1998; CAPES-DAVIS et al., 2010). Porém, é uma metodologia trabalhosa e demorada, que necessita profissionais experientes para a correta interpretação dos resultados obtidos (KAPLAN; HUKKU, 1998; RAMYA et al., 2009; CAPES-DAVIS, 2018; ATCC, 2022).
A técnica de PCR é relativamente simples, robusta e sensível, amplamente utilizada para detecção de espécies (STACEY et al., 1997; RAMYA et al., 2009). Para a autenticação de linhagens não humanas pode ser empregada a técnica de DNA barcode, com a amplificação do gene do Citocromo C Oxidade subunidade I (COI) ou Citocromo B, ambos presentes no DNA mitocondrial (DNA Mt) e que apresentam variações entre as espécies, permitindo diferenciá-las. Ono e colaboradores (2007) utilizaram nested-PCR, com primers universais para uma região mitocondrial conservada (citocromo B, alça-D, RNAr 12S e 16S) na primeira reação e, em seguida, esse produto deve ser amplificado utilizando primers específicos para distinguir as espécies. Essa variação da PCR apresentou maior sensibilidade que a análise de isoenzimas, sendo capaz de detectar contaminações menores que 1% (ONO et al., 2007; RAMYA et al., 2009; NIMS; REID, 2017).
Para linhagens de células humanas, recomenda-se a técnica de STR fingerprint, baseado em sequências curtas de DNA, que permite a diferenciação de linhagens de indivíduos distintos. Essa técnica amplifica regiões de repetições com até cinco pares de bases dos marcadores polimórficos do genoma humano, com subsequente separação dos fragmentos em eletroforese capilar, que resulta em um padrão único. Recomenda-se analisar ao menos oito locus de STR mais amelogenina para diferenciação sexual. Os padrões obtidos consistem no número de repetições das sequências em cada alelo para os locus analisados e devem ser comparados com aqueles disponíveis em bancos de dados, como do ICLAC ou de biorrepositórios. Como variações nos padrões de STR podem ser observadas, foi estabelecido um mínimo de 80% de coincidência para que uma linhagem seja considerada autêntica; valores abaixo disso sugerem identidade suspeita ou mesmo que não corresponde à esperada, se a comparação revelar menos que 55% de compatibilidade (ICLAC, 2014). A análise de STR não permite identificar se uma linhagem humana foi contaminada por células de outra espécie animal, sendo necessário complementar a verificação com outro método.
Por conseguinte, é necessário que sejam feitos testes periódicos para garantir a autenticidade das linhagens celulares, tanto para ausência de micoplasmas quanto para confirmação da espécie de origem. Como todas as técnicas apresentam vantagens e limitações, o recomendado é empregar mais de uma metodologia para confirmar os resultados obtidos nos ensaios.
CONCLUSÕES
A autenticação das linhagens celulares é fundamental para garantir a validade dos resultados obtidos com sua utilização, evitando dois grandes problemas que afetam o uso de culturas de células, a ocorrência de contaminação cruzada e por micro-organismos, especialmente por micoplasmas. Há várias técnicas disponíveis para essas verificações e a escolha do método de autenticação, bem como a periodicidade, dependem da estrutura e condições do laboratório. Além disso, recomenda-se que seja empregada mais de uma metodologia, já que todas possuem suas vantagens e limitações. Ao assegurar a espécie de origem das células e a ausência de contaminantes, evita-se o desperdício de recursos financeiros e a perda de credibilidade, além de garantir a reprodutibilidade dos estudos com culturas celulares.
REFERÊNCIAS
ALVES, E. A.; GUIMARÃES, A. C. R. Cultivo celular. In: MOLINARO, E. M.; CAPUTO, L. F. G.; AMENDOEIRA, M. R. R. (Org.). Conceitos e métodos para a formação de profissionais em laboratórios de saúde. v. 2. Rio de Janeiro: EPSJV, p. 215-253, 2010.
ATCC. American Type Culture Collection. Authentication Test Recommendations. <https://www.atcc.org/resources/technical-documents/cell-line-authentication-test-recommendations>. Acesso em: 04 jan. 2022.
ATCC. American Type Culture Collection. Quality Control Methods for Cell Lines. 2 ed. United States of America, 1992.
BAIROCH, A. The Cellosaurus, a cell-line knowledge resource. Journal of Biomolecular Techniques. v. 29, n. 2, 2018.
CAPES-DAVIS, A. Cell Line Detective Work. Advances in Molecular Pathology, v. 1, n. 1, p. 229–238, 2018.
CAPES-DAVIS, A. et al. Check your cultures! A list of cross-contaminated or misidentified cell lines. International Journal of Cancer, v. 127, n. 1, p. 1–8, 2010.
COSME, B. et al. Are your results valid? Cellular authentication a need from the past, an emergency on the present. In Vitro Cellular and Developmental Biology – Animal, v. 53, n. 5, p. 430–434, 2017.
DREXLER, H. G.; UPHOFF, C. C. Mycoplasma contamination of cell cultures: Incidence, sources, effects, detection, elimination, prevention. Cytotechnology, v. 39, n. 2, p. 75–90, 2002.
FREEDMAN, L. P. et al. Reproducibility: Changing the policies and culture of cell line authentication. Nature Methods, v. 12, n. 6, p. 493–497, 2015.
GERAGHTY, R. J. et al. Guidelines for the use of cell lines in biomedical research. British Journal of Cancer, v. 111, n. 6, p. 1021–1046, 2014.
GONÇALVES, J. C. R.; SOBRAL, M. V. Cultivo de células: da teoria à bancada. João Pessoa: Editora UFPB, 2020.
HORBACH, S. P. J. M.; HALFFMAN, W. The ghosts of HeLa: How cell line misidentification contaminates the scientific literature. PLoS ONE, v. 12, n. 10, p. 1–16, 2017.
ICLAC. International Cell Line Authentication Committee. Disponível em: <https://iclac.org/>. Acessoem: 04 jan. 2022.
ICLAC. International Cell Line Authentication Committee. Guide to human cell line authentication, 2014. Disponível em: <https://iclac.org/resources/human-cell-line-authentication/>. Acesso em: 04 jan. 2022.
KAPLAN, J.; HUKKU, B. Cell line characterization and authentication. Methods in Cell Biology, v. 57, n. 57, p. 203–216, 1998.
MACHADO, J. Z.; MIRANDA, A. C. S.; CRUZ, A. S. G6PD and MDH Isoenzyme Electrophoresis for Cell Lines Authentication. Virus Review and Research, v. 21, p. 1-4, 2016.
MAHMOOD, A.; ALI, S. Microbial and Viral Contamination of Animal and Stem Cell Cultures: Common Contaminants, Detection and Elimination. Journal of Stem Cell Research & Therapeutics, v. 2, n. 5, p. 149–155, 2017.
MARX, V. Cell-line authentication demystified. Nature Methods, v. 11, n. 5, p. 483–488, 2014.
MIGITA, N. A. “Cultivo celular in vitro : importância para a pesquisa biomédica e dimensão da problemática de autenticação de linhagens celulares”. Monografia, Instituto de Biociências de Botucatu, UNESP, 2012.
NARDONE, R. M. Eradication of cross-contaminated cell lines: A call for action. Cell Biology and Toxicology, v. 23, n. 6, p. 367–372, 2007.
NIMS, R. W. et al. Sensitivity of isoenzyme analysis for the detection of interspecies cell line cross-contamination. In Vitro Cellular and Developmental Biology – Animal, v. 34, n. 1, p. 35–39, 1998.
NIMS, R. W. et al. Short tandem repeat profiling: Part of an overall strategy for reducing the frequency of cell misidentification. In Vitro Cellular and Developmental Biology – Animal, v. 46, n. 10, p. 811–819, 2010.
NIMS, R. W.; REID, Y. Best practices for authenticating cell lines. In Vitro Cellular and Developmental Biology – Animal, v. 53, n. 10, p. 880–887, 2017.
ONO, K. et al. Species identification of animal cells by nested PCR targeted to mitochondrial DNA. In Vitro Cellular and Developmental Biology – Animal, v. 43, n. 5–6, p. 168–175, 2007.
PERES, C. M.; CURI, R.; PAFFARO, A. M. D. A.; MARTINS, A. K. A.; PIMENTA, A.; GONÇALVES, C. R; MARTINS, E. F. Como cultivar células. Rio de Janeiro: Guanabara-Koogan, 2005.
RAMYA, R. et al. Identification of cross-contaminated animal cells by PCR and isoenzyme analysis. Cytotechnology, v. 61, n. 3, p. 81–92, 2009.
STACEY, G. N.; HOELZL, H.; Stephenson, J. R.; Doyle, A. Authentication of animal cell cultures by direct visualization of repetitive DNA, aldolase gene PCR and isoenzyme analysis. Biologicals v. 25, p. 75–85, 1997.
WRIGLEY, J. D. et al. Cell banking for pharmaceutical research. Drug Discovery Today, v. 19, n. 10, p. 1518–1529, 2014.
YOUNG, L.; SUNG, J.; MASTERS, J. R. Detection of mycoplasma in cell cultures. Nature Protocols, v. 5, n. 5, p. 929–934, 2010.
