Capítulo de Livro IV Mostra
Capítulo de livro publicado no Livro da IV Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
doi.org/10.53934/9786599965821-26
Este trabalho foi escrito por:
Marcela Oliveira de Toledo1, Cinthya dos Santos Cirqueira2, Amaro Nunes Duarte Neto2, Thaís de Souza Lima2
1Estudante do curso de Vigilância Laboratorial em Saúde Pública- Programa de Imuno-histoquímica- CPA/NAP- IAL; E-mail: [email protected]
2Docente/pesquisador do Depto de Anatomia Patológica – NAP – IAL
Resumo: A Monkeypox (MPOX) é uma doença febril e exantemática aguda, causada por um poxvírus pertencente à família Poxviridae. O primeiro caso foi relatado na África Ocidental e Centralem 1971. Em 2003, houve um surto fora da África, e em maio de 2022 foi confirmado o primeiro caso do atual surto global. O presente estudo teve por objetivo realizar uma revisão bibliográfica visando, compreender os aspectos epidemiológicos e patológicos do MPOX. Foram selecionados artigos através das bases de dados PubMed, Science Direct e Web of Science. Entre os 250 artigos encontrados, 6 foram escolhidos e ajudaram na compreensão sobre o diagnóstico histopatológico, além de apontarem as perspectivas futuras para esse surto. Verificou-se que aumento da vigilância e detecção de casos são ferramentas essenciais para entender a epidemiologia dessa doença reemergente, assim como a contribuição das análises histopatológicas de necropsias, visando elucidar os mecanismos virais específicos e o comprometimento sistêmico pela infecção.
Palavras-chave: Autópsia, histologia
INTRODUÇÃO
A Monkeypox (MPOX) é uma doença febril e exantemática aguda, causada por um poxvírus pertencente à família Poxviridae, uma grande e diversificada família de vírus DNA de fita dupla, que se multiplica no citoplasma das células infectadas (ALAKUNLE etal.,2020). É membro do gênero Orthopoxvirus, grupo que apresenta outros integrantes relevantes como o vírus da varíola (Smallpox), agente causador da varíola e o vírus vaccinia (vírus usado na vacina contra a varíola humana) (WEINSTEIN et al.,2005).
O primeiro caso humano de MPOX na história médica foi relatado em 10 de setembro de 1970, em uma criança de nove meses, na República Democrática do Congo (RDC) desde então, o MPOX tornou-se endêmico em vários países africanos, principalmente na África Central e Ocidental (LIU et al.,2022). Todos os casos na RDC ocorreram em áreas que fazem fronteira com florestas tropicais e pareciam estar associados ao contato com animais (WEINSTEIN et al., 2005).
No verão de 2003, o MPOX foi identificado como a causa de um surto de varíola nos Estados Unidos (EUA), fato que representou a primeira ocorrência de MPOX fora da África. A maioria das pessoas infectadas adoeceu após o contato com cães-da-pradaria domésticados (WEINSTEIN et al., 2005). Desde 2003, casos de MPOX foram relatados em vários países.
Em sete de maio de 2022, no Reino Unido, foi confirmado o 1º caso do atual surto global de MPOX fora de áreas endêmicas. No Brasil, o primeiro caso ocorreu em nove de junho de 2022, registrado no estado de São Paulo (Brasil, 2022). No atual surto a transmissão ocorreu de pessoa para pessoa, sem ligação com viagens aos países endêmicos. Até o momento, as pesquisas não apontam a ocorrência de cadeias de transmissão conhecidas (DE BONA et al., 2022).
Os dois meios possíveis de transmissão do MPOX são a transmissão animal- humano e a transmissão humano-humano. O contato com pele lesionada de uma pessoa infectada, gotículas respiratórias, contato com fluidos corporais, ambiente ou itens contaminados do paciente, foram associados à transmissão humano-humano (ALAKUNLE et al.,2020).
Após um período de incubação de 10 a 14 dias, febre, mal-estar e linfadenopatia são observados, na maioria dos pacientes, antes do desenvolvimento de erupção cutânea (WEINSTEIN et al., 2005). A lesão cutânea típica progride através de um estágio papular, vesicular, pustulosa e crostosa, durante um período de 14 a 21 dias, antes que a descamação da crosta deixe uma cicatriz despigmentada (GARNER, 2005). A MPOX é geralmente uma doença autolimitada que dura de 2 a 4 semanas, até que as lesões descamam, mas pode resultar em doença grave em crianças e pacientes com imunodeficiências (LIU et al.,2022)
No atual surto, a maioria dos casos ocorreu em homens que se identificam como homens que fazem sexo com outros homens (HSH), com histórico de exposição durante a relação sexual. Acredita-se que a transmissão através do contato sexual, seja o principal fator do surto atual (CCD, 2022).
O diagnóstico laboratorial é feito por método molecular, que envolve o uso de PCR em tempo real (qRT-PCR). Como método alternativo, está disponível o exame histopatologico, com a pesquisa imuno-histoquímica, que permite a detecção do antígeno viral no tecido, distinguindo entre as infecções por poxvírus e herpes vírus, reservado para casos em que o diagnóstico diferencial é nebuloso, e com poucos relatos na literatura (ALAKUNLE et al,2020).
O único fármaco disponível para tratamento do MPOX é o Tecovirimat (comercializado como TPOXX, SIGA Technologies, USA), um antiviral que inibe a replicação do patógeno. Na última semana de agosto, o Brasil recebeu os primeiros antivirais para o tratamento sendo autorizado o uso compassivo em casos graves da doença (Brasil, 2022).
MATERIAL E MÉTODOS
O presente estudo trata-se de uma revisão bibliográfica cujas bases de dados escolhidas para seleção de artigos foram, PubMed, Science Direct e Web of Science. As palavras-chave empregadas para a busca de estudos cientificos originais foram “monkeypox”, “histology”, “histopathology”, “autopsy” com o proposito de ressaltar os principais dados epidemiologicos e histopatologicos da doença.
RESULTADOS E DISCUSSÃO
As doenças reemergentes indicam uma mudança no comportamento epidemiológico de doenças conhecidas, que haviam sido contidas, mas que voltaram a configurar ameaça a saúde humana, como por exemplo, o surto de MPOX em 2022 (DE BONA et al., 2022).
Através da pesquisa dentro da base de dados PubMed, utilizando as palavras chaves
“monkeypox”, “histology”, “histopathology”,”autopsy”, foram encontrados 250 artigos. Os abstracts dos artigos foram examinados e seis artigos foram selecionados, pois se enquadravam como artigos originais sobre a histopatologia do MPOX, descrevendo a patologia da doença ocorrida durante o atual surto e epidemias anteriores, em diferentes regiões do mundo, conforme quadro 1.
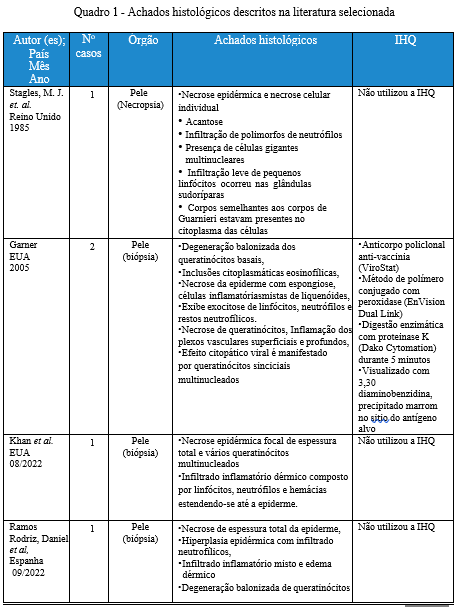
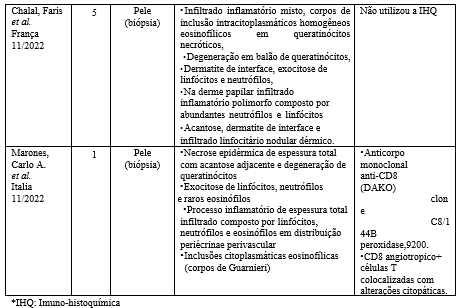
A literatura apresenta poucos relatos sobre achados histológicos, o presente trabalho utilizou dois estudos de surtos anteriores, um ocorrido no Zaire, área endêmica no ano de 1985 e o outro nos estados unidos em 2005, os demais são relatos do atual surto.
Oito dos casos de biopsia de pele citados, foram em pacientes que se enquadram nas seguintes características: todos do sexo masculino que se identificaram como HSH. Já o artigo de Garner (2005) não menciona a opção sexual dos pacientes. O diagnóstico foi confirmado por meio de método molecular em todas as biopsias de pele, e apenas um paciente era HIV positivo. O artigo de Stagles (1985) relatou o caso de uma criança que morreu após uma doença de cinco dias, no Zaire. Foi o primeiro relato da histopatologia deuma lesão cutânea de MPOX no ser humano.
Em todos os casos citados e descritos, a apresentação clínica foi similar, todos tiveram lesões cutâneas vesicopustulosas típicas e linfadenopatia, somente um apresentou complicações graves ocasionando o óbito. No surto atual, uma característica comum entre os casos foi a localização das lesões, elas estão presentes em sua maioria na região genital e ânus, diferente dos relatos anteriores onde as lesões apareciam no rosto, na palma das mãos e na sola dos pés (PATEL et al.,2022)
Os achados histopatológicos foram homogêneos em vários pontos nos onze pacientes, demonstrando degeneração balonizada dos queratinócitos, queratinócitos necróticos, contendo corpos de inclusão eosinofílicos, esféricos, homogêneos, intracitoplasmáticos do tipo Guarnieri, presença de infiltrado inflamatório dérmico misto, às vezes, com eosinófilos e linfócitos profundos perivasculares.
A histopatologia em outros órgãos não foi relatada, apesar da literatura já descrever complicações clínicas desde os surtos anteriores a 2022, como o caso descrito por Stangles (1985), onde não ocorreu nenhuma investigação em outros órgãos para determinar a extensão da doença, seu comprometimento sistêmico e a possível causa do óbito.
Uma situação parecida ocorreu com Menezes et al. (2022) que relata a primeira morte por MPOX fora da área endêmica, que ocorreu no Brasil no final de julho de 2022.
O homem de 41 anos, portador do vírus HIV e que se identificava HSH, relatou seu último ciclo de quimioterapia em cinco de julho de 2022, para o tratamento de linfoma difuso de grandes células B com metástases na coluna, crânio e fígado (MENEZES et al.,2022). As primeiras lesões cutâneas surgiram na fronte e rapidamente progrediu para o restante do corpo, o quadro clínico evoluiu rapidamente, o paciente desenvolveu repentinamente dificuldade respiratória grave, lesão renal aguda e falência múltipla de órgãos, sendo transferido para a unidade de terapia intensiva (UTI) onde faleceu (MENEZES et al.,2022).
As disfunções em múltiplos órgãos sugeriram que a causa mais provável da morte fosse sepse, mas não houve crescimento bacteriano em várias hemoculturas e o paciente estava sob antibioticoterapia de amplo espectro. Menezes et al. (2022) levantou outras hipóteses, incluindo um envolvimento visceral da doença e destacou como limitação a falta de uma necropsia. A realização de uma necropsia ajudaria a determinar até que ponto o MPOX afetou o organismo do paciente, determinando se a morte de fato foi causada por MPOX ou se o paciente foi a óbito, devido uma condição de saúde subjacente. Este unico artigo demonstra carencia de relatos investigativos acerca da causa das mortes, apontando como perspectivas futuras, a necessidade da ampliação de estudo anatomopatologico das necropsias para a contribuição da elucidação de obitos suspeitos por MPOX.
CONCLUSÃO
De acordo com esse trabalho podemos esclarecer que, o atual surto demonstra mudanças no padrão de transmissão, que antes eram em sua maioria por contato direto com animais e, agora os relatos são de transmissão humano-humano, através do contato com feridas infecciosas e fluidos corporais. Tendo em sua maioria, ocorrido na população HSH, apresentando comumente lesões na região genital, mostrando um padrão diferente dos casos típicos, em que as lesões estavam presentes na cabeça, palma das mãos e planta dos pés.
À luz do exposto, é possível inferir que a limitação do estudo está relacionada aos casos de óbitos por MPOX, nos dois artigos citados não ficou claro se a morte foi relacionada à infecção ou devido a outros agravos de saúde. Portanto, a causa primária da morte relacionada ao MPOX ainda não está clara, um aumento da vigilância e da caracterização minuciosa dos casos, será ferramenta essencial para entender a epidemiologia de doença, assim como análises histopatológicas de necropsias futuras são necessárias para elucidar o comportamento sistêmico do vírus.
REFERÊNCIAS
- ALAKUNLE, Emmanuel et al. Monkeypox virus in Nigeria: infection biology, epidemiology, and evolution. Viruses, v. 12, n. 11, p. 1257, 2020.
- WEINSTEIN, Robert A. et al. Reemergence of monkeypox: prevalence, diagnostics, and countermeasures. Clinical infectious diseases, v. 41, n. 12, p. 1765-1771, 2005.
- LIU, Jun et al. Rettrospective detetection of monkeypox virus in the testes of nonhuman primate survivors. Nature Microbiology, p. 1-7, 2022
- Brasil. Brasil confirma primeiro caso de monkeypox. Ministério da Saúde. Disponível em:https://www.gov.br/saude/ptbr/search?Searchabletext=monkeypox. Acesso em: 28 nov. 2022.
- DE BONA SARTOR, Elisiane et al. Emergência e ressurgimento de doenças infecciosas: Os desafios das fronteiras sanitárias. Visão Acadêmica, v. 23, n. 3, 2022.
- BAYER‐ GARNER, I. B. Monkeypox virus: histologic, immunohistochemical and electron‐ microscopic findings. Journal of cutaneous pathology, v. 32, n. 1, p. 28-34, 2005.
- DE DOENÇAS, Coordenadoria de Controle. MONKEYPOX-MPX.
- MARONESE, Carlo A. et al. Clinical, dermoscopic and histopathological findings in localized human monkeypox: a case from northern Italy. The British journal of dermatology, v. 187, n. 5, p. 822-823,2022.
- KHAN, Samavia; RAZI, Shazli; RAO, Babar. It’s here, monkeypox: A case report.
JAAD Case Reports, v. 28, p. 61-63, 2022 - CHALALI, Faris et al. Histological features associated with Human Monkeypox Virus Infection in 2022 outbreak in a non-endemic country.Clinical Infectious Diseases, 2022.
- RAMOS‐ RODRÍGUEZ, Daniel et al. Dermoscopy of Monkeypox:histopathology and clinical correlation. Journal of the European Academyof Dermatology and Venereology, 2022.
- STAGLES, M. J. et al. The histopathology and electron microscopy of ahuman monkeypox lesion. Transactions of the Royal Society of Tropical Medicine and Hygiene, v. 79, n. 2, p. 192-202, 1985.
- PATEL, Aatish et al. Clinical features and novel presentations of human monkeypox ina central London centre during the 2022 outbreak: descriptive case series. bmj, v. 378, 2022.
- MENEZES, Yargos Rodrigues; MIRANDA, Alexandre Braga de. Severe disseminatedclinical presentation of monkeypox virus infection in na immunosuppressed patient: first death report in Brazil. Revista da Sociedade Brasileira de Medicina Tropical, v. 55, 2022.
Capítulo de livro publicado no Livro da IV Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
doi.org/10.53934/9786599965821-25
Este trabalho foi escrito por:
Nandara Fernanda Alves Rodrigues1; Elisabete Cardiga Alves2
1Estudante do Curso de Especialização em Vigilância Laboratorial em Saúde Pública – IAL- SJRP; E-mail: [email protected]
2 Docente – CLR- IAL- SJRP X, e-mail [email protected]
Resumo: A coqueluche é uma doença infecciosa aguda do trato respiratório inferior, altamente contagiosa, causada pela bactéria B.pertussis, acomete pessoas de qualquer faixa etária, sendo o grupo de maior risco crianças menores e lactantes. A transmissão ocorre pelo contato direto com pessoa infectada por meio de gotículas de orofaringe eliminadas por espirro, tosse e fala, bem como pelo contato com objetos contaminados com secreções do doente. A doença evolui em três fases consecutivas: catarral, paroxística e de convalescença. Apesar do Programa Nacional de Imunizações, a coqueluche continua sendo um importante problema de saúde pública, devido à elevada mortalidade infantil. O objetivo deste trabalho é descrever a ocorrência de casos de coqueluche no Brasil no período de janeiro 1998 a novembro de 2022. Trata-se de uma revisão elaborada a partir de artigos científicos, livros, manuais, dissertações de mestrado e portais eletrônicos. Foram utilizadas as seguintes bases de dados: PubMed, LILACS, SciELO, MEDLINE e Google Acadêmico. O estudo realizado demonstrou que as regiões no Brasil com maior número de casos confirmados de coqueluche no intervalo de 1998 a novembro de 2022 foram respectivamente: região sudeste, região sul e a região norte. A maior ocorrência de casos de coqueluche concentrou- se na faixa etária dos menores de 1 ano de idade, principalmente menores de 6 meses. No Brasil a cobertura vacinal diminui a partir de 2018 com aumento de casos de coqueluche em indivíduos não vacinados, principalmente na faixa etária de menores de 2 meses e indivíduos com situação vacinal ignorada que ressalta a importância da vacina em crianças, adultos, gestantes, profissionais de saúde e idosos como prevenção da doença. Foi observado um decréscimo significativo da coqueluche no período 2020 a 2022 devido ao foco na assistência e prevenção da covid-19, situação que contribuiu para a diminuição do número de casos de doenças de transmissão respiratória como a coqueluche. A coqueluche ainda deve ser considerada uma doença de importância mundial em saúde pública, portanto devemos reforçar as ações de diagnóstico, prevenção e controle da doença.
Palavras-Chaves: Bordetella pertussis, coqueluche, epidemiologia, vacinação
Introdução
A coqueluche é doença infecciosa aguda do trato respiratório inferior, altamente contagiosa, causada pela bactéria Bordetella pertussis, acomete pessoas de qualquer faixa etária, sendo o grupo de maior risco crianças menores e lactantes1,2. A transmissão ocorre pelo contato direto com pessoa infectada por meio de gotículas de orofaringe eliminadas por espirro, tosse e fala, bem como pelo contato com objetos contaminados com secreções do doente3,5,7. A doença evolui em três fases consecutivas: catarral, paroxística e de convalescença4. Na fase catarral ocorrem manifestações respiratórias com sintomas leves de febre pouco intensa, mal-estar geral, coriza e tosse seca. A Fase paroxística é geralmente afebril ou com febre baixa, crise com tosse súbita, rápida e curta6. Apesar do Programa Nacional de Imunizações, a coqueluche continua sendo um importante problema de saúde pública, devido a elevada mortalidade infantil1,6. A principal forma de prevenção contra a coqueluche é a imunização. As vacinas penta (difteria, tétano, pertussis, hepatite B e Haemophilus influenza tipo b), tríplice bacteriana Difteria, Tétano e Coqueluche (DTP) e a vacina DTPa (acelular) estão disponibilizadas nos serviços de saúde do Brasil.3
O diagnóstico da coqueluche é realizado por meio de sinais e sintomas clínicos, Rx de tórax e diagnóstico laboratorial. O diagnóstico laboratorial é realizado com exames inespecífico como o hemograma. Os exames específicos utilizados são os métodos microbiológicos, métodos imunológicos e moleculares.8
Foi preconizado pelo Ministério da Saúde, Azitromicina 250 mg como droga de primeira escolha para o tratamento da coqueluche que apresenta maior efetividade quando administrada no início dos sintomas. O quadro inicial é inespecífico e pode causar atraso no diagnóstico e início do tratamento, levando à evolução da doença para quadros mais graves, portanto a prevenção da coqueluche por meio da imunização é vital, especialmente para os neonatos2.
Muitos países enfrentam desafios significativos de vigilância, principalmente no diagnóstico clínico, confirmação laboratorial, falta de notificações epidemiológicas incompletas que podem resultar em subnotificação da doença9.
Para reduzir o número de casos em lactentes, a imunização materna foi introduzida em muitos países durante a última década. Os dados demostram que a imunização reduz o número de casos e o número de mortes causadas pela doença9.
Avaliações nacionais indicam aumento do risco de doenças imunopreveníveis devido à queda de vacinação, principalmente em crianças menores de um ano. As ações de educação em saúde são fundamentais para a prevenção da coqueluche, portanto informar a população sobre os aspectos epidemiológicos da coqueluche, vacinação como principal medida de prevenção e controle da doença, ressaltam a importância do levantamento bibliográfico do referido estudo para o conhecimento do perfil epidemiológico da Coqueluche no Brasil10.
Material e Métodos
Este trabalho foi elaborado por meio de uma revisão bibliográfica baseando-se, no perfil epidemiológico da coqueluche. Definiu-se trabalhar com as seguintes bases de dado do PubMed, LILACS, MEDLINE, Google Acadêmico e Scielo, manuais contidos em sites do Ministério da Saúde, dissertações de mestrado e artigos de revista. Realizou-se buscas avançadas selecionando-se palavras-chave, Bordetella pertussis, coqueluche, epidemiologia e vacinação.
Resultado e Discussão
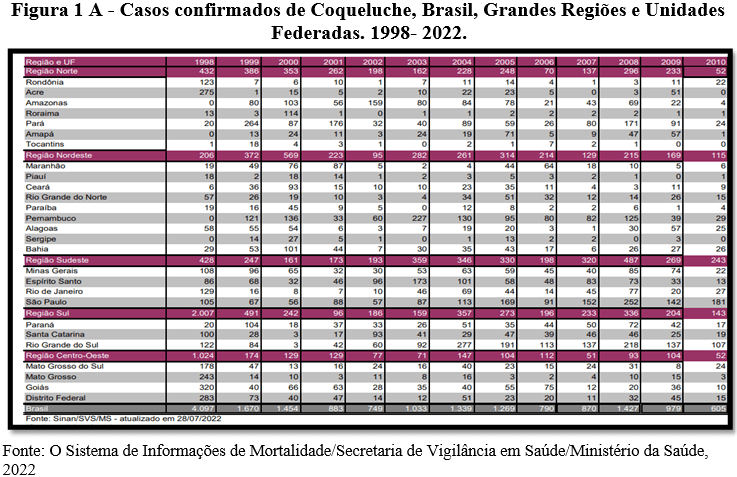
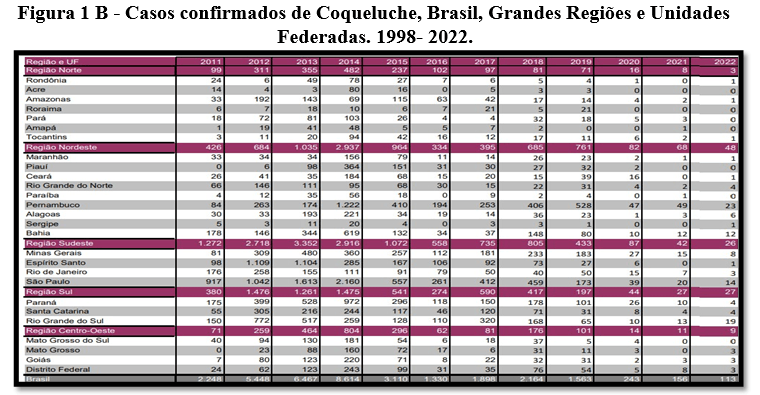
De acordo com a figura 1 A e 1 B, as regiões no Brasil com maior número de casos confirmados de coqueluche no período de 1998 a novembro de 2022 foram respectivamente: região sudeste, região sul e a região norte.
No Brasil, no intervalo de 1998 a novembro de 2022 o período com maior número de casos foi 2014. De acordo com o boletim epidemiológico do ministério da saúde referente ao período de 2010 a 2014 o aumento do número de casos de coqueluche pode ter ocorrido devido à ciclicidade da doença, baixas coberturas vacinais, melhoria no diagnóstico laboratorial e variação de cepas entre as regiões.
Houve diminuição expressiva do número de casos de coqueluche no período de 2020 a 2022. De acordo com o boletim epidemiológico do ministério da saúde referente ao período de 2020 a 2022 a queda expressiva de casos se deve-se ao foco na assistência e prevenção ao covid 19 que contribuíram para a redução doenças de transmissão respiratória, como é o caso da coqueluche.
Fonte: O Sistema de Informações de Mortalidade/Secretaria de Vigilância em Saúde/Ministério da Saúde, 2022
A figura 2 demonstra que no período de 1990 a julho de 2022 o coeficiente de incidência da coqueluche foi maior em 1990 com queda no período de 1991 a 1996 e aumento em 1997. A cobertura vacinal diminui a partir de 2018, de acordo com o boletim epidemiológico do ministério da saúde de 2018 a 2022 se deve-se a recusa as vacinas, devido a fatores econômicos, sociais, culturais, disponibilidade do imunobiológico, acessibilidade geográfica, capacidade de compreensão e acesso às informações em saúde resultando em não proteção coletiva promovendo a circulação de doenças imunopreveníveis.
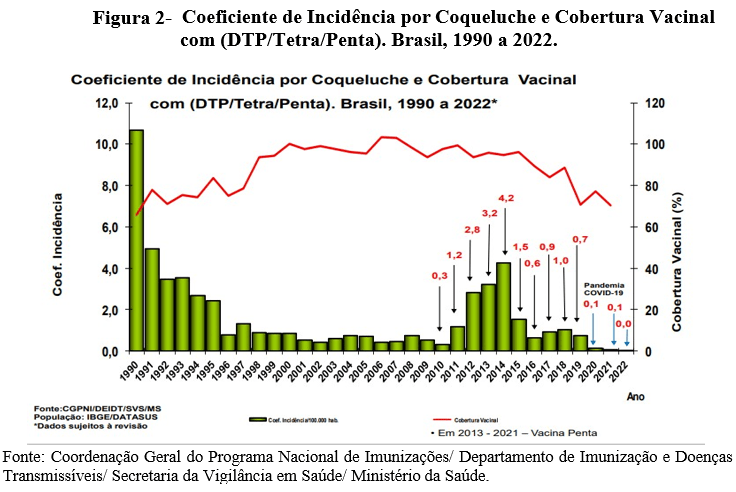
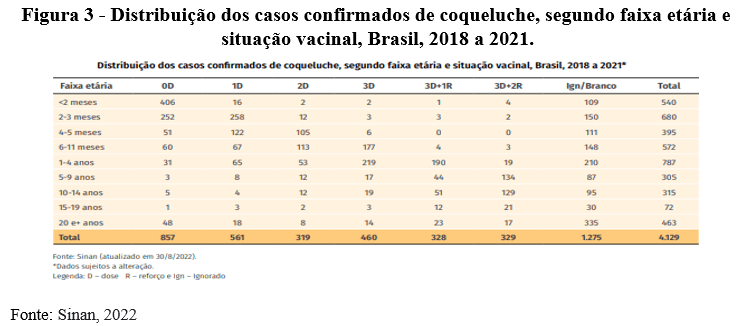
De acordo com a figura 3 a cobertura vacinal diminui a partir de 2018 com aumento de casos de coqueluche em indivíduos não vacinados, principalmente na faixa etária de menores de 2 meses e indivíduos com situação vacinal ignorada. Portanto esses dados ressaltam a importância da vacina como prevenção á coqueluche.
Conclusão
As regiões no Brasil com maior número de casos confirmados de coqueluche no período de 1998 a novembro de 2022 foram respectivamente: região sudeste, região sul e a região norte.
A maior ocorrência de casos de coqueluche concentrou-se na faixa etária dos menores de 1 ano de idade, principalmente menores de 6 meses.
No Brasil a cobertura vacinal diminui a partir de 2018 com aumento de casos de coqueluche em indivíduos não vacinados, principalmente na faixa etária de menores de 2 meses e indivíduos com situação vacinal ignorada que ressaltam a importância da vacina em crianças, adultos, gestantes, profissionais de saúde e idosos como prevenção da doença.
Foi observado um decréscimo significativo da coqueluche no período 2020 a 2022 devido ao foco na assistência e prevenção da covid-19, situação que contribuiu para a diminuição do número de casos de doenças de transmissão respiratória.
A coqueluche ainda deve ser considerada uma doença de importância mundial em saúde pública, portanto devemos reforçar as ações de diagnóstico, prevenção e controle da doença.
Referências Bibliográficas
1- BRASIL. Ministério da Saúde. Secretaria de Vigilância em Saúde. Boletim Epidemiológico: Coqueluche no Brasil: análise da situação epidemiológica de 2010 a 2014,
v. 46, n. 39. 2015. Disponível em: https://www.gov.br/saude/pt-br/assuntos/saude-de-a-a- z/c/coqueluche/arquivos/boletim-epidemiologico-da-coqueluche-brasil-2010-a-2014.pdf. Acesso em: 03 out. 2022.
2- MACHADO, L. Z.; MARCON, C. E. M. Incidência de coqueluche em crianças menores de 1 ano e relação com a vacinação materna no Brasil, 2008 a 2018. Epidemiologia e Serviços de Saúde: revista do SUS (RESS), Brasília, v. 31, n. 1, 2022. https://doi.org/10.1590/S1679-49742022000100029. Disponível em: https://www.scielo.br/j/ress/a/QwfrBxCKsLTyRVYgzKbmfGF/?lang=pt. Acesso em: 03 out. 2022.
3- BRASIL. Ministério da Saúde. Secretaria de Vigilância em Saúde. Informe epidemiológico da coqueluche Brasil 2018 a 2019. Disponível em: https://www.gov.br/saude/pt-br/assuntos/saude-de-a-a-z/c/coqueluche/arquivos/informe – epidemiologico-da-coqueluche-brasil-2018-a-2019.pdf. Acesso em: 04 nov. 2022.
4- BRASIL. Ministério da Saúde. Secretaria de Vigilância em Saúde. Coordenação-Geral de Desenvolvimento da Epidemiologia em Serviços. Guia de Vigilância em Saúde: volume único [recurso eletrônico]/Ministério da Saúde, Secretaria de Vigilância em Saúde, Coordenação-Geral de Desenvolvimento da Epidemiologia em Serviços. – 3ª. ed. – Brasília: Ministério da Saúde, 2019. 740 p. disponível em: https://bvsms.saude.gov.br/bvs/publicacoes/guia_vigilancia_saude_3ed.pdf. Acesso em: 04 nov. 2022.
5- SOARES, J. S.; BRITO, L. C. F.; COÊLHO, M.B.; COSTA, R. K. E.; OLIVEIRA, E. H.
Análise do perfil epidemiológico dos casos de coqueluche no estado do Piauí de 2013 a 2018. Research, Society and Development, v. 10, n. 2, e39810211354, 2021. DOI: http://dx.doi.org/10.33448/rsd-v10i2.11354. Disponível em: https://rsdjournal.org/ index.php/rsd/article/view/11354. Acesso em: 05 out. 2022.
6- Governo Do Estado De São Paulo. Secretaria De Estado Da Saúde. Coordenadoria De Controle De Doenças. Centro De Vigilância Epidemiológica “Prof Alexandre Vranjac”/Divisão De Doenças De Transmissão Respiratória e Divisão de Imunização. Informe Técnico. Situação Epidemiológica Da Coqueluche, Cenário Global, novembro de 2011, São Paulo, Brasil. Disponível em: https://www.cvpvacinas.com.br/pdf/IF11_ COQUELUCHE.pdf. Acesso em: 05 out. 2022.
7- FIGUEIREDO, B. Q.; ANDRADE, A. F.; OLIVEIRA, I. P.; OLIVEIRA, J. P.G.; FERREIRA, K. S.; SILVA, L. S.; SANTOS, M. C. T.; MARRA, M. S.; VALE, S.
Diminuição de casos notificados de coqueluche em crianças brasileiras: reflexo do distanciamento social e suspensão das aulas devido à pandemia de Covid-19. Research, Society and Development. v. 10, n. 11, p. e230101119631, 2021. DOI: http://dx.doi.org/10.33448/rsd-v10i11.19631. Disponível em: https://rsdjournal.org/ index.php/rsd/article/view/19631. Acesso em: 06 out. 2022.
8- HIDALGO, Neuma TR et al. Manual de vigilância epidemiológica; coqueluche: normas e instruções 2000. 2001. Disponível em: https://www.saude.sp.gov.br/ resources/cve-centro-de-vigilancia-epidemiologica/areas-de-vigilancia/doencas-de- transmissao-respiratoria/doc/2001/manu_coque.pdf. Acesso em 06 out.2022.
9- HOZBOR, D.; ULLOA-GUTIERREZ, R.; MARINO, C. et al. Pertussis in Latin America: Recent epidemiological data presented at the 2017 Global Pertussis Initiative meeting. Vaccine, v. 37, n. 36, p. 5414-5421. doi: 10.1016/j.vaccine.2019.07.007. Disponível em: https://www.sciencedirect.com/science/article/pii/ S0264410X19308850?via%3Dihub.Acesso em: 04 nov. 2022.
10- Divisão de Imunização. Centro de Vigilância Epidemiológica – “Prof Alexandre Vranjac”. Coordenadoria de Controle de Doenças. Secretaria de Estado da Saúde. São Paulo, Brasil. Coberturas Vacinais de Rotina no Estado de São Paulo – 2013 a 2017. BEPA.
Boletim Epidemiológico Paulista, São Paulo, v. 15, n. 177, p.21-25, 2018. Disponível em: https://periodicos.saude.sp.gov.br/BEPA182/article/view/37943. Acesso em 09 out.2022.
11- BRASIL. Ministério da Saúde. Secretaria de Vigilância em Saúde. Situação epidemiológica da coqueluche no Brasil, 2018 a 2021 e semanas epidemiológicas de 1 a 32 de 2022. Boletim Epidemiológico, v. 53, n. 40, p. 31-46, 2022. Disponível em: https://www.gov.br/saude/pt-br/centrais-de-conteudo/publicacoes/boletins/ epidemiologicos/edicoes/2022/boletim-epidemiologico-vol-53-no40/view. Acesso em: 04 nov. 2022.
Capítulo de livro publicado no Livro da IV Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
doi.org/10.53934/9786599965821-24
Este trabalho foi escrito por:
Isabela Fernandes Rodrigues1; Carlos Roberto Prudêncio2.
1Estudante do Curso de Especialização em Vigilância Laboratorial em Saúde Pública – Centro de Imunologia – Instituto Adolfo Lutz; E-mail: [email protected]
2Docente/pesquisador do Centro de Imunologia – Instituto Adolfo Lutz
Resumo: A pandemia ocasionada pelo SARS-CoV-2 levou a comunidade científica a uma rápida busca por antígenos virais capazes de induzirem uma resposta imunológica eficiente para a produção de vacinas, bem como na aplicação em testes diagnósticos sorológicos. A obtenção e utilização de proteínas recombinantes por meio de técnicas biotecnológicas, tem se mostrado muito eficiente em razão do seu custo-benefício e da sua diversidade de aplicação, contribuindo para o avanço científico. Este trabalho teve como objetivo otimizar o processo de expressão de uma proteína recombinante fusionada (RBD+N) do SARS-CoV- 2 em bactérias E. coli, avaliando as melhores condições para a obtenção da proteína pura, que futuramente servirá para testes imunológicos in vitro e in vivo. O vetor de expressão pH6HTC His6 HaloTag® T7 Vector (Promega Corporation, USA) foi utilizado para a expressão da sequência da proteína fusionada. As cepas de E. coli BL21 (DE3) pLysS e Rosetta foram transformadas com o plasmídeo, para a realização da expressão. A extração da proteína foi realizada utilizando os tampões Bug Buster® e Sarkosyl e submetidas à purificação por cromatografia de afinidade. As amostras foram analisadas por gel de SDS- PAGE e Western Blotting. A cepa E. coli Rosetta no tempo de 4 horas apresentou maior rendimento de proteína expressa, enquanto a E. coli BL21 no tempo de 4 horas foi a que menos expressou. Ambas as cepas no tempo de 18 horas expressaram concentrações próximas. Ao avaliar o perfil de purificação, a BL21 apresentou baixa formação de corpo de inclusão possibilitando a liberação da proteína no primeiro estágio de extração proteica. Os resultados do Western blotting sugerem maiores investigações de possíveis problemas da sequência gênica no vetor de expressão que possam ter influenciado na purificação devido ao não reconhecimento da proteína pelo anticorpo anti-His tag.
Palavras–chave: E. coli, proteína recombinante fusionada, SARS-CoV-2.
INTRODUÇÃO
Em dezembro de 2019, o aparecimento de diversos casos de pneumonia na cidade de Wuhan, na China, acendeu um alerta para que o governo chinês buscasse a fonte do surto e comunicasse aos órgãos internacionais de vigilância. Posteriormente, em fevereiro, taxonomistas classificaram o vírus em SARS-CoV-2 (do inglês, severe acute respiratory syndrome coronavirus 2) e a OMS nomeou a nova doença como COVID-19 (Coronavirus disease 2019). Em meados de março já havia a circulação do vírus por diversos países ao redor do mundo, sendo caracterizada como pandemia, impactando fortemente na saúde pública global (SUN et al., 2020).
O vírus possui tropismo pelo trato respiratório, principalmente nas células pulmonares, causando uma série de complicações ao hospedeiro (HARRISON, LIN, WANG, 2020). Os casos variam de leves, moderados e graves. Entre os sintomas comumente relatados estão a tosse, febre e dor de garganta. Em casos mais graves, os pacientes podem apresentar dor no peito, falta de ar, podendo evoluir para uma síndrome respiratória aguda grave, necessitando de internação hospitalar (WHO, 2022).
Com o rápido aumento dos números de casos mundialmente, houve a necessidade da busca por marcadores virais capazes de auxiliarem na terapêutica e no diagnóstico da nova patologia (YÜCE, FILIZTEKIN, ÖZKAYA, 2021). A procura por sítios ativos e imunogênicos para produção de vacinas foi o foco de diversos grupos de pesquisa na tentativa de desenvolver antígenos recombinantes capazes de induzir a resposta imunológica humoral e/ou celular contra o novo vírus (AWADASSEID et al., 2021).
Este trabalho teve como objetivo otimizar o processo de expressão de uma proteína recombinante fusionada (RBD+N) do SARS-CoV-2 em bactérias Escherichia coli, avaliando as melhores condições para a obtenção da proteína pura, que futuramente servirá para testes imunológicos in vitro e in vivo.
MATERIAL E MÉTODOS
O vetor de expressão pH6HTC His6 HaloTag® T7 Vector (Promega Corporation, USA) foi utilizado para a expressão da sequência da proteína recombinante resultante da fusão das proteínas RBD (domínio de ligação ao receptor, uma subunidade de proteína Spike, de superfície do vírus) e N (proteína do nucleocapsídeo). As cepas E. coli BL21 (DE3) pLysS e Rosetta foram transformadas com o plasmídeo, para a realização da expressão. Foi realizado inicialmente um teste de indução após atingirem OD600nm 0,6 em 5 mL de cultura com concentração 0,4 mM de IPTG, para avaliar as características de cada cepa e posteriormente a expressão em 100 mL de cultura a 0,5 mM de IPTG em dois tempos diferentes (4 e 18 horas). A extração da proteína foi realizada utilizando os tampões Bug Buster® (Novagen®, USA), juntamente com a adição de lisozima (Invitrogen, USA; 1000 U/mL) e Benzonase® Nuclease (Sigma-Aldrich®, USA; 25 U/mL) e Sarkosyl (1,5% Sarkosyl, 25 mM Trietanolamina, 1mM de EDTA) e submetidas à purificação por cromatografia de afinidade. As amostras foram analisadas por SDS-PAGE e Western blotting.
RESULTADOS E DISCUSSÃO
A utilização de bactérias para a produção de proteínas heterólogas tem sido amplamente aplicada no ramo da biotecnologia. Muitas proteínas de interesse comercial têm-se utilizado esse modelo de vetor para estudos estruturais e funcionais devido ao seu baixo custo, fácil manipulação, rápido crescimento e alto rendimento (ROSANO, CECCARELLI, 2014). A espécie E. coli é a principal escolha de muitos pesquisadores devido a sua simplicidade genômica passível de transformação com DNA exógeno, ciclo de divisão celular entre 20 a 30 minutos em condições de crescimento favoráveis, além de possuir sua fisiologia bem caracterizada. (FRANCIS, PAGE, 2010).
Com os avanços da engenharia genética, a E. coli passou por diversas modificações a fim de melhorar seu desempenho na produção de insumos biotecnológicos, originando diversas cepas. Neste trabalho, a expressão heteróloga da proteína fusionada (RBD+N) foi avaliada nas cepas BL21(DE3) que possui uma cópia da RNA polimerase do fago T7 e possibilita a transcrição de sequências gênicas controladas pelo promotor T7 lac UV5(ROSANO, MORALES, CECCARELLI, 2019; RESENDE, 2015), e na cepa Rosetta que é recomendada para proteínas que contenham códons raros.
Dado tal contexto, as bactérias foram transformadas com o plasmídeo contendo a sequência da proteína recombinante fusionada nessas duas cepas. Para um teste inicial de expressão, foi realizada a indução durante um período de 3 horas, com uma concentração de IPTG de 0,4 mM. Durante o teste, foi retirada uma alíquota pré-indução (T0) e pós-indução a cada uma hora até completar 3 horas (T1, T2 e T3). As amostras foram submetidas à SDS- PAGE e o resultado está demonstrado na Figura 1. Após análise, foi possível observar que a cepa Rosetta forneceu um gel mais límpido em relação a BL21, tendo uma menor expressão de proteínas basais.
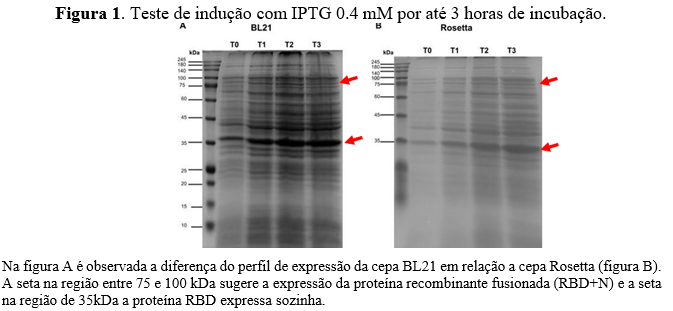
Para dar continuidade aos estudos, a expressão da proteína foi feita em uma escala maior, e decidiu-se alterar a concentração do indutor para 0,5 mM, que se baseou em estudos recém-publicados por Castro e colaboradores (2022), que trabalharam também com uma proteína fusionada (RBD+N), utilizando a concentração de 0,5 mM do indutor durante 18 horas. Portanto, as cepas foram induzidas por 4 e 18 horas de incubação a 37º C. Após a extração da proteína, utilizando dois tipos de detergentes para lisar a célula bacteriana, Bug Buster® e Sarkosyl, notou-se que os sobrenadantes obtidos dos cultivos de 18 horas apresentaram-se um aspecto viscoso, portando, essas amostras foram tratadas com DNAse (5 µg/mL), que após o tratamento ficaram fluídas, e essa característica auxilia na purificação por cromatografia de afinidade.
O vetor de expressão pH6HTC His6 HaloTag® T7 possui uma sequência de 6 resíduos de histidinas que são adicionadas na porção C-terminal da proteína alvo. Essa fusão de aminoácidos é responsável por auxiliar na purificação, através de ligações reversíveis ao níquel presente na coluna cromatográfica, permitindo que apenas as proteínas com essa cauda se liguem com maior afinidade, separando-as das demais produzidas (RESENDE, 2015). A reversão dessas ligações é realizada através da adição de imidazol no tampão de eluição, que por competição, se liga ao níquel liberando a proteína que estava imobilizada. A partir desta técnica, espera-se uma obtenção mais pura e sem contaminantes em uma única etapa (BORNHORST, FALKE, 2000). Para a purificação foi utilizado o sobrenadante após a lise celular com o tampão Sarkosyl, diluído no tampão de equilíbrio A (50 mM de Fosfato de sódio, 500 mM de Cloreto de sódio). A eluição da amostra ocorreu utilizando um gradiente de concentração de 5% a 100% do tampão B (50 mM de Fosfato de sódio, 500mM de Cloreto de sódio, 500 mM de Imidazol).
Ao analisar o perfil de purificação, as amostras da cepa Rosetta 4 e 18 horas e a BL21 18 horas apresentaram perfil de eluição próximas, entre 54-58% de imidazol (~ 280 mM). Entretanto, a amostra de BL21 de 4 horas apresentou eluição em 100% da concentração de imidazol (500 mM) presente no tampão (Figura 2). Logo, uma nova expressão dessa amostra foi realizada para observar se é perfil da cepa ou se havia algum erro durante o processo. Após a segunda purificação, observou-se que o perfil de eluição se manteve o mesmo (Figura 3) e dado tal cenário, surgiu a hipótese de que as proteínas estariam no sobrenadante do Bug Buster®, e que no tempo de 4 horas de indução nessa cepa, a formação de corpos de inclusão é baixa, sendo as proteínas liberadas no primeiro passo de extração.
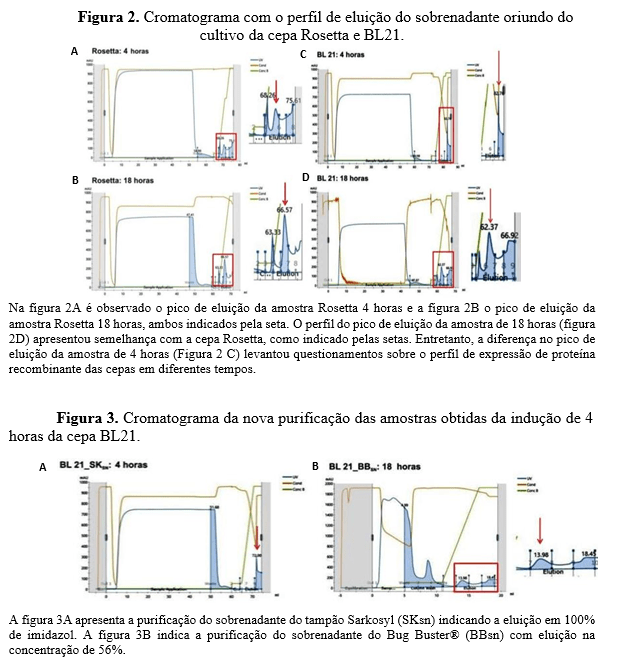
Para uma análise quantitativa das proteínas obtidas nas amostras, foi realizada a concentração das amostras para quantificação utilizando um kit comercial ((Pierce™ Rapid Gold BCA Protein Assay Kit – Thermo Fisher Scientific, USA). Os resultados da quantificação estão apresentados na Tabela 1. O eluido da purificação obtidos do cultivo da cepa Rosetta no tempo de 4 horas apresentou maior quantidade de proteína expressa dentre as amostras, enquanto os eluentes da BL21 4 horas produziu proteínas em menor concentração. As amostras de 18 horas apresentaram concentrações próximas.
Tabela 1. Quantificação da proteína expressa.

Ao final de todo o processo, as amostras foram submetidas à análise de SDS-PAGE e Western blotting. Os resultados da primeira análise revelaram que a amostras provenientes da cepa Rosetta do tempo de 4 horas apresentou uma banda de maior densidade em tempo de 18 horas (Fig. 4A), corroborando com os resultados da quantificação proteica. Observou- se também bandas entre 75 e 100 kDa, que sugere ser a proteína fusionada; porém notou-se uma banda da região de 35 kDa, característico da proteína RBD indicando que a purificação não foi eficiente, pois teoricamente a cauda de histidina estaria presente na proteína quimérica. Em relação à cepa BL21 (Fig. 4B) ocorreu a situação inversa, no tempo de 18 horas a bactéria produziu mais proteína em relação ao tempo de 4 horas. Também foi realizado um gel para analisar a segunda purificação do tempo de 4 horas da BL21 para comparar a eluição da proteína nos diferentes sobrenadantes (Fig. 4C). A partir desses resultados, concluiu-se que a maior concentração de eluição da proteína estava no sobrenadante do Bug Buster® e bem pouca no Sarkosyl. Os resultados do SDS-PAGE também indicaram a ineficiência da purificação, visto que apenas proteínas que possuem cauda de histidina deveriam ser eluída, apresentando-se em uma banda única no gel.
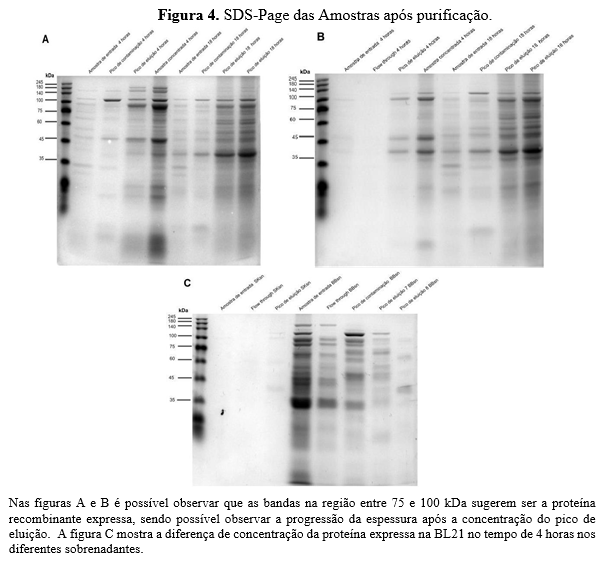
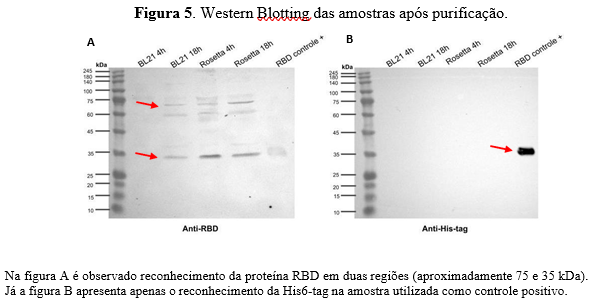
Em ambos os géis, ficou evidente uma banda espessa próxima a marcação de 35 kDa, que se assemelha ao perfil da proteína RBD sozinha e ainda há necessidade de otimizar o método de purificação. Para confirmar a expressão da proteína fusionada RBD+N, foi realizado um Western Blotting com imunorreação com anticorpo primário de coelho anti- His6-tag (Sigma-Aldrich®, USA; diluição 1:1000) e soro de coelho anti-RBD (produzido in-house pelo grupo de pesquisa). A membrana tratada com soro anti-RBD, apresentou marcação em duas regiões (~60 kDa e 35 kDa) (Figura 5A), indicando que além da proteína inteira, a bactéria possivelmente também expressou somente a porção da RBD. A membrana tratada com anticorpo anti-His6-tag para analisar o peso molecular e confirmar a expressão da proteína fusionada não apresentou imunorreação para o reconhecimento da cauda de histidina nas amostras pelo anticorpo, reconhecendo apenas o RBD com cauda de histidina (produzido pelo grupo de pesquisa) que foi utilizado como controle positivo (Figura 5B). Desta forma, supõe-se que a cauda da Histidina não está sendo produzida completa, pois está na sequência do vetor e não na sequência gênica da proteína, podendo haver algum problema na fusão entre as sequências. Fato que fica evidente durante a purificação, pois observou-se que um pico de eluição na concentração de aproximadamente 300 mM, sugerindo que apenas proteínas com poli-histidinas (mais de 3 resíduos de aminoácidos) ficariam mobilizadas nas colunas e eluiriam nessa concentração.
CONCLUSÕES
A utilização de duas cepas de E. coli, BL21(DE3) pLysS e Rosetta, permitiu avaliar o perfil em diferentes condições de tempo, mantendo a concentração do indutor e temperatura. A cepa Rosetta no tempo de 4 horas produziu maior quantidade de proteína recombinante fusionada (RBD+N), enquanto a cepa BL21 no mesmo tempo produziu menor quantidade.
Ao avaliar o perfil de purificação, a BL21 de 4 horas sugere a baixa formação de corpos de inclusão possibilitando a solubilização da proteína no citosol bacteriano e sendo liberado na primeira fase de lise celular.
Os resultados do Western Blotting sugerem a necessidade de maiores investigações quanto a possíveis interferentes na sequência gênica no vetor durante a expressão, para continuar otimizando a produção da proteína recombinante fusionada (RBD+N) a fim de obtê-la pura e sem contaminantes.
AGRADECIMENTOS
Gostaria de agradecer a todos os colaboradores do laboratório de Imunotecnologia do Instituto Adolfo Lutz que me auxiliaram durante o processo para a realização deste trabalho.
REFERÊNCIAS
AWADASSEID, Annoor et al. Current Advances in the development of SARS-CoV-2 vaccines. International Journal Of Biological Sciences, v. 17, n. 1, p. 8, 2021.
BORNHORST, Joshua A.; FALKE, Joseph J. [16] Purification of proteins using polyhistidine affinity tags. In: Methods in enzymology. Academic Press, 2000. p. 245-254.
CASTRO, Julia T. et al. Promotion of neutralizing antibody-independent immunity to wild- type and SARS-CoV-2 variants of concern using an RBD-Nucleocapsid fusion protein. Nature communications, v. 13, n. 1, p. 1-16, 2022.
FRANCIS, Dana M; PAGE, Rebecca. Strategies to optimize protein expression in E. coli. Current protocols in protein science, v. 61, n. 1, p. 5.24. 1-5.24. 29, 2010.
HARRISON, Andrew G.; LIN, Tao; WANG, Penghua. Mechanisms Of SARS-CoV-2 transmission and pathogenesis. Trends in immunology, v. 41, n. 12, p. 1100-1115, 2020.
RESENDE, Rodrigo R. Biotecnologia aplicada à saúde fundamentos e aplicações: Vol 2. Editora Blucher, 2015.
ROSANO, Germán L; CECCARELLI, Eduardo A. Recombinant protein expression in Escherichia coli: advances and challenges. Frontiers in microbiology, v. 5, p. 172, 2014.
ROSANO, Germán L; MORALES, Enrique S; CECCARELLI, Eduardo A. New tools for recombinant protein production in Escherichia coli: A 5‐year update. Protein science, v. 28, n. 8, p. 1412-1422, 2019.
SUN, Pengfei et al. Understanding Of COVID‐19 based on current evidence. Journal Of medical virology, v. 92, n. 6, p. 548-551, 2020.
TEGEL, Hanna et al. Increased levels of recombinant human proteins with the Escherichia coli strain Rosetta (DE3). Protein expression and purification, v. 69, n. 2, p. 159-167, 2010.
WANG, Mei-Yue et al. SARS-CoV-2: structure, biology, and structure-based therapeutics development. Frontiers in cellular and infection microbiology, v. 10, p. 587269, 2020.
WHO. World health organization, 2022. Coronavírus Disease (COVID-19) Symptoms. Disponível em: <https://www.who.int/health-topics/coronavirus#tab=tab_3>. Acesso em: 06 out. 2022.
WHO. World health organization, 2022. Coronavírus Disease (COVID-19) Prevention. Disponível em: <https://www.who.int/health-topics/coronavirus#tab=tab_2>. Acesso em: 06 out. 2022.
YANG, Haitao; RAO, Zihe. Structural biology Of SARS-CoV-2 and implications for therapeutic development. Nature Reviews Microbiology, v. 19, n. 11, p. 685-700, 2021.
YÜCE, Meral; FILIZTEKIN, Elif; ÖZKAYA, KorinGasia. COVID-19 diagnosis — A review of current methods. Biosensorsand Bioelectronics, v. 172, p. 112752, 2021.
Capítulo de livro publicado no Livro da IV Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
doi.org/10.53934/9786599965821-23
Este trabalho foi escrito por:
Tamires Dantas Campos1; Vanessa Nieri Zanelatto2
1Estudante do Curso de Vigilância Laboratorial e Epidemiologia – NCMO – CEFOR; E-mail: [email protected]
2Docente/pesquisadora do Núcleo de Coleção de Micro-organismos – NCMO; E-mail: [email protected]
Resumo: As coleções de micro-organismos atuam armazenando, conservando e fornecendo linhagens de importância para pesquisas em diversas áreas da saúde, meio ambiente, indústrias e controle de qualidade. Também faz parte do trabalho das coleções caracterizar e autenticar as linhagens, conferindo maior confiança e especificidade ao material fornecido. A Coleção de Culturas do Instituto Adolfo Lutz armazena e organiza cepas de grande importância para a saúde pública e pesquisas. Para realizar a autenticação do material depositado, são utilizadas técnicas de identificação fenotípica e métodos de biologia molecular. O presente trabalho teve como objetivo caracterizar e autenticar linhagens de bactérias Gram-positivas armazenadas pelo método de liofilização no Núcleo de Coleção de Micro-organismos (NCMO) do Instituto Adolfo Lutz por meio de identificações fenotípicas, espectrometria de massas (MALDI-TOF) e sequenciamento genético. Foram selecionadas linhagens dos gêneros Corynebacterium, Staphylococcus, Streptococcus e Rhodococcus, que passaram por identificação. Foi possível diferenciar as espécies dos gêneros Corynebacterium por todos os métodos usados, confirmando resultados através de sequenciamento Sanger, não conseguindo distinguir apenas seus biovares. Todas as espécies de Staphylococcus foram estabelecidas pelas técnicas tradicionais de identificação. As linhagens de Streptococcus foram classificadas como pertencentes ao grupo viridans pelas técnicas tradicionais, sendo sua espécie determinada por espectrometria de massas e sequenciamento. As linhagens de Rhodococcus equi foram identificadas por espectrometria de massas. Diante dos resultados obtidos, foi possível concluir que a metodologia do MALDI-TOF foi a mais eficiente na autenticação das linhagens estudadas e que os métodos utilizados no NCMO são eficazes na preservação das características originais dos micro- organismos.
Palavras-chave: Coleções de culturas, Corynebacterium, Rhodococcus, Staphylococcus, Streptococcus.
INTRODUÇÃO
As coleções de cultura são Centros de Recursos Biológicos (BRC), que armazenam materiais biológicos autenticados, identificados com alta precisão, servindo como repositórios da biodiversidade e centros de recursos microbianos. Estes centros possuem experiência em preservar, cultivar, caracterizar e distribuir linhagens autenticadas, fornecendo recursos para ensaios de controle de qualidade, inovação e pesquisas (JANSSENS, D. et al, 2010; BECKER, P. et al, 2019).
O Núcleo de Coleção de Micro-organismos (NCMO) do Instituto Adolfo Lutz, membro da Word Federal for Culture Collections (WFCC) é classificado como uma coleção de serviços, com acervo abrangente, com curadoria profissional e importante papel na caracterização, manutenção e distribuição de micro-organismos, desde 1940 (ANDRADE, T. S., 2008).
O acervo do NCMO é constituído por aproximadamente 10 mil linhagens, desta forma, para mantê-lo atualizado são necessárias revisões constantes, sendo necessário emprego de técnicas modernas para contribuir com este processo. Assim, o presente trabalho teve como objetivo caracterizar e autenticar bactérias Gram-positivas, presentes na coleção do Instituto Adolfo Lutz, utilizando técnicas tradicionais e modernas, tornando-as disponíveis para fornecimento. Para isso foram selecionadas linhagens dos gêneros Corynebacterium, Rhodococcus, Staphylococcus e Streptococcus, micro-organismos considerados de importância para saúde pública e relacionados a infecções em pacientes imunocomprometidos.
MATERIAL E MÉTODOS
Para este trabalho foram selecionados micro-organismos liofilizados depositados no acervo da coleção, a partir do acrônimo IAL, Tabela 1.

Para realizar a autenticação foram utilizados os seguintes métodos:
- Análise macroscópica, analisando a uniformidade do crescimento em meio de cultura, e microscópica, usando coloração de Gram e de Albert-Laybourn (identificando grânulos metacromáticos em bacilos corineformes) e microscópio óptico com aumento de 1000X;
- Identificação fenotípica por meio de provas bioquímicas, diferenciando espécies a partir das transformações químicas num determinado substrato, e antibiograma, avaliando a suscetibilidade a antimicrobianos, sendo necessário, para esta técnica, a produção de suspensão bacteriana com turbidez equivalente a escala 0,5 de McFarland e o uso de discos de Novobiocina (5 µg) ou Optoquina (5 mcg);
- MALDI-TOF (Matrix Assisted Laser Desorption/Ionization – Time of Flight Mass Spectrometry) identificando a presença de proteínas específicas através da dessorção de moléculas a laser, sendo estas aspiradas a vácuo até um detector, que avalia seu tempo de chegada, produzindo um gráfico, com picos proteicos, analisado e interpretado de forma computadorizada. Para identificação de bactérias Gram-positivas foi realizada extração proteica em tubo (usando etanol 100%, ácido fórmico 70% e acetonitrila) e extração direta (usando ácido fórmico 95%), comparando os resultados obtidos com os presentes na biblioteca padrão do equipamento Bruker, sendo os que apresentaram score entre 2,00 – 3,00, considerados de alta confiança.
- Sequenciamento genético pelo método Sanger analisando a sequência do gene codificador do RNA ribossomal da porção 16S, onde existem regiões de consenso, variabilidade e hipervariabilidade, que permitem identificar diferentes espécies. Para este trabalho foi realizada a extração de material genético; amplificação utilizando os primers 27F/518R. Confirmada amplificação e integridade, foi realizada a purificação dos produtos com o MEGAquick-spin™ Total Fragment DNA Purification, quantificação em nanoespectrofotômetro, seguindo com a reação de sequenciamento usando o kit BigDye® Terminator v3.1 Cycle Sequencing e encaminhando a placa ao Laboratório Estratégico para ser analisada no equipamento 3500 Genetic Analyzer (Thermo Fisher).
RESULTADOS E DISCUSSÃO
Análises Macroscópica e Microscópica
- Corynebacterium spp
Em macroscopia foram realizadas as seguintes observações:
- IAL 93 (C. diphtheriae) – crescimento em 48 horas, com coloração creme, aspecto cremoso, colônias pequenas, bem delimitadas e bordas uniformes;
- IAL 99 (C. diphtheriae bv. gravis) e IAL 101 (C. diphtheriae bv. mitis) crescimento em 48 horas com coloração creme, aspecto cremoso, colônias medianas, bem delimitadas e bordas uniformes;
- IAL 104 (C. pseudodiphtheriticum) – crescimento em 48 horas, com coloração creme, aspecto cremoso, colônias medianas, bordas pregueadas e centro branco;
- IAL 1817 (C. glutamicum) – crescimento em 24 horas, com coloração creme, aspecto cremoso, colônias pequenas, bordas bem delimitadas e uniformes.
Em microscopia, as linhagens IAL 93, 99, 101 foram visualizados bacilos roxos, paliçados, com grânulos metacromáticos, confirmados pela coloração de Albert-Laybourn. As linhagens IAL 104 e 1817 apresentaram bacilos de coloração roxa, mais curtos que os anteriores, paliçados, sem grânulos metacromáticos, confirmado pela coloração de Albert- Laybourn. Desta forma, pode-se dizer que todas as linhagens estavam de acordo com a literatura de referência (WITHMAN, W. B., 2012).
- Rhodococcus equi
Em macroscopia observou-se que todas as colônias cresceram da mesma forma, visto que todas as linhagens pertenciam a mesma espécie; assim, nas primeiras 24 horas de crescimento, apresentaram coloração creme, aspecto cremoso e formação de colônias isoladas; com 48 horas, o crescimento apresentou coloração alaranjada, aspecto mucoide e brilhante, formando colônias com forma de “gota de lágrima” e bordas lisas.
Em microscopia óptica foi possível visualizar a alteração morfológica decorrente do ciclo celular da espécie. Com menos de 24 horas, foram observados bacilos Gram-positivos; após 24 horas, foram visualizados cocos Gram-positivos, permitindo dizer que as linhagens estavam de acordo com relatos literários (WITHMAN, W. B. et al, 2012).
- Staphylococcus spp
Em macroscopia observou-se que todas as linhagens (IAL 1598, 1603, 1604, 2012), apresentaram crescimento abundante em 24 horas, com coloração amarelada, aspecto cremoso e colônias pequenas. Em ágar sangue, o padrão de crescimento se repetiu, porém a cepa com IAL 2012 apresentou formação halo de β – hemólise, que pode se formar nesta espécie (ANVISA, 2008). Em microscopia óptica, foram observados cocos Gram-positivos, que se distribuíam pela lâmina em diplococos, arranjos lineares e arranjos semelhantes a “cachos de uva”. Os crescimentos e as características microscópicas encontradas estavam de acordo com a literatura (WITHMAN, W. B. et al., 2009).
- Streptococcus spp
Em macroscopia foram observados crescimento em 24 horas, das linhagens IAL 1828 e 1832, em jarra de anaerobiose, com formação de zonas de α – hemólise, formando um halo esverdeado ao redor das colônias brilhantes, com aspecto seco, tamanho pequeno e bordas delimitadas. Em microscopia foram observados cocos Gram-positivos dispostos aos pares ou em alinhamento, sendo que os cocos da linhagem IAL 1828 foram considerados maiores quando comparados aos da linhagem IAL 1832. Desta forma, pode-se afirmar que para as linhagens desse gênero, as características observadas foram compatíveis com as descritas na literatura (WITHMAN, W. B. et al., 2009).
Análises Bioquímicas
- Corynebacterium spp
Para as diferentes linhagens com diferentes identificações de espécie foram realizadas provas de catalase, redução de nitrito a nitrato, consumo de glicose, sacarose e trealose, além de redução de telurito. Os resultados obtidos foram comparados com a literatura de referência (WITHMAN, W. B, 2012), permitindo concluir que as linhagens de IAL 93, 99, 101 e 104 não apresentaram divergências em relação ao que se era esperado. A linhagem IAL 1817 não apresentou consumo de trealose, divergindo da literatura.
- Rhodococcus equi
Para as diferentes linhagens da espécie trabalhada foram realizadas provas de catalase, produção de indol, consumo de lactose, glicose, manitol e xilose, descarboxilação de lisina, ornitina, arginina e degradação da ureia. Os resultados obtidos foram comparados com a literatura de referência (PRESCOTT, J. F., 1991), permitindo concluir que as linhagens IAL 1997, 2043, 2052 e 2106 não apresentaram divergências, porém, existem relatos em desacordo sobre o consumo de glicose para a espécie (SILVA, P. et al., 2010). A linhagem IAL 2042 não apresentou degradação da ureia, divergindo das outras linhagens, porém nem todas as linhagens degradam este substrato (SILVA, P. et al., 2010).
- Staphylococcus spp
Para as diferentes linhagens deste gênero foram realizadas provas de catalase, coagulação de plasma, consumo de manitol, degradação de ureia, despolimerização de DNA, além de antibiograma com Novobiocina (5µg). Os resultados obtidos foram comparados a literatura de referência (ANVISA, 2008), permitindo concluir que todas as linhagens pertenciam a espécie S. aureus.
- Streptococcus spp
Para as diferentes linhagens deste gênero foram realizadas provas de catalase, hidrólise de esculina e detecção de α-hemólise, além de antibiograma com Optoquina (5mcg). Os resultados obtidos foram comparados com a literatura de referência (ANVISA, 2008), permitindo concluir que as espécies trabalhadas pertenciam ao grupo viridans, não permitindo afirmar a espécie das linhagens.
MALDI-TOF
As diferentes espécies de Corynebacterium, Staphylococcus e Streptococcus foram identificadas por extração direta do crescimento, enquanto as diferentes cepas de Rhodococcus equi apresentaram melhor identificação usando extração proteica. A técnica permitiu a identificação de todas as espécies do gênero Corynebacterium, porém não identificou os biovares das espécies C. diphtheriae trabalhadas.
As linhagens de Rhodococcus equi foram a identificadas com nomenclatura de Rhodococcus hoagii, identificação explicada pela semelhança, considerada controversa, constatada entre as espécies de Rhodococcus equi e Corynebacterium hoagii (VÀZQUEZ- BOLAND, J. A., SCORTTI, M., MEIJER, W., 2020).
Todas as linhagens do gênero Staphylococcus foram confirmadas como da espécie S. aureus. No caso das diferentes espécies de Streptococcus, a técnica identificou a linhagem IAL 1832 como S. sanguinis, porém, a linhagem IAL 1828 teve resultado inconclusivo, pois houve identificação de duas espécies, S. mitis e S. pneumoniae, sendo possível descartar a segunda espécie pelo cultivo em ágar bile esculina, que foi negativo, diferindo do esperado para esta espécie (ANVISA, 2008).
Identificação por Sequenciamento Genético
Com base nos resultados obtidos nas técnicas de diferenciação descritas anteriormente, foram selecionadas como para identificação por sequenciamento genético as linhagens de Corynebacterium, visando diferenciar os biovares das linhagens IAL 99 e 101, e as linhagens de Streptococcus, visando confirmar suas espécies, visto que por pertencerem ao mesmo grupo de Estreptococos (viridans).
Os resultados obtidos permitem dizer que as bactérias Gram-positivas extraídas por kit apresentaram resultados melhores. Além disso, pode-se dizer que a identificação com os primers 27F/518R foi mais eficiente, visto que houve formação de leituras consensuais.
A partir do sequenciamento, foi possível dizer que as espécies do gênero Streptococcus foram identificadas como S. mitis (IAL 1828) e S. sanguinis (IAL 1832), com similaridades de 99,7% e 100%, respectivamente. As linhagens de Corynebacterium tiveram suas espécies identificadas, confirmando os resultados de outras técnicas, porém, não foi possível identificar os biovares das linhagens IAL 99 e 101, possivelmente pelos primers utilizados não abrangerem trechos do genoma que possibilitem isso.
As espécies de Corynebacterium identificadas e suas respectivas similaridades foram: C. diphtheriae (IAL 93) – 99,13%; C. diphtheriae (IAL 99) – 99,1%; C. diphtheriae (IAL 101) – 99,77%; C. pseudodiphtheriticum (IAL 104) – 98,19%; C. glutamicum (IAL 1817) – 100%.
CONCLUSÕES
As linhagens de Corynebacterium foram identificadas por meio das técnicas morfológicas, fenotípicas, MALDI-TOF e por sequenciamento genético, porém nenhuma das técnicas permitiu determinar seus biovares, sendo necessários mais estudos. As linhagens de Rhodococcus equi tiveram suas espécies confirmadas por meio das técnicas de identificação morfológica e por MALDI-TOF, porém a identificação por técnicas fenotípicas não foi conclusiva por esta espécie ser reativa ao uso de técnicas bioquímicas.
Com os resultados obtidos para as linhagens de Staphylococcus spp foi possível determinar a espécie por técnicas de identificação morfológica, fenotípicas e MALDI-TOF, permitindo autenticar a linhagem IAL 1598. As linhagens de Streptococcus spp tiveram seu gênero e grupo de classificação determinado como viridans por identificação fenotípica e morfológica, porém as suas espécies só foram confirmadas com a identificação por MALDI- TOF e sequenciamento genético.
Assim, verificou-se que os métodos de identificação utilizados foram eficientes para as bactérias Gram-positivas estudadas neste trabalho, sendo que a identificação por MALDI- TOF se destacou como a mais assertiva nos diferentes gêneros trabalhados. Também foi possível verificar que o método de liofilização empregado para a manutenção das linhagens foi eficaz, uma vez que a maioria das características informadas na época do depósito corresponderam as análises realizadas neste trabalho, após muitos anos de preservação.
REFERÊNCIAS BIBLIOGRÁFICAS
ANDRADE, T. S. Seção de coleção de culturas do Instituto Adolfo Lutz – 68 anos de história dedicados à saúde pública. Boletim Epidemiológico Paulista, São Paulo, nov. 2008.
ANVISA. Módulo 4 – Gram-positivos. Disponível em <https://www.anvisa.gov.br/servicosaude/controle/rede_rm/cursos/boas_praticas/modulo4/ imp_stre4.htm>. Acesso em 14 de outubro de 2022.
BECKER, P. et al. Public microbial resource centers: key hubs for Findable, Accessible, Interoperable and Reusable (FAIR) microorganisms and genetic materials. Disponível em: <https://pubmed.ncbi.nlm.nih.gov/31471301/>. Acesso em 17 de outubro de 2022.
JANSSENS, D., et al. The role of public biological resource centers in providing a basic infrastructure for microbial research. Disponível em: <https://pubmed.ncbi.nlm.nih.gov/20399265/>. Acesso em 17 de outubro de 2022.
MACHADO, J. B. A. Uso da biblioteca genômica RNAr 16S como ferramenta para o estudo da microbiota fecal humana. Disponível em <https://www.teses.usp.br/teses/disponiveis/9/9136/tde-16012014-140900/pt-br.php>. Acesso em 18 de novembro de 2022.
PRESCOTT, J. F. Rhodococcus equi: an animal and human pathogen. Clinical Microbiology Reviews. Vol. 4, 1991. P. 20 – 34. Disponível em < https://www.ncbi.nlm.nih.gov/pmc/articles/PMC358176/pdf/cmr00042-0036.pdf>. Acesso em 14 de novembro de 2022.
SILVA, P. et al. Rhodococcus equi isolation from sputum of patients with suspected tuberculosis. Disponível em <https://www.scielo.br/j/mioc/a/yT9zSjHLzC9tbDL5L3cX4Mz/?lang=en&format=html>. Acesso em 16 de novembro de 2022.
VÀZQUEZ-BOLAND, J. A., SCORTTI, M., MEIJER, W. Conservation of Rhodococcus equi (Magnusson 1923) Goodfellow and Alderson 1997 and rejection of Rhodococcus hoagii (Morse 1912) Kämpfer et al. 2014. International journal of systematic and evolutionary microbiology, 2020. Disponível em < https://pubmed.ncbi.nlm.nih.gov/32375930/>. Acesso em 14 de outubro de 2022.
WITHMAN, W. B. Bergey’s Manual of Systematics of Archaea and Bacteria. First Edition. 2012.
WITHMAN, W. B. et al. Bergey’s Manual of Systematic Bacteriology. Second Edition. Volume Three – The Firmicutes. 2009.
WITHMAN, W. B. et al. Bergey’s Manual of Systematic Bacteriology. Second Edition. Volume Five – The Actinobacteria, Part A. 2012.
Capítulo de livro publicado no Livro da IV Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
doi.org/10.53934/9786599965821-22
Este trabalho foi escrito por:
Letícia de Carvalho dos Santos1, Elizabeth Harummyy Takagi2
1 Estudante do Curso de Vigilância Epidemiológica em Saúde Pública – NCMO – Instituo Adolfo Lutz. [email protected]
2Docente/pesquisadora do Depto de Coleção de Micro-organismos – NCMO.
RESUMO
Os micro-organismos desempenham um importante papel na manutenção da vida no planeta devido à sua imensa biodiversidade, fornecendo recursos ilimitados para o desenvolvimento de pesquisas e criação de novos conhecimentos. O presente estudo tem como objetivo realizar a autenticação de nove linhagens bacterianas preservadas pelo método de liofilização no Núcleo de Coleção de Micro-organismos (NCMO) do Instituto Adolfo Lutz. Como metodologias foram adotadas técnicas bioquímicas fenotípicas e técnicas moleculares como MALDI-TOF e sequenciamento do gene codificante da porção 16S ribossomal. Duas linhagens de Acinetobacter baumanni foram caracterizadas por provas bioquímicas, MALDI-TOF, sequenciamento e amplificação do gene codificador da beta-lactamase do subgrupo OXA-51. Quatro linhagens da bactéria Citrobacter sp., uma linhagem de Edwardsiella tarda, uma linhagem de Klebsiella sp e uma linhagem de Raoultella planticolla foram autenticadas com o uso do MALDI-TOF complementado com provas bioquímicas. Ao término do estudo, as linhagens autenticadas ficaram disponíveis para fornecimento público, permitindo seu uso em pesquisas de grande relevância, em trabalhos industriais e controle de qualidade em Laboratórios Centrais de Saúde Pública (LACENs), contribuindo com a vigilância epidemiológica e consequentemente fazendo parte da história da saúde pública do país.
Palavras-chave: Autenticação, Bactérias Gram-negativas, Coleção, MALDI, Saúde Pública
INTRODUÇÃO
Os micro-organismos desempenham um papel significativo na manutenção da vida no planeta devido à sua imensa biodiversidade, fornecendo recursos ilimitados para o desenvolvimento de pesquisas e criação de novos conhecimentos (JANSSENS, D. et al., 2010).
Os centros de coleção de cultura atuam como depósitos de linhagens para fins de processamento de patentes no campo de biotecnologia, preservando os micro-organismos de forma confidencial e segura, disponibilizando-os após o período de sigilo, além de fornecer linhagens usadas em pesquisas para confirmações de resultados (SMITH, 2003).
O Núcleo de Coleção de Micro-organismos (NCMO) do Instituto Adolfo Lutz atua como coleção de serviço, contando com mais de dez mil linhagens de importância em saúde armazenadas desde a década de 40 em acervo, conservando e distribuindo materiais biológicos com critérios reconhecidos para indústrias e pesquisas (ANDRADE, T. S. et al, 2008). No entanto, devido à diversidade do acervo e tempo de armazenamento muitas linhagens ainda necessitam de autenticação por novas metodologias de identificação e caracterização, por isso o presente trabalho visa autenticar algumas linhagens de bactérias Gram negativas depositadas há anos, sem caracterização anterior, garantindo desta forma o fornecimento futuro de material com qualidade assegurada e contribuindo na saúde pública e vigilância epidemiológica.
As linhagens abordadas no presente trabalho são importantes patógenos associados às Infecções Relacionadas à Assistência à Saúde (IRAS), sendo elas: Acinetobacter baumannii, Citrobacter sp., Edwardsiella tarda, Klebsiella sp. e Raoultella sp.
MATERIAL E MÉTODOS
Foram selecionadas nove linhagens de bactérias Gram-negativas conservadas pelo método de liofilização, descritas na Tabela 3.
Foi realizada a análise macroscópica através da observação do crescimento dos micro- organismos em repique em placa de ágar comum, incubada por 24 horas a 37 ºC. A análise microscópica foi analisada por coloração de Gram e a leitura em aumento de 1000x.
A autenticação foi realizada por três métodos: MALDI-TOF (espectrometria de massa por ionização com dessorção a laser assistido por matriz e analisador de tempo de voo), avaliação bioquímica e sequenciamento Sanger.
A identificação por MALDI-TOF foi realizada por extração direta conforme estabelecido pelo fabricante Bruker. As proteínas do crescimento bacteriano depositado na placa do MALDI-TOF foram extraídas com ácido fórmico 95%, aplicando uma matriz HCCA (α- cyano-4-hydroxycinnamic Acid) que sofreu ionização pela incidência de um feixe de laser do equipamento e estabeleceu um espectograma de massa para leitura das proteínas.
Para as linhagens de Acinetobacter baumannii foram realizadas as provas bioquímicas: malonato, glicose, oxidase, lactose, hemólise em ágar sangue, degradação da gelatina, indol, sacarose, H2S, ureia e lisina. Para as enterobactérias foram feitas as análises de: nitrato, lisina, malonato, ornitina, ureia, citrato, glicose, oxidase, catalase, lactose, indol, sacarose, H2S, motilidade e dulcitol.
A interpretação dos resultados foi realizada através da mudança de cores dos meios pelo indicador de pH. A assimilação de carboidratos torna o meio ácido, gerando alteração de cor e sinalizando um resultado positivo. Nas provas de assimilação de aminoácidos, o meio se torna básico pela geração de um grupo amina e a cor permanece a mesma, indicando a positividade.
Para avaliação bioquímica também foi utilizado o Sistema Bactray I, seguindo as orientações do fabricante para execução e leitura dos resultados. Cada linhagem foi inoculada em água ultrapura, obtendo turvação final nº 0,5 na escala de McFarland , e dispensada no painel de provas direcionando aos depósitos de substrato. A leitura foi realizada após 24h de incubação a 37 ºC em condições de umidade 40-60%. Os resultados foram interpretados pela alteração da coloração dos substratos.
Foram realizadas reações em cadeia polimerase (PCR) com o objetivo de amplificar o material genômico das amostras a serem analisadas por sequenciamento. O alvo escolhido foi a porção do gene codificante 16S ribossomal com o uso dos oligoiniciadores 27F / 518R e 341F / 1492R para todas as amostras.
Para as linhagens de Acinetobacter baumannii também foi realizado PCR utilizando os oligoiniciadores OXA-51 F e OXA-51 R, que são específicos para esta espécie e auxiliam no direcionamento da identificação (TURTON, J. F. et al., 2006).
A reação do sequenciamento foi realizada com o uso do BigDye™ Terminator v3.1 Cycle Sequencing Kit. As amostras foram sequenciadas pelo aparelho 3500 Genetic Analyzer (ThermoFisher).
RESULTADOS E DISCUSSÃO MALDI-TOF
Os resultados do MALDI-TOF são classificados de acordo com a pontuação conferida pela pesquisa em banco de dados da Bruker, sendo a pontuação de 2,00 a 3,00 considerada uma identificação de alta confiança, 1,70 a 1,99 identificações de baixa confiabilidade e abaixo de 1,69 sem identificação.
A leitura dos padrões de proteína das linhagens de Acinetobacter baumanii, Citrobacter sp. e Raoultella planticola apresentaram diferentes resultados com um score superior a 2,0 e por isso não foi possível chegar à uma identificação conclusiva por essa técnica.
Para a linhagem Edwardsiella tarda foi concluída a identificação de gênero e espécie, com um score de 2,16. O IAL 504 Klebsiella sp. foi identificado como Klebsiella pneumoniae, com score 2,33, apresentando duas identificações para subespécie com scores considerados de alta confiança.
PERFIL BIOQUÍMICO
O resultado das provas para Acinetobacter baumannii foi compatível com o perfil do gênero e espécie (ANVISA, 2004), exceto pelo consumo do malonato da linhagem A. baumanni IAL 2458 que foi negativo.
Para a identificação das linhagens de Citrobacter foi utilizado o fluxograma desenvolvido por O’Hara e colaboradores (O’HARA, C. M. et al, 1995). Através da comparação dos resultados obtidos com o fluxograma, foi possível determinar que as linhagens IAL 239 e IAL 244 são da espécie braakii, enquanto o IAL 91 e IAL 92 são da espécie youngae.
Os resultados das provas bioquímicas direcionaram a identificação do IAL 1973 para Edwardsiella tarda e do IAL 504 como Klebsiella pneumoniae.
Segundo Walckenaer et al. (2008), isolados de R. planticola são negativos para ODC (ornitina) e variáveis para a produção de indol, enquanto a R. ornithinolytica é positiva para o teste de descarboxilase da ornitina e obrigatoriamente não produz indol. Considerando que a linhagem 2075 apresentou resultado negativo para a prova de ornitina e positivo para indol, foi possível concluir que esta linhagem pertence à espécie R. plancitcola..
BACTRAY
O sistema Bactray I é direcionado para bactérias Gram-negativas que são oxidase negativa. Os resultados foram analisados na plataforma online Bactray, sendo considerado um resultado aceitável aqueles com semelhança acima de 80%.
O sistema determinou 76,35% de semelhança com Acinetobacter baumannii para a linhagem 2457, sendo que para um perfil com 100% de semelhança pelo sistema, a bactéria deveria ser capaz de transformar fenilalanina em ácido fenilpirúvico. Contudo, a literatura demostra que apenas 87% das linhagens de A. baumannii apresentam positividade para esta prova (HOLT, J. G. 1994), levantando então o questionamento da assertividade desta plataforma na análise desta espécie. Não foi possível identificar o IAL 2458 com este sistema, apresentando diferentes relações com resultados abaixo de 50%.
As linhagens de Citrobacter sp., IAL 91 e IAL 92, apresentaram 99,97% de compatibilidade com Citrobacter freundii. Todavia, o sistema Bactray I não tem em sua composição o substrato “dulcitol”, prova importante para diferenciar as espécies C. freundii e C. youngae, e por isso este resultado não pode ser considerado como final sem a prova complementar citada.
A linhagem de Citrobacter sp. IAL 239 chegou a uma identificação com 100% de similaridade para a espécie C. braakii e o IAL 244 apresentou 97,76% de semelhança para C. braakii.
Para a linhagem IAL 1973, foi observado semelhança de 99,94% para Edwardsiella tarda, correspondendo à identificação original. Para a linhagem Klebsiella sp., IAL 504, foi observada semelhança de 97,63% para a identificação de Klebsiella pneumoniae.
Não foi possível concluir a identificação com este sistema para a linhagem de Raoultella planticola, pois este gênero não é abordado pelo Bactray I.
SEQUENCIAMENTO
Devido a não leitura das duas cadeias de polinucleotídeos das amostras, os resultados obtidos neste processo de sequenciamento não foram decisivos para concluir a autenticação das linhagens, tornando necessária a associação deste resultado com os demais obtidos pelas técnicas anteriores.
OXA-51
O resultado da PCR feita com os oligoiniciadores OXA-51F e OXA-51R foi visualizado em gel de Agarose 1,5%, apresentando bandas claras e únicas e confirmando a espécie das linhagens trabalhadas, pois, de acordo com Turton (2006), o gene codificante da beta- lactamase do subgrupo OXA-51 é intrínseco das linhagens de Acinetobacter baumannii.
IDENTIFICAÇÃO FINAL DAS LINHAGENS
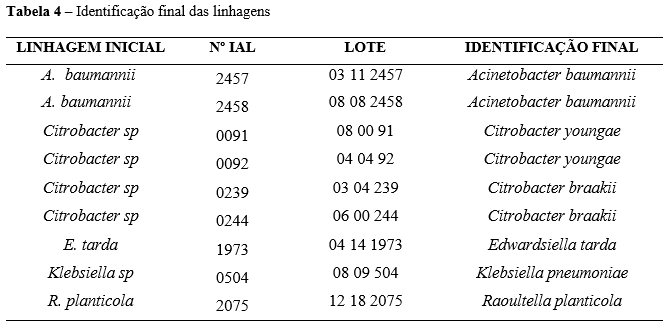
Após a realização das técnicas de autenticação, bioquímicas e moleculares, a compilação dos resultados permitiu caracterizar as linhagens para gênero e espécie, como pode ser observado na Tabela 4.
CONCLUSÃO
As provas bioquímicas, convencionais e do Sistema Bactray, foram úteis na identificação das bactérias Gram-negativas em conjunto com outras técnicas complementares. Neste trabalho, a caracterização fenotípica foi fundamental no direcionamento da identificação das espécies das linhagens de Citrobacter sp e Raoultella planticolla., que em conjunto com as demais técnicas permitiu a conclusão da autenticação.
A técnica de MALDI-TOF foi capaz de concluir a identificação de gênero e espécie das linhagens Edwardsiella tarda e Klebsiella pneumoniae e auxiliou no direcionamento das espécies das demais linhagens, como no caso da Raoultella planticolla e espécies de Citrobacter sp.
Não foi possível concluir a identificação das linhagens através do sequenciamento devido a não leitura das duas cadeias de polinucleotídeos das amostras, não formando um consenso. Todavia, não se pode determinar que este não é um bom método de autenticação das bactérias Gram-negativas pois a falha na classificação pode ter ocorrido devido à falhas do desenvolvimento do trabalho, necessitando confirmar os resultados.
A amplificação do gene OXA-51, possibilitou a identificação específica das amostras de A. baumannii (IAL 2457 e IAL 2458) em razão de sua inerência para com a espécie.
Ao final dos procedimentos, as linhagens autenticadas passaram a estar disponíveis para fornecimento, permitindo seu uso em pesquisas de grande relevância, em trabalhos industriais e em Laboratórios Centrais de Saúde Pública (LACENs), contribuindo com a vigilância epidemiológica e consequentemente fazendo parte da história da saúde pública do país.
REFERÊNCIAS
ANDRADE, T. S. et al. Seção de Coleção de Culturas do Instituto Adolfo Lutz – 68 Anos de história dedicados à saúde pública. BEPA – Boletim Epidemiológico Paulista.
DETECÇÃO e Identificação de Bactérias de Importância Médica. Agência Nacional de Vigilância Sanitária, Mod V, 2004. 95p.
HOLT, J. G. Bergey’s Manual of Determinative Bacteriology. Ninth Edition – 1994.
JANSSENS, D. et al. The role of public biological resource centers in providing a basic infrastructure for microbial research. Research in microbiology v. 161,6 (2010): 422-9. doi:10.1016/j.resmic.2010.03.009.
O’HARA, C. M.; ROMAN, S. B.; MILLER, J. M. Ability of Commercial Identification Systems To Identify Newly Recognized Species of Citrobacter. Journal of Clinical Microbiology, v.33, n.1, p. 242–245, 1995.
SMITH, D. “Culture collections over the world.” International microbiology : the official journal of the Spanish Society for Microbiology v. 6,2 (2003): 95-100. doi:10.1007/s10123- 003-0114-3
TURTON, J. F. et al. Identification of Acinetobacter baumannii by Detection of the blaOXA-51-like Carbapenemase Gene Intrinsic to This Species. Journal of Clinical Microbiology. London, V. 44, No. 8, , p. 2974–2976, 2006.
WALCKENAER, E. et al. How to identify Raoultella spp. including R. ornithinolytica isolates negative for ornithine decarboxylase? The reliability of the chromosomal bla gene. Journal of microbiological methods v. 75,3 (2008): 405-10. doi:10.1016/j.mimet.2008.07.01.
Capítulo de livro publicado no Livro da IV Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
doi.org/10.53934/9786599965821-21
Este trabalho foi escrito por:
Júlia Cavalcante Alexandrino1, Mariana Sequetin Cunha2
Estudante do Curso de Especialização em Vigilância Laboratorial em Saúde Pública – CV – IAL; E-mail: [email protected]
2Docente/ Pesquisador do Núcleo de Doenças de Transmissão Vetorial – NDTV – IAL
RESUMO
A ocorrência das arboviroses, vírus transmitidos por artrópodes, tem sido uma ameaça à saúde humana há séculos. Em média, as arboviroses são responsáveis por mais de 700.000 mortes por ano, e a frequência e a magnitude desses surtos estão aumentando globalmente. O gênero Orthobunyavírus (OBVs) compreende uma ampla gama de vírus transmitidos por artrópodes que pertencem a família Peribunyaviridae da ordem Bunyavirales e são prevalentes em todo o mundo. O presente projeto tem por objetivo avaliar a presença de Orthobunyavirus em amostras de sangue de aves coletadas na região do Vale do Ribeira, no Estado de São Paulo, durante investigações de campo realizadas em 2022. As aves foram capturadas com uso de Redes-de-neblina e marcadas com anilhas metálicas do CEMAVE/ICMBio. A coleta das amostras utilizadas aconteceu entre fevereiro e setembro de 2022, onde um total de 270 amostras de sangue foram coletadas e 21 foram selecionadas dentre as que possuíam um maior volume e que testaram negativo para Flavivírus e Alphavírus. Durante o isolamento, nenhum efeito citopático foi observado e as reações de imunofluorescência não apresentaram nenhum indicativo de reação positiva. Diversos trabalhos já evidenciaram a provável circulação do vírus desse gênero na região, e outros mostram que um deles, o Oropouche, que já foi responsável por diversas epidemias, vem se dispersando silenciosamente pelo Brasil fora dos locais endêmicos. Apesar de alta, a viremia em aves e outros vertebrados pequenos é curta, o que faz com que haja necessidade de testar um maior número de amostras e realizar uma coleta constante. Os resultados obtidos no presente estudo mostram que não há circulação de Orthobunyavirus nesta região no período estudado, no entanto, ele tem como limitação o pequeno número amostral avaliado. Uma vigilância ativa e continua permitirá uma melhor compreensão da incidência e da prevalência das infecções por Orthobunyavirus, e ajudará a prever possíveis surtos na região.
Palavras-chave: Arbovírus, Orthobunyavirus, Vigilância.
INTRODUÇÃO
A ocorrência das arboviroses, vírus transmitidos por artrópodes, tem sido uma ameaça à saúde humana há séculos. Em média, as arboviroses são responsáveis por mais de 700.000 mortes por ano, e a frequência e a magnitude desses surtos estão aumentando globalmente
(WEAVER & REISEN, 2010). Há cerca de 120 anos o primeiro arbovírus foi descoberto pelos cientistas Walter Reed, James Carroll, Aristides Agramonte e Jesse Lazear, que identificaram o Flavivírus causador da febre amarela (WEAVER et al., 2018;CASSEB et al., 2013; DOUAM & PLOSS, 2018). Posteriormente, em 1916, a Fundação Rockefeller criou a “Comissão da Febre Amarela” financiando pesquisas de cientistas nos Estados Unidos, Brasil, Colômbia, Nigéria e Uganda (DOUAM & PLOSS, 2018). Essas pesquisas geram como subproduto o isolamento de arbovírus até então desconhecidas. Esses e outros acontecimentos resultaram na descoberta e isolamento de centenas de novos arbovírus (WEAVER et al., 2018; CASSEB et al., 2013).
O gênero Orthobunyavirus (OBVs) compreende uma ampla gama de vírus transmitidos por artrópodes que pertencem a família Peribunyaviridae da ordem Bunyavirales, de acordo com o International Committee on Taxonomy of Viruses (ICTV), e são prevalentes em todo o mundo. Apesar de uma crescente consciência dos potenciais efeitos prejudiciais dos Orthobuniavírus na saúde única, o conhecimento sobre seus ciclos enzoóticos de transmissão, amplificação, hospedeiros e vetores, e fatores bióticos e abióticos relacionados aos eventos de transbordamento para humanos são limitados (WINDHABER ET AL., 2021).
REFERÊNCIAL TEÓRICO
Os vírus pertencentes ao gênero Orthobunyavirus são compostos por três segmentos genômicos distintos de RNA de fita simples (ssRNA) de polaridade negativa, identificados como grande (L), médio (M) e pequeno (S) (DOLGOVA et al., 2022). Há pouca ou nenhuma informação sobre os ciclos de transmissão para muitos dos Orthobunyavirus descritos até agora (DIAS et al., 2022).
Entre 1978 e 1990, FERREIRA et al. realizaram um trabalho de vigilância de arbovírus em São Paulo (SP) pelo Instituto Adolfo Lutz. Um total de 11.000 amostras foram coletadas na região do Vale do Ribeira, e diversos arbovírus foram detectados através de técnicas de isolamento em camundongos, seguida da fixação de complemento. Além disso, anticorpos anti- orthobunyavirus foram identificados pelo método indireto de inibição da hemaglutinação, dentre eles, duas amostras com resultado positivo para o Orthobunyavirus Caraparu (FERREIRA et al., 1994). Em estudos sorológicos realizados por IVERSSON et al., em população humana da região do Vale do Ribeira, amostras de 337 estudantes foram coletadas entre os anos de 1981 e 1982 e um total 12 amostras apresentaram anticorpos para vírus do gênero Orthobunyavirus (IVERSSON et al., 1983). IVERSSON et al 1980, já havia realizado outro estudo em 1980, em que pesquisou anticorpos para arbovírus em 516 moradores de Pariquera-Açu, região do Vale do Ribeira, em São Paulo, onde 4,6% apresentavam anticorpos para o vírus Caraparu (BUSCH IVERSSON; TRAVASSOS DA ROSA; TRAVASSOS DA ROSA, 1980).
O vírus Oropouche (OROV) é o causador da febre Oropouche e está historicamente envolvido em surtos explosivos com centenas de milhares de casos, principalmente na região amazônica. Um estudo de Romero et al., mostra que no Brasil, nos últimos anos, a disseminação do OROV tem sido cada vez mais evidenciada em outras regiões do país e não apenas em áreas endêmicas, mostrando que o vírus está mais disseminado do que se acreditava originalmente no Brasil (ROMERO-ALVAREZ & ESCOBAR, 2018; DIAS et al., 2022). Isso gera a hipótese da circulação críptica do vírus na região do Vale do Ribeira, que é uma região afetada por diversas epidemias onde presença de outros Orthobunyavirus já foi evidenciada.
OBJETIVOS
O presente projeto tem por objetivo avaliar a presença de Orthobunyavirus em amostras de sangue de aves coletadas na região do Vale do Ribeira, no Estado de São Paulo, durante investigações de campo realizadas em 2022.
MATERIAIS E MÉTODOS
Este estudo foi realizado seguindo as diretrizes e normas regulamentadoras de pesquisas envolvendo amostras biológicas e teve a aprovação do Comitê de Ética do ICMBio 794-1. Os procedimentos de manipulação e processamento das amostras biológicas foram desenvolvidos em laboratórios de Nível de Biossegurança 2 (NB2), de acordo com o preconizado pela OMS.
A coleta das amostras utilizadas aconteceu entre fevereiro e setembro de 2022, de aves da região de Iguape, município situado no Vale do Ribeira em São Paulo. As aves foram capturadas com uso de Redes-de-neblina. As revisões eram feitas de hora em hora, ou períodos mais curtos caso a temperatura do dia esteja muito alta, evitando estresse dos animais. As aves capturadas foram marcadas com anilhas metálicas do CEMAVE/ICMBio – Centro Nacional de Pesquisa e Conservação de Aves Silvestres, medidas e soltas. O sangue foi coletado com uso de capilar e as amostras foram identificadas de acordo com data e espécie, e imediatamente colocadas em nitrogênio líquido
Um total de 270 amostras de sangue foram coletadas e 21 foram selecionadas dentre as que possuíam um maior volume e que eram negativas para gêneros Flavivirus e Alphavirus por RT-qPCR. As amostras foram inoculadas em monocamadas de células Vero (células epiteliais renais extraídos de um macaco verde africano), C6/36 (uma linhagem de células originadas do mosquito Aedes albopictus) e em células TRA-171 (derivadas da larva do mosquito Toxorhynchites amboinensis), somando um total de 5 passagens. Para cada passagem uma placa com um total de 24 poços contendo a monocamada de células foi utilizada e 50 microlitros de cada amostra foram inoculados (sempre em poços separados). Não foram feitas duplicatas e para cada placa haviam 2 controles negativos e um positivo. Cada passagem durava um período de 12 dias de incubação em média e as células eram monitoradas diariamente, sob um microscópio de luz invertida, para observação de possível efeito citopático. Em seguida os Ensaios de Imunofluorescência Indireta (IFA) com anticorpos específicos para os Orthobunyavirus Oropouche, Itaqui e Murucutu foram realizados seguindo o protocolo descrito por Gluber et al 1984. As lâminas foram examinadas sob microscópio de imunofluorescência.
RESULTADOS E DISCUSSÃO
Durante o isolamento nenhum efeito citopático foi observado em nenhuma das passagens realizadas. Os controles positivos e negativos atenderam o resultado esperado, sendo que no positivo o efeito citopático foi observado e no negativo as células permaneceram íntegras. Nas reações de imunofluorescência, realizadas para verificar a presença de Orthobunyavirus nas amostras de aves, nenhum indicativo de reação positiva foi observado. Esse resultado não descarta a necessidade de ações de vigilância ativa para essas arboviroses na região, pois apesar de alta, a viremia em aves e outros vertebrados pequenos é curta, o que faz com que haja necessidade de testar um maior número de amostras e realizar uma coleta constante (ASSIS, 2013).
As aves e os roedores são considerados os hospedeiros mais importantes nas arboviroses de impacto para a saúde pública, e os mosquitos e carrapatos são considerados vetores artrópodes mais importantes (GUBLER, 2002). Acredita-se que as aves migratórias sejam responsáveis pela ampla distribuição geográfica de algumas arboviroses importantes (MANCUSO et al., 2022). Os vírus Apeu, Caraparu, Itaqui, Marituba, Nepuyo, Murutucu e Oriboca já foram isolados na floresta amazônica, que é uma das maiores reservas de arbovírus do mundo, e apesar da busca ativa naquela região ter sido realizada na década de cinquenta, ainda pouco se sabe sobre esses vírus (MACEDO et al., 1961). A literatura sobre os alguns sorogrupos do gênero Orthobuyavirus é escassa e ainda hoje sua importância é subestimada (ASSIS, 2013).
O vírus Oropouche é responsável por uma das doenças febris mais importantes do Brasil. O conhecimento atual sobre a distribuição do Oropouche, assim como da maioria dos Orthobunyavirus, é baseado na vigilância epidemiológica passiva de amostras coletadas em surtos (ROMERO-ALVAREZ & ESCOBAR, 2018). Por outro lado, a vigilância ativa revelou a circulação críptica do vírus em até 2% das pessoas que vivem em áreas não endêmicas, o que sugere uma distribuição silenciosa desse vírus para regiões não endêmicas, reforçando a necessidade de uma vigilância ativa mesmo nessas áreas (F. P. PINHEIRO et al., 1981).
Outra evidência da necessidade de uma vigilância ativa, é o relato do vírus Caraparu na região do Vale do Ribeira no estado de São Paulo (SP), em um trabalho de vigilância em aves realizado pelo Instituto Adolfo Lutz entre 1978 e 1990 (FERREIRA et al., 1994), bem como outros inquéritos sorológicos também realizados nessa região em crianças, adolescentes e adultos, que tiveram como resultado a detecção de anticorpos para o vírus Caraparu, Guaroa, Xingu, Utinga e Tacaiuma ( ROSA, 1980; IVERSSON et al., 1983).
Esses resultados indicam que para uma melhor perspectiva do atual cenário epidemiológico dessa região, uma testagem maior e uma coleta constante são medidas necessárias. Certamente, a distribuição subestimada dos Orthobunyavirus decorre do simples fato de que as investigações epidemiológicas para estes vírus estão concentradas apenas nas imediações de onde foram inicialmente isolados. Além disso, existem dificuldades nas investigações, devido à interpretação dos resultados dos testes sorológicos, já que são comuns reações cruzadas entre os vírus (ASSIS, 2013).
Nos últimos 30 anos, o surgimento e/ou ressurgimento de arboviroses tem representado uma considerável ameaça à saúde global, e são as ações de vigilância que podem ajudar a prever possíveis surtos. Um exemplo de que novas epidemias podem surgir a qualquer momento é a expansão geográfica contínua dos vírus da Dengue, juntamente com os surtos explosivos do vírus do Nilo Ocidental, do vírus Chikungunya e, mais recentemente, o vírus Zika (VASUDEVAN & HILGENFELD, 2018).
Grande parte das doenças causadas por arbovírus circulam em ambientes rurais entre seus vetores e seus hospedeiros vertebrados, que fazem a amplificação do vírus no ciclo de transmissão (VASUDEVAN & HILGENFELD, 2018). O ser humano pode atuar como um hospedeiro acidental ou como um reservatório, podendo dessa forma transmitir o vírus na presença de vetores competentes, e assim, disseminá-lo para fora do ambiente natural. Um conhecimento mais aprofundado de quais mecanismos impulsionam as mudanças nos ciclos de transmissão que resultam em que a população humana se torne significativamente mais exposta, ajudará a nos preparar para a próxima epidemia/pandemia emergente (OLIVAL et al., 2017).
CONCLUSÃO
Os Orthobunyavirus têm um grande potencial para causar surtos. Os resultados obtidos no presente estudo mostram que não há circulação de Orthobunyavirus nesta região no período estudado, no entanto, ele tem como limitação o pequeno número amostral avaliado. Uma vigilância ativa e continua permitirá uma melhor compreensão da incidência e da prevalência das infecções por Orthobunyavirus, e ajudará a prever possíveis surtos na região.
REFERÊNCIAS
ASSIS, M. (2013). “Caracterização dos vírus ITAQUI e CARAPARU (Orthobunyavirus do sorogrupo C): patogênese e genes induzidos da resposta imune inata” (Tese de Doutorado). Universidade Federal de Minas Gerais.
BUSCH IVERSSON, L., & COIMBRA, T. L. M. (1984). Encefalite na região do Vale do Ribeira, São Paulo, Brasil, no período pós-epidêmico de 1978 a 1983: situação do diagnóstico etiológico e características epidemiológicas. Revista de Saúde Pública, 18(4), 323–332. https://doi.org/10.1590/S0034-89101984000400007
BUSCH IVERSSON, L., TRAVASSOS DA ROSA, A. P., & TRAVASSOS DA ROSA, J. Estudos sorológicos para pesquisa de anticorpos de arbovírus em população humana da região do Vale do Ribeira. Ii-inquérito em pacientes do Hospital Regional de Pariquera-Açú, 1980.https://doi.org/10.1590/S0034-89101981000600002
CASSEB, A. DO R., CASSEB, L. M. N., SILVA, S. P., & VASCONCELOS, P. F. DA C. (2013). Vista do ARBOVÍRUS: Vet. e Zootec. https://rvz.emnuvens.com.br/rvz/article/view/1030/585
DIAS, H. G., DOS SANTOS, F. B., & PAUVOLID-CORRÊA, A. (2022). An Overview of Neglected Orthobunyaviruses in Brazil. Viruses, 14(5). https://doi.org/10.3390/V14050987
DOLGOVA, A. S., SAFONOVA, M. V., FAYE, O., & DEDKOV, V. G. (2022). Current View on Genetic Relationships within the Bunyamwera Serological Group. Viruses, 14(6), 1135. https://doi.org/10.3390/V14061135/S1
DOUAM, F., & PLOSS, A. (2018). Yellow fever virus: Knowledge gaps impeding the fight against an old foe. Trends in Microbiology, 26(11), 913. https://doi.org/10.1016/J.TIM.2018.05.012
FERREIRA, I., PEREIRA, L., ROCCO, I., MARTI, A., SOUZA, L., & IVERSON, L. (1994). Surveillance of arbovirus infections in the atlantic forest region state of São Paulo, Brazil. I. Detection of hemagglutination- inhibiting antibodies in wild birds between 1978 and 1990. Revista Instituto de Medicina Tropical, 36(3), 265– 274.
GUBLER, D. J. (2002). The global emergence/resurgence of arboviral diseases as public health problems. Archives of Medical Research, 33(4), 330–342. https://doi.org/10.1016/S0188-4409(02)00378-8
MACEDO, D. G., MAROJA, O. M., CAUSEY, C. E., & CAUSEY, O. R. (1961). The Isolation of Arthropod-Borne Viruses, including Members of Two Hitherto Undescribed Serological Groups, in the Amazon Region of Brazil *. The American Journal of Tropical Medicine and Hygiene, 10(2), 227–249. https://doi.org/10.4269/ajtmh.1961.10.227
MANCUSO, E., CECERE, J. G., IAPAOLO, F., DI GENNARO, A., SACCHI, M., SAVINI, G., SPINA, F., &
MONACO, F. (2022). West Nile and Usutu Virus Introduction via Migratory Birds: A Retrospective Analysis in Italy. Viruses, 14(2), 416. https://doi.org/10.3390/V14020416/S1
OLIVAL, K. J., HOSSEINI, P. R., ZAMBRANA-TORRELIO, C., ROSS, N., BOGICH, T. L., & DASZAK, P.
(2017). Host and viral traits predict zoonotic spillover from mammals. Nature 2017 546:7660, 546(7660), 646–650. https://doi.org/10.1038/nature22975
RADFORD, A. D., COYNE, K. P., DAWSON, S., PORTER, C. J., & GASKELL, R. M. (2007). Feline calicivirus. Veterinary Research, 38(2), 319–335. https://doi.org/10.1051/VETRES:2006056
ROMERO-ALVAREZ, D., & ESCOBAR, L. E. (2018). Oropouche fever, an emergent disease from the Americas. Microbes and Infection, 20(3), 135–146. https://doi.org/10.1016/j.micinf.2017.11.013
VASUDEVAN, S. G., & HILGENFELD, R. (2018). Dengue and Zika: Control and Antiviral Treatment Strategies (R. Hilgenfeld & S. G. Vasudevan, Eds.; 1st ed.).https://doi:10.1007/978-981-10-8727-1
Vector-borne diseases. 2010. https://www.who.int/en/news-room/fact-sheets/detail/vector-borne-diseases
WEAVER, S. C., & REISEN, W. K. (2010). Present and Future Arboviral Threats. Antiviral Research, 85(2), 328. https://doi.org/10.1016/J.ANTIVIRAL.2009.10.008
WINDHABER, S., XIN, Q., & LOZACH, P. Y. (2021). Orthobunyaviruses: From Virus Binding to Penetration into Mammalian Host Cells. Viruses, 13(5). https://doi.org/10.3390/V13050872
Capítulo de livro publicado no Livro da IV Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
doi.org/10.53934/9786599965821-20
Este trabalho foi escrito por:
Bryan Kaique de Carvalh¹; Dra Tânia Sueli de Andrade²
1Estudante do Curso de Vigilância Epidemiológica em Saúde Pública – NCMO – IAL – Central. [email protected]
2Pesquisadora Docente do Núcleo de Coleção de Micro-organismos– NCMO– IAL – Central.
Resumo: As coleções de micro-organismos são responsáveis pela preservação da biodiversidade, para isso é preciso que as linhagens armazenadas estejam corretamente autenticadas. O Núcleo de Coleção Micro-organismos do Instituto Adolfo Lutz possui em seu acervo aproximadamente 10.000 linhagens, sendo pouco mais de 600 fungos, importantes causadores de infecções oportunistas. O objetivo desse trabalho foi avaliar os métodos de liofilização e criopreservação; e caracterizar vinte linhagens fúngicas depositadas no NCMO, por meio de sua morfologia (micro e macroscópicas) e técnicas moleculares como MALDI-TOF e sequenciamento Sanger para os alvos ITS e gene da calmudolina. As linhagens de Apergillus fumigatus e A. terreus foram identificadas por MALDI-TOF; A. flavus apontado como membro do grupo Aspergillus flavus oryzae. As linhagens de Fusarium oxysporum e F. solani foram identificadas por MALDI-TOF e sequenciamento para alvo ITS; F. equiseti não pode ser identificado por técnicas moleculares. Uma linhagem de Curvularia foi identificada por sequenciamento do gene rDNA região ITS, outra linhagem foi parcialmente identificada, ambas tendo duas espécies como resultado, sendo diferenciadas por macromorfologia. As linhagens de Fonsecaea pedrosoi não apresentavam identificação prévia, pois foram encaminhadas para depósito recentemente, os resultados obtidos do sequenciamento do rDNA região ITS, confirmaram a identificação de origem para uma delas, e duas foram parcialmente identificadas. Porém uma linhagem foi identificada como Phialophora sp por sequenciamento por micromorfologia, não correspondendo à sua identificação de origem. Todos os gêneros trabalhados apresentaram características descritas na literatura. Tanto os métodos de identificação quanto os de armazenamento se mostram eficientes.
Palavras-chaves: armazenamento, autenticação, coleções de micro-organismos, Fonsecaea, fungos
Introdução
A preservação ex situ, fora do habitat natural, dos recursos microbiológicos é uma prática indispensável para o conhecimento e uso da biodiversidade, as chamadas coleções de cultura de micro-organismos tem um papel importante neste contexto, como o Núcleo de Coleção Micro-organismos do Instituto Adolfo Lutz, que possui em seu acervo aproximadamente
10.000 linhagens, sendo pouco mais de 600 fungos, importantes causadores de infecções consideradas clássicas e oportunistas. A preservação adequada dos isolados fúngicos é imprescindível para uso em investigações científicas (SETTE; PAGNOCCA; RODRIGUES, 2013; BOUNDY-MILLS et. al. 2019). Para a conservação de fungos filamentosos a preservação a longo prazo pode ser realizada através da criopreservação a – 80°C ou também pela liofilização, processo de sublimação a vácuo (OLIVEIRA; CORTEZ; SOUZA 2016; COSTA et al 2009). Após os processos de manutenção, a autenticação das linhagens garante a qualidade do acervo. No caso dos fungos filamentosos a taxonomia baseia-se principalmente em critérios morfológicos e nas características das estruturas de reprodução assexuada, sem que haja a necessidade de analisar os atributos bioquímicos e fisiológicos, como é realizado com as leveduras (WALSH; HAYDEN; LARONE, 2018). No entanto, alguns gêneros possuem grande similaridade morfológica dentre seus membros, dificultando a caraterização de suas espécies. Para essa diferenciação é necessário o uso de técnicas moleculares, como o sequenciamento genético para regiões ITS (Internal Transcribed Spacer) (WHITE et. al. 1990) e para o gene de calmodulina para o gênero Aspergillus (SABINO et al. 2015) assim como a identificação por MALDI-TOF (Matrix Assisted Lazer Desorption/Ionization – Time Of Flight Mass Spectrometry) técnica que analisa padrões de espectrometria de massa capaz de identificar linhagens fúngicas de forma extremamente rápida (ANTUNES 2019, PEREIRA 2019, PAZIANI 2021). Com o intuito de garantir a qualidade das linhagens disponibilizadas para a comunidade científica, o objetivo deste trabalho foi avaliar os diferentes métodos de manutenção e autenticação disponíveis no Núcleo, para inclusão no acervo de novas linhagens de fungos filamentosos depositados recentemente no NCMO.
MATERIAL E MÉTODOS
Foram utilizadas vinte linhagens depositadas no NCMO, tabela 1, pertencentes aos gêneros Aspergillus, Fusarium, Curvularia e Fonsecaea.
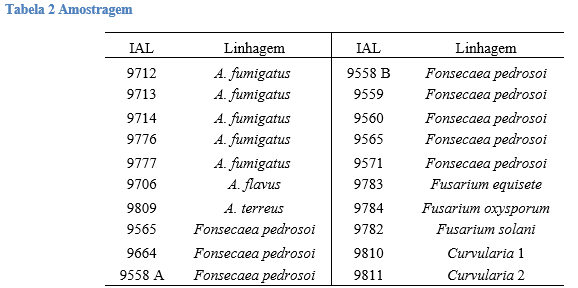
Identificação morfológica
Para identificação morfológica foram utilizadas às técnicas de microcultivo em lâmina e de colônia gigante, ambas em ágar dextrose batata (WALSH; HAYDEN; LARONE, 2018).
Reação de Sequenciamento
A extração do DNA genômico foi realizada utilizando o kit de extração Invitrogen PureLink® Genomic DNA mini kit (Invitrogen-Estados Unidos). Para as culturas de Aspergillus, Fusariun e Curvularia foram utilizadas culturas com 3-4 dias de crescimento em ágar dextrose batata e culturas de Fonsecaea sp foram utilizadas após 14 dias de crescimento em caldo malte. A identificação molecular foi realizada com sequenciamento Sanger para ITS utilizando os oligoiniciadores ITS1-ITS4 e ITS5-ITS4 para as vinte linhagens, e para o gene de calmodulina para as linhagens de Aspergillus. Foi utilizando o kit BigDye® Terminator v3.1 Cycle Sequencing e encaminhando a placa ao Laboratório Estratégico e analisada no equipamento 3500 Genetic Analyzer (Thermo Fisher). As sequencias foram analisadas no banco de dados GenBank, comparando com outras já depositadas
MALDI-TOF
MALDI-TOF (Matrix Assisted Lazer Desorption/Ionization – Time Of Flight Mass Spectrometry) foi utilizado para a identificação dos gêneros Aspergillus, Fusarium e Curvularia. Os fungos foram incubados em caldo Saboraud (OxoidⓇ) por três dias em banho seco com agitação (LoccusⓇ, modelo B-HS) a 26°C e 500 RPM, para se obter micélio uniforme. Esse micélio foi submetido ao processo de extração de proteínas, o produto obtido na extração foi posto em um spot da placa de MALDI-TOF (1 µL), em seguida cobertos com 1 µL de matriz HCCA. Por fim a placa com as extrações foi inserida no equipamento MALDI Biotyper® (Bruker Daltonics inc). O equipamento analisa os picos da espectrometria de massa do micro-organismo e o compara com seu banco de dados interno, a similaridade encontrada é avaliada num escore de 0,00 à 3,00, sendo os valores superiores a 2,00 considerados como de alta confidencialidade.
Métodos de conservação
Para avaliar os métodos de preservação as linhagens de A. fumigatus e A. flavus foram utilizadas para a confecção de criotubos com miçangas, usando caldo Sauton como crioprotetor e para as ampolas de liofilização leite (skim milk ) com glutamato de sódio a 5%.
RESULTADOS E DISCUSSÃO
Das vinte linhagens avaliadas no trabalho, dezoito puderam ser identificadas por ao menos uma das metodologias aplicadas, a exceção foram duas linhagens de F. pedrosoi.
Das seis linhagens enviadas para sequenciamento do gene de calmudolina somente as linhagens Aspergillus fumigatus IAL 9712 e IAL 9714, puderam ser parcialmente identificadas como A. fumigatus pela sequência de CMD 6, com 100% de similaridade para ambas. Das quinze linhagens encaminhadas para o sequenciamento para região ITS, treze puderam ser sequenciadas. Seis foram confirmadas com o depósito de origem sendo elas F. oxysporum (IAL 9783), identificada como F. oxysporum com 99,82% de similaridade e F. solani (IAL 9782) identificada como F. petroliphilum / F. solani com 99,625 de similaridade, ambas com gene consenso (ITS5-ITS4), de acordo com a base de dados Mycobank, (https://www.mycobank.org/), F. petroliphilum é uma variação de F. solani; Fonsecaea pedrosoi IAL 9560 e IAL 9664 foram parcialmente identificadas com 95,49% e 95,49% de similaridade; F. pedrosoi (IAL 9559) com 97,03% de similaridade (ITS 4); A
fumigatus (IAL 9777) identificado parcialmente pela sequencia com 100% de similaridade (ITS1). Para a confirmação dessas linhagens é preciso a utilização de novos alvos para o sequenciamento.
A linhagem de F. pedrosoi IAL 9558 apresentou resultados compatível com o gênero Phialophora sp para duas metodologias. As linhagens de Curvularia sp apresentaram como resultado duas possíveis espécies para cada amostra, uma identificada por sequenciamento do gene rDNA região ITS, outra linhagem foi parcialmente identificada, ambas tendo duas espécies como resultado, sendo diferenciadas por micromorfologia.
Todas as linhagens do gênero Aspergillus tiveram suas espécies identificadas com MALDI- TOF, correspondendo com a identificação prévia, todas com escore acima de 2,30. Para a linhagem de A. flavus o resultado foi Aspergillus oryzae group.
Já para as linhagens de Fusarium spp. todas foram identificadas como espécie (escores entre 2,13 e 2,25), contudo F. equisiti (IAL 9783) apresentou dois resultados divergentes da identificação original – F. oxysporum / F. verticilloides. A linhagem F. solani IAL 9782 apresentou como resultado F. petroliphilum, mesmo resultado do sequenciamento.
PAZIANI et. al. (2021) descreve em seu trabalho a dificuldade de identificação por MALDI- TOF de espécies de Fusarium, a diferença da expressão proteica em ambientes/meios de cultura diferentes de uma mesma espécie torna difícil sua identificação por esse método, em especial os membros do complexo Fusarium solani (FSSC). Com as linhagens de Curvularias sp. somente foi possível identificar o gênero da Amostra 2 com escore de 2,28, sem picos identificados para a Amostra 1.
Todas as linhagens apresentaram características macro e microscópicas como as descritas na literatura (WALSH; HAYDEN; LARONE et. al. 2018) e no banco de dados Mycobank. Contudo devido à grande similaridade entre algumas espécies somente essas características não são capazes de caracterizar a espécie de fungos filamentosos, sendo necessárias análises moleculares (ANTUNES 2019). A linhagem de F. pedrosoi IAL 9558 identificada pelo sequenciamento como Phialophora sp apresentou em sua micromorfologia conídios alongados terminados em forma triangular semelhante a uma taça, característico do gênero Phialophora.
Tanto o método de liofilização quanto de criopreservação mostraram-se eficazes para
Aspergillus fumigatus e A. flavus, não foi observado nenhum sinal de contaminação.
CONCLUSÃO
A identificação por sequenciamento da região ITS foi eficaz na identificação de espécies dos gêneros Fusarium, Fonsecaceae, Phialophora, Curvularia e para Aspergillus fumigatus. A. flavus IAL 9706 e A. terreus (sem IAL) não puderam ser identificados por sequenciamento para ITS assim como para gene da calmodulina, outros alvos são necessários para a caracterização completa dessas linhagens. A identificação por MALDI-TOF não foi capaz de caracterizar a linhagem A. flavus IAL 9706 como espécie; somente como grupo.
As espécies do gênero Aspergillus podem ser identificadas por MALDI-TOF. Para Curvularia a identificação somente foi possível em gênero, sendo necessário adaptar o método de extração de proteínas.
O gênero Fusarium trabalhado nesse estudo se mostrou mais difícil de identificar quanto suas espécies devido a grande similaridade dentre os membros de um mesmo complexo.
Somente a morfologia das linhagens não é capaz de caracterizar fungos em espécies, mas podem auxiliar na caracterização de gênero combinados com métodos de biologia molecular. É necessário autenticar toda linhagem depositada para confirmar as informações originais de depósito.
A liofilização com leite e glutamato de sódio a 5% e a criopreservação em miçangas usando caldo Sauton são eficazes na preservação de linhagens do gênero Aspergillus;
As metodologias de autenticação aplicadas se mostraram eficientes para a maioria das linhagens estudadas. Das 20 linhagens trabalhadas, 18 puderam ser identificadas por pelo menos um método proposto.
REFERENCIAS BIBLIOGRÁFICAS
ANTUNES, Fernanda Marques Americo. Avaliação da metodologia de espectrometria de massas MALDI-TOF (VITEK MS®) para identificação de espécies de Aspergillus de importância médica. 2019. Dissertação (Mestrado em Dermatologia) – Faculdade de Medicina, University of São Paulo, São Paulo, 2019. doi:10.11606/D.5.2019.tde-05062019-092951. Acesso em: 2022-11-23.
BOUNDY‐MILLS, K. et al. Preserving US microbe collections sparks future discoveries. Journal of Applied Microbiology, v. 129, n. 2, p. 162-174, 2020.
COSTA, Edmara Chaves et al. Princípios da estocagem e preservação de amostras microbiológicas. Ciência Animal, v. 19, n. 2, p. 111-122, 2009.
OLIVEIRA, Luciana Aires; CORTEZ, Ana Cláudia A.; SOUZA, João Vicente Braga de. Avaliação de metodologias de preservação para manutenção de microrganismos do Filo Ascomycota e leveduras do gênero Candida pertencentes à Coleção de Microrganismos de Interesse Médico do INPA. Diversidade Microbiana da Amazônia, pgs. 58-62, 2016.
PAZIANI, Mario Henrique. Perfil fenotípico e genotípico de Fusarium spp. provenientes de episódios clínicos de fusarioses no Estado de São Paulo. 2021. Tese (Doutorado em Bioagentes e Biotecnologia Aplicados à Farmácia) – Faculdade de Ciências Farmacêuticas de Ribeirão Preto, Universidade de São Paulo, Ribeirão Preto, 2021. doi:10.11606/T.60.2021.tde-29092021-053538. Acesso em: 2022-11-28.
PEREIRA, Anielli Souza. Diversidade e potencial para a produção de enzimas de Penicillium, Aspergillus, Talaromyces e Paecilomyces isolados de solo de Mata Atlântica do Quadrilátero Ferrífero. 2019. 101f. Tese (Doutorado em Microbiologia Agrícola) – Universidade Federal de Lavras, Programa de Pós-Graduação em Microbiologia Agrícola, Lavras, 2019.
SABINO, R. et al. Molecular epidemiology of Aspergillus collected from cystic fibrosis patients. Journal of Cystic Fibrosis, v. 14, n. 4, 2015.
SETTE, Lara Durães; PAGNOCCA, Fernando Carlos; RODRIGUES, André. Microbial culture collections as pillars for promoting fungal diversity, conservation and exploitation. Fungal Genetics and Biology, v. 60, p. 2-8, 2013.
WALSH, T. J.; HAYDEN, R. T.; LARONE, D. H. Larone’s Medically Important Fungi. 6o ed. Washington: [2018]
WHITE, Thomas J. et al. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. PCR protocols: a guide to methods and applications, v. 18, n. 1, p. 315-322, 1990.
Capítulo de livro publicado no Livro da IV Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
doi.org/10.53934/9786599965821-19
Este trabalho foi escrito por:
Vanessa da Silva Campos¹; Valéria Medina Camprigher2; José Eduardo Tolezano3 Virgínia Bodelão Richini-Pereira4
¹ Estudante do curso de Vigilância Laboratorial em Saúde Pública – Centro de Laboratórios Regionais II Bauru (CLR II Bauru); E-mail: [email protected]
2 Médica Veterinária da Secretaria Municipal de Saúde de Bauru-SP
3 Pesquisador do Centro de Parasitologia e Micologia do Instituto Adolfo Lutz São Paulo
4 Docente/Pesquisador do CLR II Bauru.
RESUMO
As leishmanioses são zoonoses causadas por protozoários pertencentes ao gênero Leishmania, estando entre as doenças negligenciadas em Saúde Pública. O objetivo deste trabalho foi avaliar o protocolo de PCR convencional (cPCR) para diagnóstico e acompanhamento de cães em tratamento para Leishmaniose Visceral Canina com miltefosina e alopurinol, comparando com o diagnóstico sorológico preconizado pelo Ministério da Saúde. Participaram 34 cães de quatro áreas do município de Bauru-SP, soropositivos no teste rápido imunocromatográfico (Teste Rápido de Plataforma de Duplo Percurso TR DPP®) e Ensaio Imunoenzimático ELISA. Destes animais, 44,11% foram positivos no exame molecular em pelo menos uma das amostras avaliadas (sangue, soro ou linfonodo). A cPCR foi realizada em três etapas, a primeira avaliação antes do início do tratamento (T0), e as demais avaliações após um mês (T1) e dois meses (T2) do início do tratamento. Das 184 amostras examinadas ao longo do tratamento, 76 (41,3%) foram de sangue, 75 (40,8%) de soro e 33 (17,9%) de linfonodo. No T0 a positividade no sangue foi de 32,35%, no soro de 11,76% e linfonodo 37,5%. No T1 a positividade no sangue foi de 17,86%, no soro 7,14% e no linfonodo 28,57%. No T2 a positividade no sangue foi de 7,14%, no soro não houve amostras positivas e no linfonodo apenas uma amostra positiva. O tratamento induziu melhora dos sinais clínicos e decaimento da positividade na cPCR, sugerindo efetividade do tratamento. A cPCR é um bom método complementar de confirmação do diagnóstico sorológicos e monitoramento do tratamento, contribuindo com as ações de vigilância e controle da LVC de forma mais assertiva e eficiente.
Palavras-chave: Diagnóstico Molecular, Leishmaniose, Reação em Cadeia da Polimerase, Saúde Única, Vigilância em Saúde Pública
INTRODUÇÃO
O conceito de Saúde Única (One Health) correlaciona a saúde humana, animal e ambiental com o objetivo de planejar e implementar programas, políticas, legislação e pesquisa por meio da comunicação de múltiplos setores, envolvendo profissionais de forma multissetorial e transdisciplinar com uma visão unificada para melhoria da saúde de todos os seres vivos e ambiente (RÜEGG, et al., 2018; CARNEIRO; BREWER, 2021).
Ainda pensando em Saúde Única e sua correlação com todos os seres vivos, como assunto de relevância em saúde pública temos as zoonoses, que de acordo com a Organização Mundial da Saúde (OMS) são doenças naturalmente transmitidas de animais vertebrados para seres humanos e vice-versa, dentre estas, de grande importância podemos destacar as leishmanioses, que são causadas por protozoários pertencentes à família Trypanosomatidae, do gênero Leishmania spp. que acomete tanto seres humanos quanto outros mamíferos. No ambiente urbano, o cão doméstico é o principal reservatório da Leishmaniose Visceral (LV) (BIDAISEE; MACPHERSON, 2014; ASOKAN; ASOKAN, 2015; GEBREYES et al., 2020; AKHOUNDI et al., 2016).
O Ministério da Saúde preconiza para o diagnóstico canino, o TR-DPP® (Dual Path Plataform, Biomanguinhos, Brasil) como triagem, e o confirmatório com o ensaio imunoenzimático (EIE, BioManguinhos, Brasil) (BRASIL, 2011), porém, a reação em Cadeia da Polimerase (PCR) vem sendo demonstrada como um exame diagnóstico complementar com boa sensibilidade e especificidade para a confirmação de casos (FERREIRA; MORAES, 2021). Em cães soropositivos, a recomendação do Programa Nacional de Vigilância e Controle de Leishmaniose Visceral, é a realização da eutanásia. Porém há uma demanda crescente por métodos alternativos com o uso de fármacos como medida auxiliar ao controle da doença.
Assim, o objetivo deste estudo foi avaliar a PCR convencional (cPCR) no diagnóstico complementar da LV canina, bem como o acompanhamento dos cães em tratamento para LV Canina no município de Bauru – SP.
MATERIAL E MÉTODOS
Foram avaliados 34 cães soropositivos, utilizando-se de sangue, soro e linfonodos. Os cães foram submetidos ao tratamento de LV, e os mesmos eram procedentes de quatro áreas mais críticas em relação à incidência da LV canina e humana no município. Como proteção ao ambiente, os animais receberam uma coleira impregnada por deltametrina 4% (Scalibor™/MSD), que está sendo trocada a cada 6 meses. O protocolo de tratamento iniciou de forma simultânea com a administração de Miltefosina a 2mg⁄ kg por via oral uma vez ao dia por 28 dias e alopurinol a 20 mg/kg por via oral duas vezes ao dia por 6 meses e depois a diminuição para 10 mg/kg com uso contínuo; associado a Domperidona a 0,5 – 1 mg/kg por via oral duas vezes ao dia durante 30 dias a cada 6 meses; além da Prednisona a 0,5 mg/kg por via oral duas vezes ao dia, quando necessário.
Para a realização do teste rápido das amostras de soro canino, foi utilizado o kit TR- DPP® para leishmaniose visceral canina (Bio-Manguinhos), seguido do teste confirmatório imunoenzimático EIE – Leishmaniose Visceral Canina (Bio-Manguinhos). A extração do DNA das amostras, foi realizada com o kit Extracta–DNA e RNA (Loccus®, Brasil), conforme instruções do fabricante. A PCR convencional foi realizada com os primers para Leishmania spp, da região kDNA, LINR4 e LIN19 (ARANSAY et al., 2000). O material amplificado foi visualizado em transluminador de luz Syngene® (DigiGenius, USA).
Os dados fazem parte de resultados parciais do Projeto de Doutorado de Valéria Medina Camprigher, “Uso da miltefosina como tratamento em cães sorologicamente positivos para Leishmaniose visceral no município de Bauru – São Paulo, aprovado pelo Conselho Técnico Científico (CTC) do Instituto Adolfo Lutz (nº59L-2019) e Comissão de Ética no Uso de Animais (CEUA) no 02/2020.
RESULTADOS E DISCUSSÃO
Os cães foram avaliados clinicamente e após confirmação laboratorial de LV pelos métodos diagnósticos preconizados pelo Ministério da Saúde, foi realizada a avaliação da cPCR, nos seguintes tempos: T0 – antes do início do tratamento, T1 – após um mês do início do tratamento e T2 – após dois meses do início do tratamento. Destes cães, 38,2% (13/34) eram fêmeas e 61,8% (21/34) machos. A maior parte dos animais não tinha raça definida 82,3% (28/34), o restante dos cães estava distribuído nas seguintes raças: Boxer, Chihuahua, Labrador, Pastor Alemão, Pitbull e Schnauzer. A tabela 1 mostra a distribuição dos animais de acordo com os sinais clínicos e demonstra que após o tratamento, houve regressão de alguns deles. Em estudo realizado em Florianópolis, também foi demonstrado a eficácia do tratamento de cães com Milteforan® (ROSAR, 2022) e em Andradina, houve diminuição do escore clínico durante as análises, porém 24% dos animais foram a óbito (LISBOA et al., 2018), sendo que nos nossos estudos, a porcentagem de óbitos foi de 8,8% e desistência no tratamento de 11,8%. A taxa de perda de animais ao longo de um período de análise é comum, sendo nossos índices considerados baixos se comparado com outros estudos (REITHINGER et al., 2004; LEITE et al., 2018).
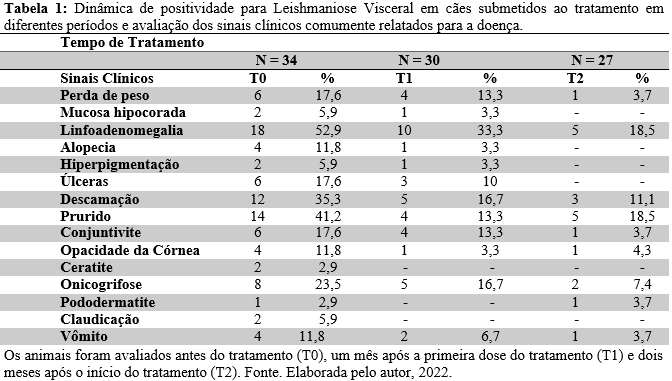
Dos 34 animais soropositivos, 44,11% também foram positivos na cPCR em pelo menos uma das amostras avaliadas (sangue, soro ou linfonodo). De acordo com Lopes et al. (2016) a cPCR é um método bastante eficiente, apresentando resultados melhores quando se utiliza mais de uma amostra do mesmo animal, isto porque algumas amostras biológicas são mais sensíveis do que as outras. Outro ponto importante a se destacar é que a associação entre duas amostras aumenta a sensibilidade do diagnóstico molecular da leishmaniose, assim como em nosso trabalho, em que se utilizou mais de uma amostra para realizar a PCR, o resultado foi confirmado como positivo, em ao menos uma dessas amostras.
Em áreas endêmicas, normalmente os exames sorológicos já apresentam sensibilidade maior, pois há uma grande exposição do cão ao parasita (MARTINEZ et al., 2011; LOPES et al., 2017). Em uma região endêmica de Pernambuco, os valores de positividade obtidos nos testes sorológicos foram 71,4% (127/178) com TR-DPP®, 70,2% (125/178) Exame Imunoenzimático (EIE) e 50,6% (90/178) com reação de (RIFI) e nos testes moleculares, a positividade foi de 20,2% (36/178) na cPCR e 38,8% (69/178) na qPCR (EVARISTO et al., 2021). Já, em um trabalho na mesma região do presente estudo, ao realizar os ensaios moleculares em amostras TR-DPP® positivas, foi identificado 63,9% amostras positivas para cPCR e 65% para qPCR (RAMOS-AUGUSTO, 2018),
Alguns fatores podem interferir na PCR, entre eles a presença de inibidores, fatores relacionados aos primers e ao protocolo de extração do DNA, bem como a sensibilidade da cPCR pode depender do tipo de amostra avaliada (SILVA et al., 2017). Assim, foram analisadas amostras de linfonodo, sangue e soro nos diferentes tempos: T0, T1 e T2. A tabela 2 demonstra a distribuição da positividade na cPCR. Importante salientar, que não foi realizada a coleta de linfonodo de todos os animais, pois alguns não apresentavam os mesmos aumentados. Além disso, no T1 e T2, não foi possível coletar amostras de alguns animais, e desta forma o número de amostras foi menor. No T2, apesar da positividade da amostra de linfonodo ser de 50%, isso não indica que houve elevação da positividade e sim, diminuição do número amostral final.

Nossos resultados demonstraram uma maior positividade em amostras de linfonodo, corroborando com dados da literatura que indicam uma melhor sensibilidade nessas amostras (PALTRINIERI et al., 2016; MARCELINO et al., 2020). Monteiro (2014), ao utilizar amostras de linfonodo de 58 cães para realização da PCR, demonstrou sensibilidade alta, identificando o DNA de Leishmania spp. em 47 (81%) dos cães, sendo 35 sintomáticos e 12 assintomáticos. De acordo com Travi et al. (2018), cães com sintomatologia clínica apresentam uma maior carga parasitária, facilitando sua identificação tanto em amostras de sangue quanto de medula óssea, contudo nos casos em que os cães são assintomáticos, o material biológico a ser analisado deve ser o mais sensível, preferindo amostras de medula óssea e de linfonodo.
A identificação de animais positivos não é tão sensível quando se utiliza o sangue periférico, isso porque o nível de parasitemia no momento da coleta é algo incerto, podendo resultar em falso negativo (BRAGA et al., 2014; PIETRO et al., 2020). Além disso, outro fator a ser levado em consideração para analisar a diferença entre os exames sorológicos e moleculares é o estadiamento clínico da leishmaniose visceral canina (JERICÓ et al., 2019). Nos nossos estudos, a positividade na cPCR, utilizando amostra de sangue foi de 32,35% e isso pode estar relacionada aos animais possuirem uma carga parasitária baixa, não sendo possível sua detecção. Embora a cPCR tenha se mostrado eficaz na detecção de Leishmania spp., seus resultados são determinados apenas em dois valores possíveis, positivo ou negativo e desta forma, a PCR em tempo real quantitativo (qPCR) pode ser mais sensível, permitindo a avaliação da carga parasitária pela avaliação da quantidade de DNA específico na amostra avaliada (STRAUSS-AYALI et al., 2004; DA SILVA, et al., 2010). Estudos demonstraram que amostras de sangue podem ser uma alternativa de diagnóstico e/ou complemento para os exames sorológicos, devido a capacidade do qPCR em detectar menor carga parasitária e por ser mais fácil de se obter em grande escala (FRANCINO et al., 2006; MAIA et al., 2009).
Assim, o trabalho demonstrou que a cPCR é um bom método diagnóstico complementar a ser utilizado no monitoramento de cães positivos, confirmando os diagnósticos sorológicos de leishmaniose e para o monitoramento da carga parasitária ao longo do tratamento, será fundamental a inclusão de exames de qPCR, a fim de avaliar a carga parasitária nesses animais e contribuir com as ações de vigilância e controle da Leishmaniose Visceral Canina de forma mais assertiva e eficiente.
CONCLUSÕES
O tratamento foi capaz de induzir uma melhora dos sinais clínicos, com manutenção ou melhora no estadiamento clínico, sugerindo efetividade no tratamento de cães com LVC, mesmo não havendo cura parasitológica total. As técnicas moleculares podem ser utilizadas como ferramentas complementares ao diagnóstico de LVC, sendo que a utilização de mais de uma amostra biológica por animal, melhora a sensibilidade diagnóstica.
AGRADECIMENTOS
Ao Centro de Controle de Zoonoses de Bauru-SP pelas amostras caninas e à FAPESP (2018/25889-4), pelo apoio financeiro ao projeto.
REFERÊNCIAS
AKHOUNDI, M. et al. A Historical Overview of the Classification, Evolution, and Dispersion of Leishmania Parasites and Sandflies. PLOS Negl. Trop. Dis., v.10, n.3, p.e0004349, 2016.
ARANSAY, A. M.; SCOULICA, E.; TSELENTIS, Y. Detection and identification of Leishmania DNA within Naturally infected Sand Flies by Seminested PCR on Minicircle Kinetoplastic DNA. Appl. Environ. Microbiol., v.66, n.5, p. 1933-1938, 2000.
ASOKAN G.V.; ASOKAN V. Bradford Hill’s criteria, emerging zoonoses, and One Health. J. Epidemiol. Glob. Health. v.6, n.3, p.125-129, 2015.
BIDAISEE, S.; MACPHERSON, C. N. L. Zoonoses and One Health: A Review of the Literature. J. Parasitol. Res., v.2014, 2014.
BRAGA, A. R. C.; LANGONI, H.; LUCHEIS, S. B. Evaluation of canine and feline leishmaniasis by the association of blood culture, immunofluorescent antibody test and polymerase chain reaction. J.Venom. Anim. Toxins Incl. Trop. Dis. Botucatu, v.20, p.1-7, 2014.
BRASIL. Ministério da Saúde (MS). Secretaria de Vigilância em Saúde, Departamento de Vigilância das Doenças Transmissíveis. Nota Técnica conjunta nº1, de 29 de dezembro de 2011. Esclarecimento sobre substituição do protocolo diagnóstico da leishmaniose visceral canina (LVC). Brasília: Ministério da Saúde; 2011.
CARNEIRO, L. A.; BREWER, C. P. One Health: Conceito, história e questões relacionadas – Revisão e reflexão. Cientifica Digital, v.1, p. 219-240, 2021.
DA SILVA, R. N. et al. Real-time PCR in clinical practice: a powerful tool for evaluating Leishmania chagasi loads in naturally infected dogs. Ann. Trop. Med. Parasitol., v.104, n.2, p.137-143, 2010.
EVARISTO, A. M. C. F. et al. Comparison of serological and molecular tests to investigate Leishmania spp. infections in stray dogs from an area of intense visceral leishmaniasis transmission in Brazil. Rev. Bras. Parasitol. Vet,. v.30, n.3, p.e006621
FERREIRA, A. W.; MORAES, S. L. Diagnóstico Laboratorial das Principais Doenças Infecciosas e Autoimunes. 3. ed. Rio de Janeiro: Guanabara Koogan, 2021.
FRANCINO, O. et al. Advantages of real-time PCR assay for diagnosis and monitoring of canine leishmaniosis. Vet. Parasitol., v.137, n.3-4, p.214-221, 2006.
GEBREYES, W. A. et al. Molecular Epidemiology of Infectious Zoonotic and Livestock Diseases:
Microbiol. Spectr., v.8, n.2, p.8.2.2.
JERICÓ, M. M.; NETO, P. A.; KOGIKA, M. M. Tratado de Medicina Interna de Cães e Gatos. 1. ed. Rio de Janeiro: Roca, 2019.
LEITE, B.M.M. et al. The mass use of deltamethrin collars to control and prevent canine visceral leishmaniasis: A field effectiveness study in a highly endemic area. PLOS Negl. Trop. Dis., v.12, n.5, p.e006496, 2018.
LISBOA, J. C. L. et al. Acompanhamento clínico e laboratorial de cães parasitologicamente positivos para leishmaniose visceral submetidos à terapia com miltefosina associada ao alopurinol. Rev. M&Z, v.16, n.3, p.79-80. 2018.
LOPES E.G. et al. Serological and molecular diagnostic tests for canine visceral leishmaniasis in Brazilian endemic area: one out of five seronegative dogs are infected. Epidemiol. Infect., v.145, n.12, p.2436-2444, 2017.
LOPES, E. G. et al. Performance of conventional PCRs based on primers directed to nuclear and mitochondrial genes for the detection and identification of Leishmania spp. Rev. Inst. Med. Trop. São Paulo, v.58, n.41, p.1-7, 2016.
MAIA, C. et al. Diagnosis of canine leishmaniasis: conventional and molecular techniques using different tissues. Vet. J., v.179, p.142-144, 2009.
MARCELINO, A. P. et al. Comparative PCR-based diagnosis for the detection of Leishmania infantum in naturally infected dogs. Acta Trop., v.207, p.105495, 2020.
MARTINEZ, V. et al. Canine leishmaniasis: the key points for qPCR result interpretation. Parasit. Vectors, v.4, n.1, p.1-5, 2011.
MONTEIRO, A. G. Diagnóstico molecular e identificação das espécies de Leishmania na leishmaniose visceral canina no Distrito Federal, Brasil. 2014 33f. Dissertação (Mestre em Biologia Microbiana da Universidade de Brasília) – Universidade de Brasília, Brasília, 2014.
PALTRINIERI, S. et al. Laboratory tests for diagnosing and monitoring canine leishmaniasis. Vet. Clin. Pathol., v.45, n.4, p.552-578, 2016.
PIETRO, S. D. et al. Parasitemia and its variation in canine in canine leishmaniasis. Parasitol. Res., v.119, n.10, p.3541-3148, 2020.
RAMOS-AUGUSTO, L. C. Estudo de Leishmania spp. em amostras provenientes de cães e humanos e suas implicações em saúde pública na região de Bauru, São Paulo. 2018. 114f. Dissertação (Mestrado em Doenças Tropicais) – Universidade Estadual Paulista, Botucatu, 2018.
REITHINGER, R. et al. Are insecticide impregnated dog collars a feasible alternative to dog culling as a strategy for controlling canine visceral leishmaniasis in Brazil?. Int. J. Parasitol., v.34, n.1, p.55-62, 2004.
READY, P. D. Biology of phlebotomine sand flies as vectors of disease agents. Ann. Rev. Entomol.
v.58, p.227-250, 2013.
ROSAR, A. S. Estudo da eficácia do Milteforan® no tratamento da leishmaniose visceral canina na região da grande Florianópolis, SC. 2022. 115f. Dissertação (Mestre em Biotecnologia e Biociênciais) – Universidade Federal de Santa Catarina, Florianópolis, 2022.
RÜEGG, S. R.; HASLER, B.; ZINSSTAG, J. Integrated Approaches to Health: A Handbook for the Evaluation of One Health. Wageningen Academic Publishers, Wageningen, Netherlands. 2018.
SILVA, R. C. et al. Detection of Leishmania (L.) infantum in stray dogs by molecular techniques with sensitive species-specific primers. Vet. Q., v.37, n.1, p.23-30, 2017.
STRAUSS-AYALI, D. et al. Polimerase chain reaction using noninvasively obtained samples, for the detection of Leishmania infantum DNA in dogs. J. Infect. Dis., v.189, n.9, p.1729-1733, 2004.
TRAVI, B. L. et al. Canine visceral leishmaniasis: Diagnosis and management of the reservoir living amoung us. PLOS Negl. Trop. Dis., v.12, n.1, p.e0006082, 2018.
Capítulo de livro publicado no Livro da IV Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
doi.org/10.53934/9786599965821-18
Este trabalho foi escrito por:
Amanda Vieira Lima1; Julia Gabriela dos Santos Pedro¹; Clóvis Roberto Abe Constantino²; Adriana Parise Compri²
1 Estudante do Curso de Vigilância Laboratorial em Saúde Pública do Laboratório de Hepatites do Instituto Adolfo Lutz Central; E-mail: [email protected]
2 Pesquisador Científico do Laboratório de Hepatites do Instituto Adolfo Lutz Central; E-mail: [email protected]
RESUMO
A infecção pelo vírus da hepatite C é um grave problema de saúde pública, sendo que 85% dos indivíduos infectados progridem para a fase crônica. Além disso, muitos pacientes são assintomáticos, dificultando o diagnóstico e levando a doença a avançar durante décadas sem suspeita clínica, o que aumenta o risco de complicações como cirrose e carcinoma hepatocelular. O diagnóstico envolve o uso de técnicas sorológicas para a triagem e técnicas de biologia molecular como o RT-qPCR, para a confirmação do resultado. O RT-qPCR faz a quantificação do vírus HCV e, por isso, é usado não apenas para o diagnóstico, mas também para medir a eficácia do tratamento. O objetivo do presente trabalho foi avaliar a rotina dos exames de RT-qPCR para o diagnóstico e tratamento da hepatite C. Para tanto, foram selecionados os exames realizados entre os meses de janeiro a julho de 2022 no Laboratório de Hepatites do Instituto Adolfo Lutz Central. O levantamento de dados foi feito por meio do Gerenciador de Ambiente Laboratorial (GAL) e das requisições de solicitação de exames dos pacientes. Os resultados mostraram que a maioria dos infectados eram do sexo masculino 55% com média de idade de 53,2 anos. Para o diagnóstico, 57,7% dos exames apresentaram resultado não detectável, evidenciando a importância do RT-qPCR para confirmar a infecção ativa. Nos exames para avaliação da doença após o tratamento, 96,7% foram não detectáveis indicando que os medicamentos administrados foram eficazes. Em relação à rotina, falhas foram encontradas sendo propostas medidas para a sua melhoria, o que impacta diretamente nos resultados liberados aos pacientes atendidos pela rede. Portanto, com as ferramentas utilizadas foi possível avaliar a rotina que envolve o diagnóstico e tratamento da hepatite C no Laboratório de Hepatites do Instituto Adolfo Lutz Central e propor ações para reduzir as não conformidades.
Palavras-chave: Diagnóstico, Hepatite C, Rotina, RT-qPCR, Tratamento
INTRODUÇÃO
A hepatite C é responsável por aproximadamente 71 milhões de infecções no mundo, sendo 700 mil no Brasil, representando uma das principais doenças crônicas hepáticas, o que levou a Organização Mundial da Saúde (OMS) a propor que até 2030 houvesse um esforço global para reduzir a incidência e mortalidade desse agravo.(1, 2, 3)
O vírus da hepatite C (HCV) é transmitido através do sangue contaminado que está presente principalmente nos objetos perfurocortantes, e por esse motivo, usuários de drogas
injetáveis e profissionais da área da saúde estão entre os grupos mais suscetíveis. Cerca de 85% dos infectados não conseguem eliminar espontaneamente o vírus e a doença evolui para o estado crônico que é caracterizado pela persistência viral por mais de 6 meses. Somado a isso, muitos pacientes progridem de maneira assintomática, dificultando o diagnóstico e levando a doença a avançar durante décadas sem suspeita clínica, aumentando o risco de complicações como cirrose e carcinoma hepatocelular.(4, 3)
O diagnóstico envolve o uso de técnicas sorológicas para a triagem e técnicas de biologia molecular como a transcrição reversa seguida da reação em cadeia da polimerase em tempo real (RT-qPCR) para a confirmação do resultado. A RT-qPCR faz a quantificação do vírus HCV e por isso é usada não apenas para o diagnóstico, mas também para medir a eficácia do tratamento.(5)
No estado de São Paulo o Laboratório de Hepatites do Instituto Adolfo Lutz (IAL) Central é o laboratório de referência e coordenador da rede de biologia molecular, que além de realizar as rotinas de PCR quantitativo em tempo real para as hepatites B e C atua auxiliando outros laboratórios da rede.(6)
Tendo em vista os aspectos envolvendo a hepatite C e a importância dos exames de biologia molecular para o monitoramento dessa doença, o presente trabalho teve como objetivo avaliar a rotina dos exames de RT-qPCR para o diagnóstico e tratamento da hepatite C.
MATERIAL E MÉTODOS
Para que o objetivo pudesse ser atingido foi realizado um levantamento de dados secundários dos pacientes com suspeita ou confirmação de hepatite C, atendidos entre os meses de janeiro a julho de 2022 no Laboratório de Hepatites do IAL Central. As informações foram retiradas do Gerenciador de Ambiente Laboratorial (GAL) e das requisições de exames dos pacientes, que foram encaminhadas junto com as amostras, levando em consideração parâmetros que possibilitassem conhecer o perfil dos pacientes atendidos pela rede, como sexo e idade, além dos motivos para as solicitações dos exames e as ocorrências que causaram o cancelamento de parte deles. As tabelas de resultados foram geradas através do programa Microsoft Excel 2010.
RESULTADOS E DISCUSSÃO
Durante o período analisado 1307 exames de biologia molecular do tipo RT-qPCR foram solicitados ao Laboratório de Hepatites do IAL Central. Os motivos assinalados na requisição para justificar a realização do exame são apresentados conforme a Tabela 1. O campo marcado como vazio 19,9% representa exames que foram cadastrados no GAL, mas o resultado não foi liberado. O campo ignorado 12,9% representa requerimentos em que o motivo do exame não foi preenchido pela unidade solicitante. A falta ou o incorreto preenchimento desse campo pode levar a incompatibilidade entre o motivo e o valor da carga viral prejudicando a análise do resultado.
Como já mencionado, uma parcela dos exames não foram liberados ficando retidos nos status de aguardando triagem (3,1%), exames cancelados (7,7%) e exames não realizados (89,2%). As ocorrências que levaram aos seus cancelamentos são apresentadas na Tabela 2. Nota-se que muitas das ocorrências são devido a falhas na etapa pré-analítica, por exemplo, o envio da amostra para o local errado de realização do exame, como em “O IAL Central não é referência para esta solicitação” 19,6% fazendo com que a solicitação não esteja em conformidade com o que é determinado pelo Ministério da Saúde (MS) ocasionando desperdício de tempo e mão de obra. Além disso, quase metade dos cancelamentos 46,2% não teve um motivo registrado, o que prejudica a adoção de medidas para evitar falhas no futuro. Vale ressaltar que, dentre os exames não liberados, nos exames cancelados e aguardando triagem o sistema GAL não permite colocar o motivo do cancelamento, só sendo permitido para os exames com status não realizados.

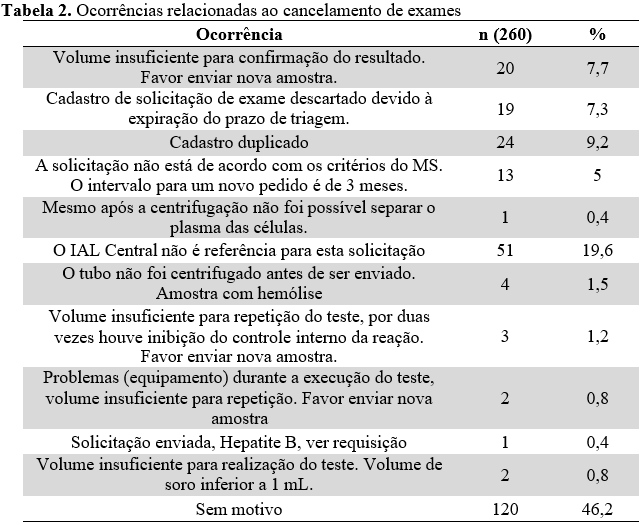
Do total de solicitações, 1047 exames foram liberados 80,1% sendo que destes foram considerados abaixo do limite de quantificação 0,4% (n=4), detectáveis 21,8% (n=229) e não detectáveis 77,7% (n=814). A maioria dos resultados detectáveis foram em pacientes do sexo masculino 55,0% (n=126), semelhante à proporção relatada no Boletim Epidemiológico do MS (2021), onde os homens representaram 57,6% dos infectados entre os anos de 1999 a 2020.(7)
Em relação à idade, a maioria dos resultados detectáveis 31,4% (n=72) foi na faixa etária de 40 a 49 anos e média de 53,2 anos, estando de acordo com Carvalho-Louro et al. (2020) que menciona uma maior prevalência de HCV depois dos 40 anos e sugere a intensificação de campanhas para o diagnóstico a partir dessa idade.(1)
Das requisições que tiveram como motivo o diagnóstico (Tabela 3) mais da metade deles foi não detectável 57,7%, pois os anticorpos detectados no teste rápido (TR) são anticorpos totais não diferenciando infecção ativa de passada, isto mostra a importância do exame de RT-qPCR para confirmação do diagnóstico. Dentre os detectáveis, a maioria apresentou log alto com média de 5,7 indicando a necessidade de se iniciar o tratamento o quanto antes a fim de evitar as complicações clínicas e a disseminação do vírus, como demonstrado na literatura.(5, 8).

No decorrer do ano de 2022, casos de hepatite aguda grave de etiologia desconhecida vêm acometendo crianças. Diante disso, o IAL Central enquanto laboratório de referência recebeu amostras de hepatite de causa desconhecida para a investigação do HCV. Das 30 amostras analisadas, todas apresentaram resultado não detectável, confirmando que o HCV não é o agente etiológico desse agravo como observado em outros países.(9, 10)
Quanto ao motivo de solicitação para genotipagem, os resultados são em sua maioria detectáveis 88,9% (n=8) e com valores de carga viral alta, o que é esperado já que o recomendado é que a genotipagem seja feita antes do início do tratamento. No momento da solicitação, a requisição do paciente foi preenchida tendo como motivo a genotipagem, mas como o Laboratório de Hepatites do IAL Central não é referência para a solicitação deste exame, somente o RT-qPCR foi realizado.
O rastreio periódico do HCV é recomendado principalmente entre os grupos mais expostos aos fatores de risco considerando a possibilidade de reinfecção já que a hepatite C não confere imunidade protetora após a primeira exposição(2). Dentre as 11 amostras analisadas tendo como finalidade a suspeita de reinfecção, 18,2% (n=2) apresentaram resultado detectável.
A maioria dos resultados foi para verificar a avaliação pós-tratamento (Tabela 4), para averiguar se a resposta virológica sustentada (RVS) foi alcançada. Destes, 96,7% foram não detectáveis, evidenciado a eficácia do tratamento. Dos 2,6% que foram detectáveis todos apresentaram valor de carga viral alta com log acima de 4. Dentre as explicações prováveis está, a de que, parte dos pacientes não aderiram ao tratamento, não responderam ao tratamento indicado ou a finalidade do exame estava incorreta quando preenchida pela unidade requisitante.(1, 11)
Exames de RT-qPCR também foram feitos para os pacientes que precisavam monitorar o tratamento, com o intuito de saber se a carga viral estava diminuindo ao longo do tempo. Destes, 92,9% (n=13) apresentaram resultado não detectável, mostrando que o tratamento estava funcionando como mencionado por Duarte et al (2021).(12)
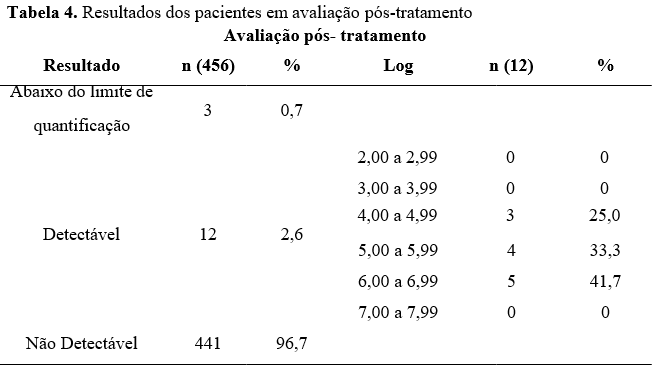
Para os casos em que o motivo foi a indicação de tratamento, 67,9% (n=19) dos pacientes apresentaram resultado detectável. A indicação de tratamento é importante para a obtenção de um valor de carga viral atualizado uma vez que o paciente só consegue retirar o medicamento se apresentar um resultado de carga viral de até 3 meses como definido pelo MS.(2)
Através das requisições também foi possível verificar quais pacientes fizeram uso de medicamentos considerando a somatória dos que estavam em avaliação pós-tratamento e monitorando o tratamento (n=470). Desse total, 13,0% das requisições estavam com o campo em branco e 70,0% não possuíam o campo de medicação para ser preenchido em decorrência do modelo de ficha mais recente. Dada a importância dessa informação para acompanhar a evolução clínica do paciente seria interessante a adaptação do modelo atual com a inclusão desse campo.
Portanto, é fundamental o comprometimento de todos os profissionais envolvidos e a possibilidade de treinamento e/ou reciclagem, bem como a revisão periódica dos protocolos visando o aprimoramento das etapas da rotina para a correta realização do exame.
CONCLUSÃO
O desenvolvimento desse estudo possibilitou conhecer o perfil dos pacientes atendidos, identificar parâmetros para melhorar e aumentar a qualidade dos exames de rotina de RT-qPCR para HCV realizados pelo Laboratório de Hepatites do IAL Central e pela rede estadual, que coordena.
REFERÊNCIAS
- CARVALHO-LOURO, D. M. et al. Hepatitis C screening, diagnosis, and cascade of care among people aged > 40 years in Brasilia, Brazil. BMC Infectious Diseases, v. 20, n. 1, 10 fev. 2020.
- MS – MINISTÉRIO DA SAÚDE. Secretaria de Vigilância em Saúde. Departamento de Vigilância, Prevenção e Controle das Infecções Sexualmente Transmissíveis, do HIV/Aids e das Hepatites Virais. Protocolo Clínico e Diretrizes Terapêuticas para Hepatite C e Coinfecções. Brasília: Ministério da Saúde; 2019.
- ABU-FREHA, N. et al. Chronic hepatitis C: Diagnosis and treatment made easy. European Journal of General Practice, v. 28, n. 1, p. 102–108, 31 dez. 2022.
- MS – MINISTÉRIO DA SAÚDE. Secretaria de Vigilância em Saúde. Departamento de Vigilância, Prevenção e Controle das Infecções Sexualmente Transmissíveis, do HIV/Aids e das Hepatites Virais. Manual Técnico para o Diagnóstico das Hepatites Virais. Brasília: Ministério da Saúde, 2018.
- . Secretaria de Vigilância em Saúde, Departamento de DST, Aids e Hepatites Virais. Protocolo Clínico e Diretrizes Terapêuticas para Hepatite C e Coinfecções. Brasília: Ministério da Saúde, 2018.
- LEMOS, M. F. et al. Diagnóstico Molecular da Hepatite C dos pacientes atendidos pelo Instituto Adolfo Lutz Central, entre março de 2012 e março de 2013. Boletim Epidemiológico Paulista – BEPA. V. 12 No 139. Julho de 2015.
- MS – MINISTÉRIO DA SAÚDE. Secretaria de Vigilância em Saúde. Departamento de Vigilância, Prevenção e Controle das Infecções Sexualmente Transmissíveis, do HIV/Aids e das Hepatites Virais. Boletim Epidemiológico – Hepatites Virais 2021. Brasília: Ministério da Saúde, No. especial jul. 2021.
- BATTISTAM, D. C. Avaliação da Carga Viral do Vírus da Hepatite C (VHC) no Plasma Pobre em Plaquetas. Dissertação (Mestrado em Pesquisa e Desenvolvimento: Biotecnologia Médica. Faculdade de Medicina, Universidade Estadual Paulista (UNESP). Botucatu – SP. 2019.
- MARSH, K. et al. Investigation into cases of hepatitis of unknown aetiology among young children, Scotland, 1 January 2022 to 12 April 2022. Eurosurveillance, v. 27, n. 15, p. 1–7, 14 abr. 2022.
- VIDAL, A. R. et al. Hepatitis of unknown aetiology in children – epidemiological overview of cases reported in Europe, 1 January to 16 June 2022. Eurosurveillance. European Centre for Disease Prevention and Control (ECDC), v. 27, n. 31. 4 ago. 2022.
- ROCHA, L. S. Avaliação das mutações de resistência ao tratamento com os novos antivirais de ação direta (DAA) em pacientes com hepatite C crônica. 2019. Dissertação (Mestrado em Biotecnologia em Saúde e Medicina Investigativa). Fundação Oswaldo Cruz, Salvador, Bahia, 2019.
- DUARTE, G. et al. Protocolo Brasileiro para Infecções Sexualmente Transmissíveis 2020: hepatites virais. Epidemiologia e Serviços de Saúde, v. 30, n. especial 1, 2021.
Capítulo de livro publicado no Livro da IV Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
doi.org/10.53934/9786599965821-17
Este trabalho foi escrito por:
José Eduardo Gonçalves Sbroggio Pereira1; Marcia Maria Costa Nunes Soares2; Fernanda Modesto Tolentino2; Milena Polotto de Santi2
1Estudante do Curso de Vigilância Laboratorial em Saúde Pública – CLR – IAL – SJRP; E-mail: [email protected]
2Docente/pesquisadora do Núcleo de Ciências Biomédicas – CLR – IAL – SJRP
Resumo: As retroviroses humanas emergiram como pandemias nas últimas décadas e são importantes causas de morbidade e mortalidade humana. Dentre elas, estão os vírus linfotrópicos de células T humana do tipo 1 e do tipo 2 (HTLV-1 e 2) que foram os primeiros retrovírus humanos isolados e identificados, e o HIV (vírus da imunodeficiência adquirida). As principais vias de transmissão desses vírus são a vertical, sexual e por componentes sanguíneos contaminados. Sendo assim, o objetivo deste estudo foi investigar a existência de coinfecções HTLV com HIV em pacientes atendidos em unidades pertencentes à divisão regional de saúde XV do estado de São Paulo. Para isso, foi realizada uma busca e análise dos resultados de HTLV e HIV nos bancos de dados Siscel, Sigh e GAL. A análise dos dados mostrou que 28 pacientes apresentaram coinfecção HTLV-HIV, sendo 60,7% dos soropositivos para HIV coinfectados com HTLV-1 e 39,3% coinfectados com HTLV-2. Dos coinfectados, 26% apresentaram idade entre 38 e 48 anos e 74% idade igual ou superior a 49 anos; 43% dos pacientes eram do sexo feminino e 57% do sexo masculino. Os municípios que apresentaram maior número de pacientes coinfectados foram Catanduva e São José do Rio Preto. Grande parte dos pacientes foram provenientes de serviços especializados em HIV/AIDS (Coordenadoria DST/AIDS, Serviço De Atendimento Especializado Saecrt HIV/AIDS (SAE) e alas específicas de infectologia dos hospitais). Estes achados evidenciam a importância do rastreamento de ambos os vírus, principalmente em pacientes de grupos de risco para HTLV como usuários de drogas injetáveis com compartilhamento de seringas.
Palavras-Chaves: Coinfecção, Infecções por HTLV 1, Infecções por HTLV-2, HIV.
Introdução
As retroviroses humanas emergiram como pandemias nas últimas décadas e são importantes causas de morbidade e mortalidade humana. Dentre elas, estão os vírus linfotrópicos de células T humana do tipo 1 e do tipo 2 (HTLV-1 e 2) e o HIV (vírus da imunodeficiência adquirida). O HTLV-1 e HTLV-2 foram os primeiros retrovírus humanos isolados e identificados e podem persistir no hospedeiro, de forma silenciosa, durante vários anos ou décadas (MONTANHEIRO, 2007).
As principais áreas endêmicas para a infecção pelo vírus HTLV-1 são sudoeste do Japão, algumas áreas do Caribe e redondezas, América do Sul com foco na Colômbia e Guiana Francesa, algumas áreas da África intertropical e Oriente Médio e regiões isoladas na Austrália (GESSAIN e CASSAR, 2012). O número de infectados no mundo é entre 10 a 20 milhões, e, no Brasil, estima-se que existam cerca de dois milhões de pessoas infectadas com HTLV-1, e dessas, aproximadamente 90% permanecerão assintomáticas ao longo de suas vidas. Em bancos de sangue em algumas regiões do país os infectados correspondem a até 2% (EDLICH et al., 2000).
O HTLV-2 é endêmico em populações indígenas isoladas em tribos de pigmeus na África e tem se disseminado entre usuários de drogas intravenosas e seus parceiros sexuais na América do Norte e Europa (Itália, Espanha, França, Noruega, Suécia, Irlanda e Reino Unido) (LOWIS et al., 2002). A presença do HTLV-2 entre índios americanos é antiga, e teria sido introduzida em áreas urbanas das Américas, Europa e Ásia, principalmente entre usuários de drogas intravenosas, transmitido principalmente por exposição ao sangue contaminado, resultante do compartilhamento de agulhas e seringas (HALL et al., 1994). O HTLV-2 predomina na população nativa do Brasil (CATALAN-SOARES e CARNEIRO- PROIETTI, 2005).
O HIV (vírus da imunodeficiência humana) é um retrovírus, classificado na subfamília dos Lentiviridae. É o causador da AIDS, uma doença que ataca o sistema imunológico, cujas células alvo são os linfócitos T CD4+. O ciclo de infecção ocorre através da alteração do DNA dessa célula, onde o HIV multiplica-se. Após o processo de multiplicação, os linfócitos são rompidos e os novos vírus continuam o ciclo. A transmissão pode ocorrer através de relação sexual sem camisinha, compartilhamento de seringas, transmissão vertical, transfusão de sangue contaminado, instrumentos perfurocortantes (Ministério da Saúde, 2018).
A principal via de transmissão do HTLV é a transmissão vertical, principalmente relacionada à amamentação (HINO, 2011). A transmissão sexual, principalmente, não exclusivamente, ocorre do homem para a mulher e supõe se que isto seria a explicação para o aumento da soroprevalência com a idade em mulheres (MURPHY et al., 1989; STUVER et al., 1993; TAKEZAKI et al., 1995; KAPLAN et al., 1996; ROUCOUX et al., 2005). A
transmissão por componentes sanguíneos contaminados (contendo linfócitos infectados com HTLV) é responsável por uma alta proporção (15-60%) de infecção em receptores de sangue (OKOCHI et al., 1984). A infecção por HTLV-1 está presente também entre usuários de drogas intravenosas, mas em extensão menor que o HTLV-2 (MURPHY et al., 1999). A relação sexual desprotegida, o contato com sangue ou tecido e a amamentação podem expor os indivíduos não apenas ao HTLV, mas a uma variedade de outros patógenos, como vírus da imunodeficiência humana (HIV), Treponema pallidum, vírus da hepatite C (HCV), vírus da hepatite B (HBV), vírus do papiloma humano (HPV) e vírus do herpes simples (HSV) (ROSADAS et al, 2022). O HTLV e o HIV possuem similares vias de transmissão, o que favorece a coinfecção (PEREIRA, 2019). Sendo assim, o objetivo do presente estudo foi determinar a ocorrência de coinfecções HTLV-HIV em pacientes diagnosticados para HTLV no Instituto Adolfo Lutz de São José do Rio Preto e atendidos na abrangência da Divisão Regional de Saúde XV (DRS-XV). Os resultados são importantes para serem utilizados nas estratégias de profilaxia junto à população em geral e, principalmente, entre os grupos de risco.
Material e Métodos
O estudo foi realizado no CLR-IAL de São José do Rio Preto, onde é realizada o diagnóstico sorológico de HTLV dos pacientes de todos os municípios pertencentes à DRS- XV, distribuídos em sete colegiados (Santa Fé do Sul, Jales, Fernandópolis, Votuporanga, São José do Rio Preto, José Bonifácio e Catanduva). Trata-se de um estudo retrospectivo
com levantamento de banco de dados, no qual foi investigada a presença da coinfecção HTLV-HIV entre os pacientes que apresentaram sorologia reagente para HTLV no período de março de 2015 a dezembro de 2021.
Os dados foram obtidos nas plataformas SIGH (Sistema de Informação e Gestão Hospitalar), GAL (Gerenciador de Ambiente Laboratorial) e SISCEL (Sistema de Controle de Exames Laboratoriais da Rede Nacional de Contagem de Linfócitos e Carga Viral), e, posteriormente organizados em uma tabela no Microsoft Excel para serem analisados.
Resultados e Discussão
A análise dos dados mostrou que das 38 amostras, 28 pacientes apresentaram coinfecção HTLV-HIV 73,68% (28/38). Das 28 amostras com coinfecção, 17 (60,7%) coinfectados HTLV-1-HIV e 11 (39,3%) coinfectados HTLV-2-HIV, conforme descrito na Tabela 1.
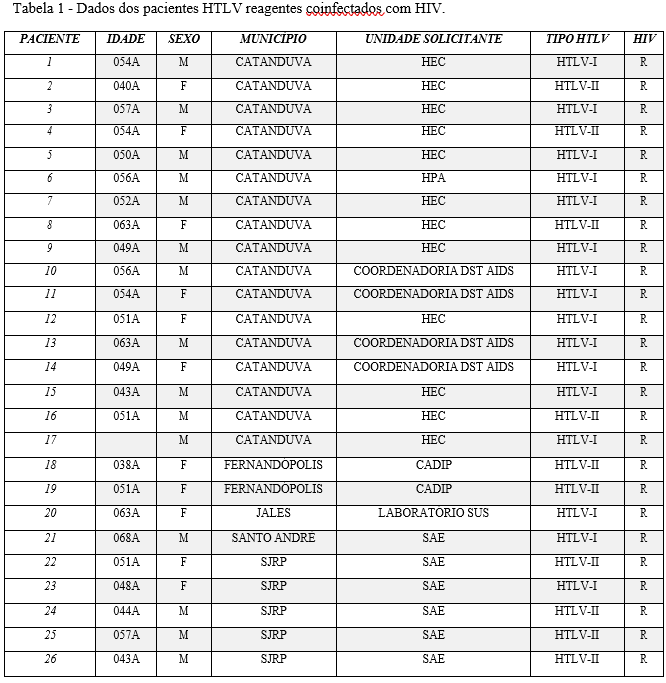

Os pacientes que apresentaram coinfecção foram provenientes dos municípios de Catanduva, Fernandópolis, Jales, Santo André e São José do Rio Preto. Os municípios com maior número de pacientes infectados foram os de Catanduva, com 60,7% (17/28) pacientes, e São José do Rio Preto com 21,4% (6/28) pacientes.
Analisando a faixa etária, foi observado que a idade mínima dos infectados foi de 38 anos e a máxima de 68 anos, sendo 26% (7/27) com idade variando de 38 a 48 anos e 74% (20/27) com idade igual e superior a 49 anos, isso pode ser um indicativo da demora na suspeita e consequente diagnóstico da infecção por HTLV. O diagnóstico em idade mais avançada nos coinfectados pode ser explicado devido ao curso assintomático, do vírus HTLV, sendo descobertos somente quando algumas das complicações crônicas são evidenciadas (PEREIRA et al, 2020). A implementação de estratégias de prevenção do HIV em meados da década de 1980 pode ter reduzido o risco de novas infecções por HTLV-I (Dourado et al, 2003). Segundo a Assembleia Legislativa do Estado de São Paulo, desde 1986, o Estado de São Paulo conta com leis que visam o controle do HIV, baseados na Lei 5.190/1986, que vem a estabelecer que todo o material para transfusão de sangue em hospitais e bancos de sangue seja testado para detecção da presença do vírus. A PORTARIA Nº 1.376, DE 19 DE NOVEMBRO DE 1993, inseriu a obrigatoriedade da realização dos testes sorológicos para as doenças de Chagas, Sífilis, Hepatite, SIDA/AIDS, HTLV I/II nos bancos de sangue. Segundo o Protocolo Clínico e Diretrizes Terapêuticas para Manejo da Infecção pelo HIV em Adultos (2013), todos os indivíduos infectados pelo HIV-1 devem ser testados para HTLV-1/2. Isso pode ter corroborado com a diminuição da transmissão, não só do HIV como do HTLV também, entre outras doenças. Além disso, pode ser um possível indicativo do fato da maioria dos pacientes serem de idade mais avançada, já que existe a coinfecção mesmo quando o HTLV encontra-se latente, podendo ter sido infectados em um período anterior à introdução dos testes sorológicos em bancos de sangue.
Em relação ao gênero, 43% (12/28) dos pacientes coinfectados foram do sexo feminino e 57% (16/28) foram do sexo masculino. A coinfecção se mostrou mais frequente em pacientes do gênero masculino em comparação ao gênero feminino assim como em estudo de Pereira e colaboradores publicado em 2020.
A maioria dos pacientes coinfectados com foram provenientes de serviços especializados em HIV/AIDS como Coordenadoria DST/AIDS, Serviço De Atendimento Especializado Saecrt HIV/AIDS (SAE) e alas específicas para doenças infecciosas em hospitais, mostrando que a coinfecção entre essas duas doenças é comum e ocorre de forma frequente (CATERINO-DE-ARAUJO, 2015). O estudo de Caterino-de-Araujo e colaboradores (2015) realizado na cidade de São Paulo, demonstrou que a coinfecção HTLV-HIV teve como maior fator de risco o compartilhamento de seringas por usuários de drogas injetáveis, e que, a maioria dos coinfectados descobriram a infecção por HIV entre 1990 e 1991, quando compartilhavam seringas e agulhas ao injetar drogas ilícitas ou mantinham relações sexuais com usuários de drogas injetáveis. Sendo assim, é de extrema importância o rastreio de ambos os vírus em pacientes pertencentes aos grupos de risco.
Conclusão
A análise dos dados mostrou que 28 pacientes apresentaram coinfecção HTLV-HIV, sendo 17 (60,7%) coinfectados com os vírus HTLV-1-HIV e 11 (39,3%) coinfectados com os vírus HTLV-2-HIV.
A coinfecção se mostrou mais frequente em pacientes do gênero masculino em comparação ao gênero feminino.
Os municípios com maior número de coinfectados HTLV-HIV foram Catanduva com 60,7% (17/28) seguido por São José do Rio preto com 21,4% (6/28) pacientes.
Em relação à faixa etária, observou-se que o maior número de indivíduos infectados tinham idade igual ou superior a 49 anos 74% (20/27).
Referências Bibliográficas
Assembleia Legislativa do estado de São Paulo. Notícias. Dezembro Vermelho: novos casos de HIV têm alta de quase 20% no Estado de São Paulo, 2021. Disponivel em:
<https://www.al.sp.gov.br/noticia/?id=431668#:~:text=Desde%201986%2C%20o%20Esta do%20de,detec%C3%A7%C3%A3o%20da%20presen%C3%A7a%20do%20v%C3%ADr us.>. Acesso em: 10 de outubro de 2022.
BRASIL. Ministério da Saúde. Portaria 1.376, de nov. 1993. Diário Oficial da União, Brasília, 2 de dez. 1993.
CATALAN-SOARES, Bernadette; CARNEIRO-PROIETTI, Anna Bárbara de F.; PROIETTI, Fernando Augusto. Heterogeneous geographic distribution of human T-cell lymphotropic viruses I and II (HTLV-I/II): serological screening prevalence rates in blood donors from large urban areas in Brazil. Cadernos de Saúde Pública, v. 21, p. 926-931, 2005.
CATERINO-DE-ARAUJO, Adele et al. Current prevalence and risk factors associated with human T lymphotropic virus type 1 and human T lymphotropic virus type 2 infections among HIV/AIDS patients in São Paulo, Brazil. AIDS Research and Human Retroviruses, v. 31, n. 5, p. 543-549, 2015.
DA INFECÇÃO, TERAPÊUTICAS PARA MANEJO. PROTOCOLO CLÍNICO E DIRETRIZES TERAPÊUTICAS PARA MANEJO DA INFECÇÃO PELO HIV EM ADULTOS. 2013.
DOURADO, Inês et al. HTLV-I in the general population of Salvador, Brazil: a city with African ethnic and sociodemographic characteristics. JAIDS Journal of Acquired Immune Deficiency Syndromes, v. 34, n. 5, p. 527-531, 2003.
EDLICH, Richard F.; ARNETTE, Jarrett A.; WILLIAMS, Freddie M. Global epidemic of human T-cell lymphotropic virus type-I (HTLV-I). The Journal of emergency medicine, v. 18, n. 1, p. 109-119, 2000.
GESSAIN, Antoine; CASSAR, Olivier. Epidemiological aspects and world distribution of HTLV-1 infection. Frontiers in microbiology, v. 3, p. 388, 2012.
HALL, William W. et al. Human T cell leukemia/lymphoma virus, type II (HTLV-II): emergence of an important newly recognized pathogen. In: Seminars in Virology. Academic Press, 1994. p. 165-178.
HINO, Shigeo. Establishment of the milk-borne transmission as a key factor for the peculiar endemicity of human T-lymphotropic virus type 1 (HTLV-1): the ATL Prevention Program Nagasaki. Proceedings of the Japan Academy, Series B, v. 87, n. 4, p. 152-166, 2011.
KAPLAN, Jonathan E. et al. Male-to-female transmission of human T-cell lymphotropic virus types I and II: association with viral load. JAIDS Journal of Acquired Immune Deficiency Syndromes, v. 12, n. 2, p. 193-201, 1996.
LOWIS, George W.; SHEREMATA, William A.; MINAGAR, Alireza. Epidemiologic features of HTLV-II: serologic and molecular evidence. Annals of epidemiology, v. 12, n. 1, p. 46-66, 2002.
MINISTÉRIO DA SAÚDE (BR). SECRETARIA DE VIGILÂNCIA EM SAÚDE. DEPARTAMENTO DE VIGILÂNCIA, PREVENÇÃO E CONTROLE DAS INFECÇÕES SEXUALMENTE TRANSMISSÍVEIS, DO HIV/AIDS E DAS HEPATITES VIRAIS.
Manual técnico para o diagnóstico da infecção pelo HIV em adultos e crianças. 2018.
MONTANHEIRO, Patricia Aparecida. Quantificação de carga proviral do vírus linfotrópico de células T humanas tipo 1 (HTLV-1) e marcadores imunológicos em indivíduos portadores e pacientes com TSP/HAM. 2007. Tese de Doutorado. Universidade de São Paulo.
MURPHY, Edward L. et al. Evidence among blood donors for a 30-year-old epidemic of human T lymphotropic virus type II infection in the United States. Journal of Infectious Diseases, v. 180, n. 6, p. 1777-1783, 1999.
MURPHY, Edward L. et al. Sexual transmission of human T-lymphotropic virus type I (HTLV-I). Annals of internal medicine, v. 111, n. 7, p. 555-560, 1989.
OKOCHI, Kazuo; SATO, Hiroyuki; HINUMA, Yorio. A retrospective study on transmission of adult T cell leukemia virus by blood transfusion: seroconversion in recipients. Vox sanguinis, v. 46, n. 5, p. 245-253, 1984.
PEREIRA, Felicidade Mota et al. Distribution of human T-lymphotropic virus (HTLV) and hepatitis C co-infection in Bahia, Brazil. PloS one, v. 15, n. 7, p. e0223087, 2020.
PEREIRA, Felicidade Mota et al. Taxa de infecção do vírus linfotrópico de células humanas (HTLV), vírus da hepatite C (HCV), coinfecção HTLV/HCV no Estado da Bahia e impacto da coinfecção HTLV/HCV no perfil de citocinas. 2019. Tese de Doutorado. Instituto Gonçalo Moniz.
ROSADAS, Carolina; TAYLOR, Graham P. HTLV-1 and co-infections. Frontiers in Medicine, v. 9, 2022.
ROUCOUX, Diana F. et al. A prospective study of sexual transmission of human T lymphotropic virus (HTLV)–I and HTLV-II. The Journal of infectious diseases, v. 191, n. 9, p. 1490-1497, 2005.
STUVER, Sherri O. et al. Heterosexual transmission of human T cell leukemia/lymphoma virus type I among married couples in southwestern Japan: an initial report from the Miyazaki Cohort Study. Journal of Infectious Diseases, v. 167, n. 1, p. 57-65, 1993.
TAKEZAKI, Toshiro et al. Incidence of human T lymphotropic virus type I seroconversion after age 40 among Japanese residents in an area where the virus is endemic. Journal of Infectious Diseases, v. 171, n. 3, p. 559-565, 1995.
