Capítulo de livro publicado no livro do I CONGEB. Para acessa-lo clique aqui.
DOI: https://doi.org/10.53934/9786585062046-18
Este trabalho foi escrito por:
Elvia Jéssica da Silva Oliveira1 *; Vanessa Emanuelle Santos 1; Francielly Negreiros de Araújo1 ; Wilson José da Silva Júnior2 ; Éderson Akio Kido1
1. Laboratório de Genética Molecular de Plantas – LGM, UFPE, Recife, PE, Brasil.
2 Hospital Israelita Albert Einstein, São Paulo, SP, Brasil.
*Autor correspondente (Elvia Jéssica da S. Oliveira) – Email: [email protected]
Resumo: A fotorrespiração é uma via alternativa que impede a fotoinibição e o acúmulo de espécies reativas de oxigênio. A dupla afinidade da ribulose-1,5-bifosfato carboxilase/oxigenase (RubisCO) para fixação de gás carbônico (CO2) e oxigênio (O2), quando assume a função de oxigenase, resulta na fixação de oxigênio em um processo denominado fotorrespiração. Dentre as enzimas responsáveis por esse processo está a SHMT que converte a glicina em serina em uma de suas fases que ocorre na mitocôndria. As plantas mediante estresse abiótico tendem a oferecer um aparato menos danoso para realizar suas atividades fisiológicas. Neste trabalho foram caracterizados os genes de SHMT e suas respectivas proteínas em Jatropha curcas, em três espécies pertencentes a família Euphorbiaceae e em Arabidopsis. A estrutura gênica e os motivos conservados seguem um padrão para cada grupo de SHMT caracterizado. As proteínas SHMT tem apenas um domínio (SHMT). A árvore fenética obtida ficou agrupada em dois clados, que correspondem a Classe I, subgrupos SHMT7 e SHMT4, e a Classe II, subgrupos SHMT3 e SHMT1. Na análise das regiões promotoras foram identificados sítios de ligação aos fatores de transcrição Dof, HSF, C2H2, bZIP, Myb-related, bHLH e B3, os quais são intimamente relacionados a estresses abióticos. A análise de expressão gênica de raízes do genótipo Jc171 de J. curcas submetido à 150 mM de NaCl identificou apenas a SHMT4 que é comumente encontrada no tecido radicular, assim, estudos adicionais utilizando o tecido foliar possibilitará um respaldo significativo sobre as outras SHMT envolvidas na via da fotorrespiração de J. curcas.
Palavras–chave: Pinhão-manso; Fotorrespiração; Estresse Salino.
INTRODUÇÃO
O pinhão-manso (Jatropha curcas), pertence à família Euphorbiaceae e é uma oleaginosa, encontrada em regiões áridas, com potencial para a produção de biocombustíveis, além de apresentar um sistema radicular eficaz para controle da erosão dos solos, que pode ser utilizado para proteger áreas degradadas (1). Esta espécie é considerada tolerante a seca, devido, principalmente, ao seu caule suculento que contribui para reserva de água, equilíbrio na perda de folhas, e uma taxa de transpiração conservada (2). Em virtude da correlação existente entre os estresses de seca e salinidade, os diferentes genótipos de pinhão-manso apresentam respostas variáveis quando comparados entre si, podem ser tolerantes a salinidade dos solos ou apresentar uma resposta mais próxima a um genótipo sensível (3).
As condições ambientais adversas como salinidade, seca e resfriamento podem estimular o acúmulo de serina hidroximetiltransferase (SHMT) em células vegetais. A SHMT está amplamente distribuída nas plantas, e desempenha um papel positivo na regulação e resistência das plantas a estresse abiótico (4). Esta enzima minimiza os danos causados pelo estresse abiótico através da modulação do nível de espécies reativas de oxigênio (ERO), reduzindo danos oxidativos; recupera o metabolismo celular afetado e; está correlacionada com a atividade fotorrespiratória (5).
Diferentes trabalhos têm constatado a influência positiva de SHMT para a tolerância ao sal. Em arroz, por exemplo, foi observada uma alta expressão de SHMT associada a resposta ao estresse salino, quando comparado com a variedade sensível ao sal (6). Em Arabidopsis, a mutação S31D em SHMT1 reduziu o fechamento estomático induzido por sal, destacando a importância de manter a atividade fotorrespiratória de SHMT1 sob condições salinas e de estresse hídrico (7).
Sob condições de alta salinidade, as plantas têm a fotossíntese inibida pelo fechamento estomático e diminuição de gás carbônico (CO2), a irregularidade da assimilação fotossintética de CO2 são capazes de elevar as taxas da fotorrespiração (8). Os indicativos de taxas mais altas de fotorrespiração, mostram aumentar o CO2, a estimulação da fotossíntese, diminuindo a concentração de oxigênio (O2), a atividade da glicolato oxidase e a formação de metabólitos fotorrespiratórios (glicina e serina), além de manter as taxas de transporte de elétrons em CO2 (8).
A fotorrespiração é uma importante via alternativa sob condições limitantes de CO2, pois a Rubisco assume a função de oxigenase e incorpora os O2 e equivalentes redutores em excesso das reações fotoquímicas, evitando a fotoinibição (9). A rota metabólica da fotorrespiração ocorre em três compartimentos subcelulares, iniciando no cloroplasto, seguido do peroxissomo e por fim na mitocôndria, onde ocorre a conversão de serina em glicina através da enzima SHMT (EC 2.1.2.1), uma enzima piridoxal-5’-fosfato, desempenhando um papel vital no metabolismo de um carbono (5).
Neste trabalho foram analisados, a estrutura éxon-íntron dos genes SHMT; a distribuição dos fatores de transcrição associados aos promotores; os motivos conservados, ponto isoelétrico, massa molecular e localização subcelular das proteínas SHMT; e o padrão de expressão tecido-específico de genes SHMT em plantas de Jatropha curcas.
MATERIAL E MÉTODOS
Dados disponíveis de RNA-Seq
A origem dos dados que correspondem ao RNA-Seq de J. curcas estão disponíveis em Souza et al (10) e de Lima Cabral et al (11). Os acessos utilizados foram selecionados com base em ensaios prévios (3). O experimento seguiu delineamento inteiramente casualizado com dois acessos (Jc171e Jc183), dois tratamentos (com e sem exposição salina de 150 mM NaCl /3 h), e três plantas meio-irmãs de cada acesso. Após 3 h de exposição ao NaCl, amostras de raízes foram coletadas e RNAs de boa qualidade (RNA Integrity Number, RIN>8; Agilent Bioanalyzer RNA 6000) foram usados na geração de bibliotecas RNA-Seq (Illumina TruSeq Stranded mRNA Sample Prep LS Protocol), que foram posteriormente sequenciadas (HiSeq Flow Cell v4; leituras pareadas de 100 bp) pelo Centro de Genômica da Escola Superior de Agricultura “Luiz de Queiroz” (ESALQ/USP, Piracicaba, SP, Brasil).
Montagem por referência: Tratamento e análise dos reads RNA-seq
O genoma representativo de J. curcas, recuperado do portal NCBI (RefSeq) correspondeu à montagem RJC1_Hi-C (GCF_014843425.1), de 05/10/2020, que juntamente com o transcriptoma de referência foram indexados ao Bowtie2 v.2.3.5.1. Os reads brutos foram avaliados pelo FastQC v.0.11.9, sendo os adaptadores, removidos pelo Cutadapt v.3.7. Os reads “clipados” foram filtrados pelo Sickle v.1.33, pelos critérios de qualidade e comprimento (20 para ambos). Os dados filtrados foram alinhados contra o transcriptoma de referência pelo Tophat2 v.2.1.1 e o Cufflinks v.2.2.1 foi usado para gerar as planilhas e cálculos de estatísticas. A montagem gerada foi anotada a partir dos dados de referência (RJC1_Hi-C) de J. curcas disponíveis no NCBI.
Visualização da expressão gênica de SHMT em Jatropha curcas
A expressão dos transcritos considerou os valores de Log2 FoldChange (Log2FC) ≥ 1 para transcritos induzidos (UR, up-regulated) e Log2FC ≤ 1 para transcritos reprimidos (DR, down-regulated), sendo visualizados através da ferramenta TBTools (https://github.com/CJ-Chen/TBtools).
Caracterização de SHMT em Jatropha curcas
As sequências polipeptídicas de serina hidroximetiltransferase (SHMT) foram obtidas do proteoma de Jatropha curcas (GCF_014843425.1) disponível no NCBI (https://www.ncbi.nlm.nih.gov/assembly). Estas sequências foram submetidas na ferramenta online Blastp (https://blast.ncbi.nlm.nih.gov/Blast.cgi) para encontrar sequências similares nas espécies Manihot esculenta, Ricinus communis e Hevea brasilienses, pertencentes a família Euphorbiaceae, e Arabidopsis thaliana como grupo externo. Os domínios das sequências foram confirmados pelo Pfam [https://pfam.xfam.org/(12)] e visualizados no Tbtools (13). A análise de motivos conservados das proteínas SHMT foi realizada no software MEME [https://meme-suite.org/meme/tools/meme(14)] e visualizados no Tbtools. A anotação dos motivos foi realizada através da ferramenta online InterProScan (https://www.ebi.ac.uk/interpro/search/sequence/).
A massa molecular (kDa) e os valores de ponto isoelétrico foram previstos pela ferramenta Expasy [https://web.expasy.org/compute_pi/(15)] das as proteínas SHMT enquanto a localização subcelular foi prevista pela ferramenta DeepLoc 2.0 [https://services.healthtech.dtu.dk/service.php?DeepLoc-2.0(16)].
Alinhamento de múltiplas sequências e análise fenética
As sequências polipeptídicas foram alinhadas no JalView (17), visando analisar os padrões de conservação por identidade, seguindo metodologia de alinhamento do ClustalW e a árvore fenética foi construída no MEGA11 (18), pelo método de Neighboor Joining-NJ, utilizando bootstrap de 10000. A estrutura éxon-íntron dos genes foi visualizada no programa Tbtools, utilizando o identificador dos transcritos de cada espécie e arquivo GTF (https://www.ncbi.nlm.nih.gov/assembly/) correspondente, como arquivo de entrada.
Predição de fatores de transcrição associados às sequências promotoras de genes SHMT
As regiões promotoras (1,0 kb) dos potenciais genes de J. curcas que codificariam enzimas componentes das via de fotorrespiração foram extraídas pelo pacote GFF-Ex [Genome Feature Extraction Package; http://bioinfo.icgeb.res.in/gff/ (19)] a partir da linha de comando: “caminho da pasta”/gffex -in (gff) -db (sequence).
A prospecção dos motivos candidatos a elementos cis regulatórios (CECRs) em cada região promotora foi feita através do software MEME v5.0. [http://meme-suite.org/tools/meme(14)], adotando-se e10−2 para caracterização como CECRs e máximo de 10 motivos. Motivos foram restritos entre 6 e 50 nucleotídeos de comprimento.
Após a análise supracitada utilizou-se o software Tomtom v4.11.2 (http://meme-suite.org/tools/tomtom)(20), comparando os identificadores detectados com um banco de dados de motivos curados (JASPAR), nas opções “JASPAR (NON-REDUNDANT) DNA” e “JASPARCORE (2022) plants”, considerando os alinhamentos mais significativos gerados pelo programa (p-value<0,05). O identificador JASPAR, correspondente a cada alinhamento, foi pesquisado no banco [https://jaspar.genereg.net/(21)], , onde é possível obter os fatores de transcrição que estão relacionados a região promotora de interesse.
RESULTADOS E DISCUSSÃO
Identificação de SHMT
A enzima hidroximetiltransferase (SHMT) foi identificada em Jatropha curcas, Manihot esculenta, Ricinus communis, Hevea brasilienses e Arabidopsis thaliana (Tabela 1). As proteínas correspondentes a enzima SHMT que apresentaram domínio incompleto foram descartadas das análises posteriores.
Tabela 1 – Quantitativo de serina hidroximetilttranferase (SHMT) nas espécies.
| Espécie | Loci | Transcritos | Proteínas |
| Arabidopsis thaliana | 5 | 5 | 3 |
| Jatropha curcas | 6 | 9 | 5 |
| Manihot esculenta | 10 | 14 | 9 |
| Ricinus communis | 6 | 7 | 6 |
| Hevea brasilienses | 10 | 23 | 10 |
| Total | 37 | 58 | 33 |
Fonte: Bancos de dados NCBI
Caracterização de SHMT em Jatropha curcas
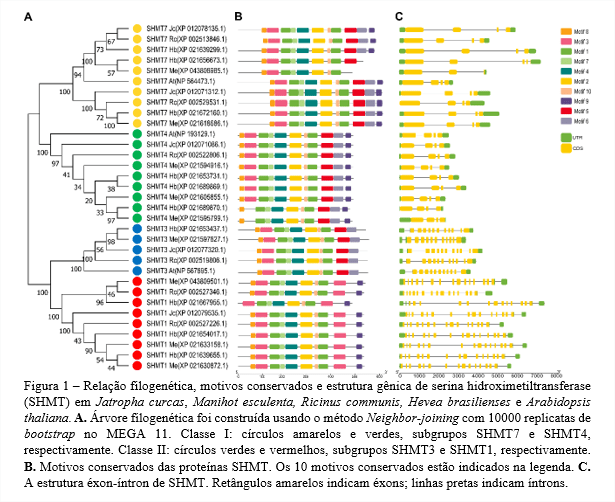
A árvore filogenética de serina hidroximetiltransferase (SHMT) (Fig. 1A) ficou agrupada em dois clados, que correspondem a Classe I, subgrupos SHMT7 e SHMT4, e a Classe II, subgrupos SHMT3 e SHMT1, nela estão representantes das espécies Manihot esculenta, Ricinus communis e Hevea brasilienses, pertencentes a família Euphorbiaceae, e Arabidopsis thaliana como grupo externo. A anotação dos motivos conservados destas proteínas encontra-se na Tabela 2, os quais variaram entre 15 e 50 aminoácidos. Além disso, esses motivos conservados (Fig. 1B) seguem o mesmo padrão de evolução da estrutura éxon-íntron (Fig. 1C) de seus respectivos genes.
O subgrupo SHMT7 possui todos os motivos, o que confere a este subgrupo um alto nível de conservação, exceto SHMT7_Hb(XP_021656673.1) que não possui os motivos 6 e 9,e SHMT7_Me(XP_043808985.1) que não possui os motivos 5, 6 e 9. Em SHMT4, os motivos seguem um padrão bem conservado exceto em SHMT4_Hb(XP_021689870.1) e SHMT4_Me(XP_021595799.1) que não possuem o motivo 3. O subgrupo SHM3, Classe II possui todos os motivos exceto o motivo 9, e o subgrupo SHMT1 possui todos os motivos, exceto o motivo 5 e SHMT1_Hb(XP_021667955.1) não possui os motivos 5 e 8. As SHMT de Jatropha curcas possuem todos os motivos que agrupam cada clado.
A análise da estrutura dos genes SHMT indicaram que membros intimamente relacionados tem uma estrutura éxon-íntron semelhante como observado na Figura (1C). A divergência da estrutura éxon-íntron é importante para observar a evolução de genes duplicados e as diferentes composições de motivos e sua diversidade funcional (22). Em SHMT7 e SHMT4 o número de éxons e íntrons correspondem a 4 e 3, respectivamente. Em SHMT3 tem um padrão de 11 éxons e 11 íntrons, e SHMT 15 éxons e 15 íntrons (Fig. 1C). As respectivas proteínas de SHMT tiveram o domínio SHMT previsto.

As proteínas SHMT são formadas por 458 a 538 aminoácidos, apresentam massa molecular variando de 50,15 a 66,29 kDa, ponto isoelétrico entre 5,65 a 9,13 e a localização subcelular desta proteína é no citoplasma, nas organelas mitocôndria e plastídio ou no núcleo, conforme Tabela 3. Esta localização também foi observada na cultura de tomate em Liu et al (23). O SHMT presente no cloroplasto apresenta um papel vital para fotorrecepção e biossíntese de metabolismo de um carbono (24).
Tabela 3 – Localização subcelular, massa molecular (Mw) e ponto isoelétrico (pI) das proteínas serina hidroximetiltransferase (SHMT) em Jatropha curcas, Manihot esculenta, Ricinus communis, Hevea brasilienses e Arabidopsis thaliana.
| Proteína / Espécie | Identificador NCBI | Aa | pI | Mw (kDa) | Localização subcelular |
| SHMT1_Hb | XP_021639655.1 | 515 | 8,56 | 57,10 | Mitocôndria |
| SHMT1_Hb | XP_021654017.1 | 515 | 8,37 | 57,26 | Mitocôndria |
| SHMT1_Hb | XP_021667955.1 | 466 | 9,13 | 51,65 | Citoplasma | Mitocôndria |
| SHMT1_Jc | XP_012079535.1 | 515 | 8,88 | 56,88 | Mitocôndria |
| SHMT1_Me | XP_021630872.1 | 515 | 8,69 | 57,03 | Mitocôndria |
| SHMT1_Me | XP_021633158.1 | 515 | 8,88 | 56,96 | Mitocôndria |
| SHMT1_Me | XP_043809501.1 | 516 | 8,65 | 57,10 | Mitocôndria |
| SHMT1_Rc | XP_002527226.1 | 515 | 8,84 | 56,88 | Mitocôndria |
| SHMT1_Rc | XP_002527346.1 | 513 | 8,11 | 56,57 | Mitocôndria |
| SHMT3_At | NP_567895.1 | 529 | 9,03 | 57,98 | Plastídio |
| SHMT3_Hb | XP_021653437.1 | 520 | 8,08 | 56,91 | Plastídio |
| SHMT3_Jc | XP_012077320.1 | 527 | 8,48 | 57,19 | Plastídio |
| SHMT3_Me | XP_021597827.1 | 533 | 7,62 | 57,98 | Plastídio |
| SHMT3_Rc | XP_002519806.1 | 527 | 7,21 | 57,54 | Plastídio |
| SHMT4_At | NP_193129.1 | 471 | 6,80 | 51,72 | Citoplasma |
| SHMT4_Hb | XP_021653731.1 | 471 | 8,24 | 52,01 | Citoplasma |
| SHMT4_Hb | XP_021689869.1 | 471 | 7,19 | 51,81 | Citoplasma |
| SHMT4_Hb | XP_021689870.1 | 458 | 7,61 | 50,15 | Citoplasma |
| SHMT4_Jc | XP_012071086.1 | 471 | 7,17 | 51,73 | Citoplasma |
| SHMT4_Me | XP_021594916.1 | 471 | 7,59 | 51,65 | Citoplasma |
| SHMT4_Me | XP_021595799.1 | 468 | 5,65 | 51,59 | Citoplasma |
| SHMT4_Me | XP_021605855.1 | 471 | 8,18 | 51,79 | Citoplasma |
| SHMT4_Rc | XP_002522806.1 | 471 | 7,59 | 51,87 | Citoplasma |
| SHMT7_At | NP_564473.1 | 598 | 8,03 | 66,29 | Núcleo |
| SHMT7_Hb | XP_021639299.1 | 557 | 6,97 | 61,41 | Núcleo |
| SHMT7_Hb | XP_021656673.1 | 510 | 7,56 | 56,45 | Núcleo |
| SHMT7_Hb | XP_021672160.1 | 590 | 5,80 | 65,95 | Núcleo |
| SHMT7_Jc | XP_012071312.1 | 588 | 6,62 | 65,36 | Núcleo |
| SHMT7_Jc | XP_012078135.1 | 558 | 7,24 | 61,74 | Núcleo |
| SHMT7_Me | XP_021618686.1 | 591 | 5,94 | 66,08 | Núcleo |
| SHMT7_Me | XP_043808985.1 | 466 | 7,87 | 51,41 | Núcleo |
| SHMT7_Rc | XP_002513846.1 | 567 | 7,55 | 62,66 | Núcleo |
| SHMT7_Rc | XP_002529531.1 | 590 | 5,98 | 66,11 | Núcleo |
Montagem por Referência
A montagem de referência para o acesso Jc171 resultou em 13389 transcritos, destes, os transcritos considerados significantes para o corte (p-value ≤ 0,05; q-value ≤ 0,05; Log 2 FC ≥ 1 ou ≤ -1) estabelecido foram distribuídos da seguinte forma 3001 UR (up-regulated), 2965 DR (down-regulated) e 1865 n.s. (no significant), os demais foram excluídos da análise pois não atingiram o ponto de corte estabelecido. Enquanto para o acesso Jc183 foram 438 UR, 217 DR e 116 n.s, os demais foram excluídos da análise pois não atingiram o ponto de corte estabelecido.
Expressão diferencial in sílico de transcritos SHMT em Jatropha curcas
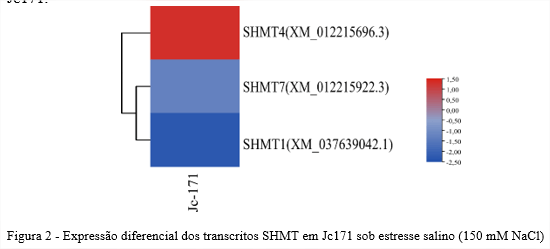
Em Jatropha curcas foram identificados nove transcritos SHMT, porém o genótipo Jc171 apresentou seis transcritos de SHMT. Enquanto o genótipo Jc183 não apresentou SHMT diferencialmente expressas, isso se explica pela concentração de NaCl utilizada ser inferior ao que este genótipo suporta, e por ser considerado um genótipo tolerante quando comparado ao Jc171(3). Em Jc171 havia um transcrito que estava regulado positivamente, o SHMT4(XM_012215696.3), e dois negativamente, como observado na Figura 2. Em Arabidopsis a acumulação AtSHMT4 foi restrita a raiz em plântulas jovens (24), o presente estudo é com base em dados provenientes de raiz de Jc171.
A serina hidroximetiltransferase é a enzima responsável pela conversão de glicina em serina (Figura 3). A SHMT também possui um papel indispensável na via fotorrespiratória de organismos fotossintéticos aeróbicos (25). Já foi observado a superexpressão de SHM1 em Arabidopsis resgatando parcialmente seu fenótipo sensível ao sal (26). A SHMT tem um papel crucial na interação com glutamato sintase na formação do complexo fotorrespiratório (27). Em Arabidopsis, foi superexpresso o gene OsSHMT3 (Oryza sativa) o qual exibiu tolerância ao estresse salino (150 mM NaCl) (28).
Fatores de transcrição associados aos genes SHMT de Jatropha curcas
Os fatores de transcrição (FTs) funcionam como reguladores capazes de controlar processos que especificam tipos celulares, padrões de desenvolvimento e modulam vias específicas nas plantas, tendo um papel essencial quando elas estão sob algum tipo de estresse abiótico (29). Neste estudo foram analisados os FTs (Figura 4) associados as SHMT do genótipo Jc171 de J. curcas sob salinidade. Os FTs B3 e bHLH foram exclusivos de SHMT4 e SHMT7, respectivamente. O FT Myb-related aparece em SHMT1 e SHMT7. O FT Dof e HSF apareceram em SHMT1, SHMT3 e SHMT4. O FT C2H2 em SHMT1, SHMT4, SHMT7. O FT bZIP em SHM4. O FT GATA20 aparece em todos os SHMTs, exceto a SHMT(LOC105630748).
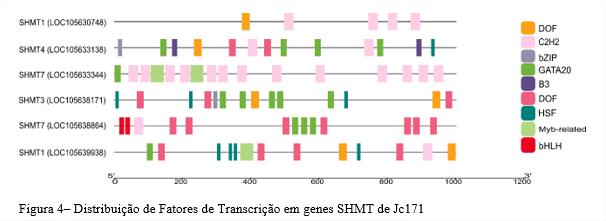
Em pinhão manso (Jatropha curcas), bZIP conferiu resistência a seca e a salinidade, este FT é envolvido com processos biológicos que incluem, desenvolvimento e crescimento das plantas (11,30). Em arroz (Oryza sativa), houve acúmulo de Myb-related, conferindo tolerância ao estresse salino e a seca, este FT que tem suas atividades essenciais no desenvolvimento e crescimento da planta (31). Em pimenta (Capsicum annuum L.), o bHLH estava regulando positivamente sob estresse salino, aquecimento, frio e seca, este FT está relacionado com estresses ambientais e envolvido na transdução de sinal e vias anabólicas (32). Os fatores de transcrição citados promovem a transcrição de genes importantes para a adaptação e respostas das plantas aos fatores abióticos.
CONCLUSÕES
A caracterização estrutural de serina hidroximetiltransferase é de extrema relevância para indicar sua participação em diferentes compartimentos subcelulares. O seu acúmulo e sua expressão influenciam na via metabólica de fotorrespiração, atuando na conversão de glicina em serina. No genótipo Jc171 de J. curcas foram poucas identificadas, pois o tecido o qual estava sendo analisado foi o radicular e apresentou apenas a SHMT4 que é mais comum nesta região. Uma análise utilizando tecido foliar poderá fornecer mais informações sobre esse gene, uma vez que ele atua diretamente na via fotorrespiratória a qual é ativada pelo excesso de O2 provocado por fatores externos, como os estresses ambientais.
AGRADECIMENTOS
À Capes pelo financiamento do projeto e concessão de bolsas.
REFERÊNCIAS
1. Reubens B, Achten WMJ, Maes WH, Danjon F, Aerts R, Poesen J, et al. More than biofuel? Jatropha curcas root system symmetry and potential for soil erosion control. J Arid Environ [Internet]. 2011;75(2):201–5. Available from: http://dx.doi.org/10.1016/j.jaridenv.2010.09.011
2. Maes WH, Achten WMJ, Reubens B, Raes D, Samson R, Muys B. Plant-water relationships and growth strategies of Jatropha curcas L. seedlings under different levels of drought stress. J Arid Environ. 2009;73(10):877–84.
3. Lozano-Isla F, Campos MLO, Endres L, Bezerra-Neto E, Pompelli MF. Effects of seed storage time and salt stress on the germination of Jatropha curcas L. Ind Crops Prod. 2018;118(July 2017):214–24.
4. Kopriva S, Bauwe H. Serine hydroxymethyltransferase from Solanum tuberosum. Plant Physiol. 1995;107(1):271–2.
5. Fang C, Zhang P, Li L, Yang L, Mu D, Yan X, et al. Serine hydroxymethyltransferase localised in the endoplasmic reticulum plays a role in scavenging H2O2 to enhance rice chilling tolerance. BMC Plant Biol. 2020;20(1):1–13.
6. Patel MK, Joshi M, Mishra A, Jha B. Ectopic expression of SbNHX1 gene in transgenic castor (Ricinus communis L.) enhances salt stress by modulating physiological process. Plant Cell Tissue Organ Cult. 2015;122(2):477–90.
7. Liu Y, Guérard F, Glab N. Photorespiratory serine hydroxymethyltransferase 1 activity impacts abiotic stress tolerance and stomatal closure. 2019;(May):2567–83.
8. Wingler A, Lea PJ, Quick WP, Leegood RC. Photorespiration: Metabolic pathways and their role in stress protection. Philos Trans R Soc B Biol Sci. 2000;355(1402):1517–29.
9. Lima Neto MC, De Oliveira Martins M, Ferreira-Silva SL, Silveira JAG. Jatropha curcas and ricinus communis display contrasting photosynthetic mechanisms in response to environmental conditions. Sci Agric. 2015;72(3):260–9.
10. Souza MCP, Silva MD, Binneck E, de Lima Cabral GA, Benko Iseppon AM, Pompelli MF, et al. RNA-Seq transcriptome analysis of Jatropha curcas L . accessions after salt stimulus and unigene-derived microsatellite mining. Ind Crop Prod [Internet]. 2020;147(April 2019):112168. Available from: https://doi.org/10.1016/j.indcrop.2020.112168
11. de Lima Cabral GA, Binneck E, de Souza MCP, da Silva MD, Costa Ferreira Neto JR, Pompelli MF, et al. First Expressed TFome of Physic Nut (Jatropha curcas L.) After Salt Stimulus. Plant Mol Biol Report. 2020;38(2):189–208.
12. Finn RD, Bateman A, Clements J, Coggill P, Eberhardt RY, Eddy SR, et al. Pfam: The protein families database. Nucleic Acids Res. 2014;42(D1):222–30.
13. Chen C, Chen H, Zhang Y, Thomas HR, Frank MH, He Y, et al. TBtools : An Integrative Toolkit Developed for Interactive Analyses of Big Biological Data. Mol Plant [Internet]. 2020;13(8):1194–202. Available from: https://doi.org/10.1016/j.molp.2020.06.009
14. Bailey TL, Elkan C. Fitting a mixture model by expectation maximization to discover motifs in biopolymers. Proc Int Conf Intell Syst Mol Biol. 1994;2:28–36.
15. Duvaud S, Gabella C, Lisacek F, Stockinger H, Ioannidis V, Durinx C. Expasy, the Swiss Bioinformatics Resource Portal, as designed by its users. Nucleic Acids Res. 2021;49(W1):W216–27.
16. Thumuluri V, Armenteros, Juan Almagro, Johansen AR, Nielsen H, Winther O. Prediction Using Protein Language Models. 2022;50(April):1–7.
17. Clamp M, Cuff J, Searle SM, Barton GJ. The Jalview Java alignment editor. Bioinformatics. 2004;20(3):426–7.
18. Tamura K, Stecher G, Kumar S. MEGA11: Molecular Evolutionary Genetics Analysis Version 11. Mol Biol Evol. 2021;38(7):3022–7.
19. Rastogi A, Gupta D. GFF-Ex: A genome feature extraction package. BMC Res Notes. 2014;7(1):7–9.
20. Gupta S, Stamatoyannopoulos JA, Bailey TL, Noble WS. Quantifying similarity between motifs. Genome Biol. 2007;8(2).
21. Sandelin A, Alkema W, Engström P, Wasserman WW, Lenhard B. JASPAR: An open-access database for eukaryotic transcription factor binding profiles. Nucleic Acids Res. 2004;32(DATABASE ISS.).
22. Yu J, Xie Q, Li C, Dong Y, Zhu S, Chen J. Comprehensive characterization and gene expression patterns of LBD gene family in Gossypium. Planta [Internet]. 2020;251(4):1–16. Available from: https://doi.org/10.1007/s00425-020-03364-8
23. Liu Z, Pan X, Wang C, Yun F, Huang D, Yao Y, et al. Genome-wide identification and expression analysis of serine hydroxymethyltransferase (SHMT) gene family in tomato (Solanum lycopersicum). PeerJ. 2022;10.
24. Zhang Y, Sun K, Sandoval FJ, Santiago K, Roje S. One-carbon metabolism in plants : characterization of a plastid serine hydroxymethyltransferase. 2010;105:97–105.
25. Waditee R, Bhuiyan NH, Hirata E, Hibino T, Tanaka Y, Shikata M, et al. Metabolic Engineering for Betaine Accumulation in Microbes. J Biol Chem [Internet]. 2007;282(47):34185–93. Available from: http://dx.doi.org/10.1074/jbc.M704939200
26. Zhou H, Zhao J, Yang Y, Chen C, Liu Y, Jin X, et al. UBIQUITIN-SPECIFIC PROTEASE16 modulates salt tolerance in arabidopsis by regulating Na+/H+ antiport activity and serine hydroxymethyltransferase stability. Plant Cell. 2013;24(12):5106–22.
27. Jamai A, Salomé PA, Schilling SH, Weber APM, McClung CR. Arabidopsis photorespiratory serine hydroxymethyltransferase activity requires the mitochondrial accumulation of ferredoxin-dependent glutamate synthase. Plant Cell. 2009;21(2):595–606.
28. Mishra P, Jain A, Takabe T, Tanaka Y, Negi M, Wood A. Heterologous Expression of Serine Hydroxymethyltransferase-3 From Rice Confers Tolerance to Salinity Stress in E . coli and Arabidopsis. 2019;10(March):1–17.
29. Villano C, Esposito S, D’Amelia V, Garramone R, Alioto D, Zoina A, et al. WRKY genes family study reveals tissue-specific and stress-responsive TFs in wild potato species. Sci Rep. 2020;10(1):1–12.
30. Wang Z, Zhu J, Yuan W, Wang Y, Hu P, Jiao C, et al. Genome-wide characterization of bZIP transcription factors and their expression patterns in response to drought and salinity stress in Jatropha curcas. Int J Biol Macromol [Internet]. 2021;181:1207–23. Available from: https://doi.org/10.1016/j.ijbiomac.2021.05.027
31. Xiong H, Li J, Liu P, Duan J, Zhao Y, Guo X, et al. Overexpression of OsMYB48-1, a novel MYB-related transcription factor, enhances drought and salinity tolerance in rice. PLoS One. 2014;9(3):1–13.
32. Zhang Z, Chen J, Liang C, Liu F, Hou X, Zou X. Genome-Wide Identification and Characterization of the bHLH Transcription Factor Family in Pepper (Capsicum annuum L.). Front Genet. 2020;11(September):1–14.
Próximo


