ARMAZENAMENTO E IDENTIFICAÇÃO DE FUNGOS FILAMENTOSOS POR MÉTODOS MORFOLÓGICOS E POR BIOLOGIA MOLECULAR
- Home
- /
- Blog
- /
- Livros
- /
- Capítulo de Livro IV Mostra
- /
- ARMAZENAMENTO E IDENTIFICAÇÃO DE FUNGOS FILAMENTOSOS POR MÉTODOS MORFOLÓGICOS E POR BIOLOGIA MOLECULAR
Capítulo de livro publicado no Livro da IV Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
doi.org/10.53934/9786599965821-20
Este trabalho foi escrito por:
Bryan Kaique de Carvalh¹; Dra Tânia Sueli de Andrade²
1Estudante do Curso de Vigilância Epidemiológica em Saúde Pública – NCMO – IAL – Central. [email protected]
2Pesquisadora Docente do Núcleo de Coleção de Micro-organismos– NCMO– IAL – Central.
Resumo: As coleções de micro-organismos são responsáveis pela preservação da biodiversidade, para isso é preciso que as linhagens armazenadas estejam corretamente autenticadas. O Núcleo de Coleção Micro-organismos do Instituto Adolfo Lutz possui em seu acervo aproximadamente 10.000 linhagens, sendo pouco mais de 600 fungos, importantes causadores de infecções oportunistas. O objetivo desse trabalho foi avaliar os métodos de liofilização e criopreservação; e caracterizar vinte linhagens fúngicas depositadas no NCMO, por meio de sua morfologia (micro e macroscópicas) e técnicas moleculares como MALDI-TOF e sequenciamento Sanger para os alvos ITS e gene da calmudolina. As linhagens de Apergillus fumigatus e A. terreus foram identificadas por MALDI-TOF; A. flavus apontado como membro do grupo Aspergillus flavus oryzae. As linhagens de Fusarium oxysporum e F. solani foram identificadas por MALDI-TOF e sequenciamento para alvo ITS; F. equiseti não pode ser identificado por técnicas moleculares. Uma linhagem de Curvularia foi identificada por sequenciamento do gene rDNA região ITS, outra linhagem foi parcialmente identificada, ambas tendo duas espécies como resultado, sendo diferenciadas por macromorfologia. As linhagens de Fonsecaea pedrosoi não apresentavam identificação prévia, pois foram encaminhadas para depósito recentemente, os resultados obtidos do sequenciamento do rDNA região ITS, confirmaram a identificação de origem para uma delas, e duas foram parcialmente identificadas. Porém uma linhagem foi identificada como Phialophora sp por sequenciamento por micromorfologia, não correspondendo à sua identificação de origem. Todos os gêneros trabalhados apresentaram características descritas na literatura. Tanto os métodos de identificação quanto os de armazenamento se mostram eficientes.
Palavras-chaves: armazenamento, autenticação, coleções de micro-organismos, Fonsecaea, fungos
Introdução
A preservação ex situ, fora do habitat natural, dos recursos microbiológicos é uma prática indispensável para o conhecimento e uso da biodiversidade, as chamadas coleções de cultura de micro-organismos tem um papel importante neste contexto, como o Núcleo de Coleção Micro-organismos do Instituto Adolfo Lutz, que possui em seu acervo aproximadamente
10.000 linhagens, sendo pouco mais de 600 fungos, importantes causadores de infecções consideradas clássicas e oportunistas. A preservação adequada dos isolados fúngicos é imprescindível para uso em investigações científicas (SETTE; PAGNOCCA; RODRIGUES, 2013; BOUNDY-MILLS et. al. 2019). Para a conservação de fungos filamentosos a preservação a longo prazo pode ser realizada através da criopreservação a – 80°C ou também pela liofilização, processo de sublimação a vácuo (OLIVEIRA; CORTEZ; SOUZA 2016; COSTA et al 2009). Após os processos de manutenção, a autenticação das linhagens garante a qualidade do acervo. No caso dos fungos filamentosos a taxonomia baseia-se principalmente em critérios morfológicos e nas características das estruturas de reprodução assexuada, sem que haja a necessidade de analisar os atributos bioquímicos e fisiológicos, como é realizado com as leveduras (WALSH; HAYDEN; LARONE, 2018). No entanto, alguns gêneros possuem grande similaridade morfológica dentre seus membros, dificultando a caraterização de suas espécies. Para essa diferenciação é necessário o uso de técnicas moleculares, como o sequenciamento genético para regiões ITS (Internal Transcribed Spacer) (WHITE et. al. 1990) e para o gene de calmodulina para o gênero Aspergillus (SABINO et al. 2015) assim como a identificação por MALDI-TOF (Matrix Assisted Lazer Desorption/Ionization – Time Of Flight Mass Spectrometry) técnica que analisa padrões de espectrometria de massa capaz de identificar linhagens fúngicas de forma extremamente rápida (ANTUNES 2019, PEREIRA 2019, PAZIANI 2021). Com o intuito de garantir a qualidade das linhagens disponibilizadas para a comunidade científica, o objetivo deste trabalho foi avaliar os diferentes métodos de manutenção e autenticação disponíveis no Núcleo, para inclusão no acervo de novas linhagens de fungos filamentosos depositados recentemente no NCMO.
MATERIAL E MÉTODOS
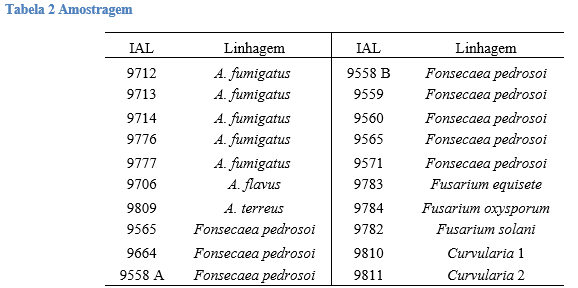
Foram utilizadas vinte linhagens depositadas no NCMO, tabela 1, pertencentes aos gêneros Aspergillus, Fusarium, Curvularia e Fonsecaea.
Identificação morfológica
Para identificação morfológica foram utilizadas às técnicas de microcultivo em lâmina e de colônia gigante, ambas em ágar dextrose batata (WALSH; HAYDEN; LARONE, 2018).
Reação de Sequenciamento
A extração do DNA genômico foi realizada utilizando o kit de extração Invitrogen PureLink® Genomic DNA mini kit (Invitrogen-Estados Unidos). Para as culturas de Aspergillus, Fusariun e Curvularia foram utilizadas culturas com 3-4 dias de crescimento em ágar dextrose batata e culturas de Fonsecaea sp foram utilizadas após 14 dias de crescimento em caldo malte. A identificação molecular foi realizada com sequenciamento Sanger para ITS utilizando os oligoiniciadores ITS1-ITS4 e ITS5-ITS4 para as vinte linhagens, e para o gene de calmodulina para as linhagens de Aspergillus. Foi utilizando o kit BigDye® Terminator v3.1 Cycle Sequencing e encaminhando a placa ao Laboratório Estratégico e analisada no equipamento 3500 Genetic Analyzer (Thermo Fisher). As sequencias foram analisadas no banco de dados GenBank, comparando com outras já depositadas
MALDI-TOF
MALDI-TOF (Matrix Assisted Lazer Desorption/Ionization – Time Of Flight Mass Spectrometry) foi utilizado para a identificação dos gêneros Aspergillus, Fusarium e Curvularia. Os fungos foram incubados em caldo Saboraud (OxoidⓇ) por três dias em banho seco com agitação (LoccusⓇ, modelo B-HS) a 26°C e 500 RPM, para se obter micélio uniforme. Esse micélio foi submetido ao processo de extração de proteínas, o produto obtido na extração foi posto em um spot da placa de MALDI-TOF (1 µL), em seguida cobertos com 1 µL de matriz HCCA. Por fim a placa com as extrações foi inserida no equipamento MALDI Biotyper® (Bruker Daltonics inc). O equipamento analisa os picos da espectrometria de massa do micro-organismo e o compara com seu banco de dados interno, a similaridade encontrada é avaliada num escore de 0,00 à 3,00, sendo os valores superiores a 2,00 considerados como de alta confidencialidade.
Métodos de conservação
Para avaliar os métodos de preservação as linhagens de A. fumigatus e A. flavus foram utilizadas para a confecção de criotubos com miçangas, usando caldo Sauton como crioprotetor e para as ampolas de liofilização leite (skim milk ) com glutamato de sódio a 5%.
RESULTADOS E DISCUSSÃO
Das vinte linhagens avaliadas no trabalho, dezoito puderam ser identificadas por ao menos uma das metodologias aplicadas, a exceção foram duas linhagens de F. pedrosoi.
Das seis linhagens enviadas para sequenciamento do gene de calmudolina somente as linhagens Aspergillus fumigatus IAL 9712 e IAL 9714, puderam ser parcialmente identificadas como A. fumigatus pela sequência de CMD 6, com 100% de similaridade para ambas. Das quinze linhagens encaminhadas para o sequenciamento para região ITS, treze puderam ser sequenciadas. Seis foram confirmadas com o depósito de origem sendo elas F. oxysporum (IAL 9783), identificada como F. oxysporum com 99,82% de similaridade e F. solani (IAL 9782) identificada como F. petroliphilum / F. solani com 99,625 de similaridade, ambas com gene consenso (ITS5-ITS4), de acordo com a base de dados Mycobank, (https://www.mycobank.org/), F. petroliphilum é uma variação de F. solani; Fonsecaea pedrosoi IAL 9560 e IAL 9664 foram parcialmente identificadas com 95,49% e 95,49% de similaridade; F. pedrosoi (IAL 9559) com 97,03% de similaridade (ITS 4); A
fumigatus (IAL 9777) identificado parcialmente pela sequencia com 100% de similaridade (ITS1). Para a confirmação dessas linhagens é preciso a utilização de novos alvos para o sequenciamento.
A linhagem de F. pedrosoi IAL 9558 apresentou resultados compatível com o gênero Phialophora sp para duas metodologias. As linhagens de Curvularia sp apresentaram como resultado duas possíveis espécies para cada amostra, uma identificada por sequenciamento do gene rDNA região ITS, outra linhagem foi parcialmente identificada, ambas tendo duas espécies como resultado, sendo diferenciadas por micromorfologia.
Todas as linhagens do gênero Aspergillus tiveram suas espécies identificadas com MALDI- TOF, correspondendo com a identificação prévia, todas com escore acima de 2,30. Para a linhagem de A. flavus o resultado foi Aspergillus oryzae group.
Já para as linhagens de Fusarium spp. todas foram identificadas como espécie (escores entre 2,13 e 2,25), contudo F. equisiti (IAL 9783) apresentou dois resultados divergentes da identificação original – F. oxysporum / F. verticilloides. A linhagem F. solani IAL 9782 apresentou como resultado F. petroliphilum, mesmo resultado do sequenciamento.
PAZIANI et. al. (2021) descreve em seu trabalho a dificuldade de identificação por MALDI- TOF de espécies de Fusarium, a diferença da expressão proteica em ambientes/meios de cultura diferentes de uma mesma espécie torna difícil sua identificação por esse método, em especial os membros do complexo Fusarium solani (FSSC). Com as linhagens de Curvularias sp. somente foi possível identificar o gênero da Amostra 2 com escore de 2,28, sem picos identificados para a Amostra 1.
Todas as linhagens apresentaram características macro e microscópicas como as descritas na literatura (WALSH; HAYDEN; LARONE et. al. 2018) e no banco de dados Mycobank. Contudo devido à grande similaridade entre algumas espécies somente essas características não são capazes de caracterizar a espécie de fungos filamentosos, sendo necessárias análises moleculares (ANTUNES 2019). A linhagem de F. pedrosoi IAL 9558 identificada pelo sequenciamento como Phialophora sp apresentou em sua micromorfologia conídios alongados terminados em forma triangular semelhante a uma taça, característico do gênero Phialophora.
Tanto o método de liofilização quanto de criopreservação mostraram-se eficazes para
Aspergillus fumigatus e A. flavus, não foi observado nenhum sinal de contaminação.
CONCLUSÃO
A identificação por sequenciamento da região ITS foi eficaz na identificação de espécies dos gêneros Fusarium, Fonsecaceae, Phialophora, Curvularia e para Aspergillus fumigatus. A. flavus IAL 9706 e A. terreus (sem IAL) não puderam ser identificados por sequenciamento para ITS assim como para gene da calmodulina, outros alvos são necessários para a caracterização completa dessas linhagens. A identificação por MALDI-TOF não foi capaz de caracterizar a linhagem A. flavus IAL 9706 como espécie; somente como grupo.
As espécies do gênero Aspergillus podem ser identificadas por MALDI-TOF. Para Curvularia a identificação somente foi possível em gênero, sendo necessário adaptar o método de extração de proteínas.
O gênero Fusarium trabalhado nesse estudo se mostrou mais difícil de identificar quanto suas espécies devido a grande similaridade dentre os membros de um mesmo complexo.
Somente a morfologia das linhagens não é capaz de caracterizar fungos em espécies, mas podem auxiliar na caracterização de gênero combinados com métodos de biologia molecular. É necessário autenticar toda linhagem depositada para confirmar as informações originais de depósito.
A liofilização com leite e glutamato de sódio a 5% e a criopreservação em miçangas usando caldo Sauton são eficazes na preservação de linhagens do gênero Aspergillus;
As metodologias de autenticação aplicadas se mostraram eficientes para a maioria das linhagens estudadas. Das 20 linhagens trabalhadas, 18 puderam ser identificadas por pelo menos um método proposto.
REFERENCIAS BIBLIOGRÁFICAS
ANTUNES, Fernanda Marques Americo. Avaliação da metodologia de espectrometria de massas MALDI-TOF (VITEK MS®) para identificação de espécies de Aspergillus de importância médica. 2019. Dissertação (Mestrado em Dermatologia) – Faculdade de Medicina, University of São Paulo, São Paulo, 2019. doi:10.11606/D.5.2019.tde-05062019-092951. Acesso em: 2022-11-23.
BOUNDY‐MILLS, K. et al. Preserving US microbe collections sparks future discoveries. Journal of Applied Microbiology, v. 129, n. 2, p. 162-174, 2020.
COSTA, Edmara Chaves et al. Princípios da estocagem e preservação de amostras microbiológicas. Ciência Animal, v. 19, n. 2, p. 111-122, 2009.
OLIVEIRA, Luciana Aires; CORTEZ, Ana Cláudia A.; SOUZA, João Vicente Braga de. Avaliação de metodologias de preservação para manutenção de microrganismos do Filo Ascomycota e leveduras do gênero Candida pertencentes à Coleção de Microrganismos de Interesse Médico do INPA. Diversidade Microbiana da Amazônia, pgs. 58-62, 2016.
PAZIANI, Mario Henrique. Perfil fenotípico e genotípico de Fusarium spp. provenientes de episódios clínicos de fusarioses no Estado de São Paulo. 2021. Tese (Doutorado em Bioagentes e Biotecnologia Aplicados à Farmácia) – Faculdade de Ciências Farmacêuticas de Ribeirão Preto, Universidade de São Paulo, Ribeirão Preto, 2021. doi:10.11606/T.60.2021.tde-29092021-053538. Acesso em: 2022-11-28.
PEREIRA, Anielli Souza. Diversidade e potencial para a produção de enzimas de Penicillium, Aspergillus, Talaromyces e Paecilomyces isolados de solo de Mata Atlântica do Quadrilátero Ferrífero. 2019. 101f. Tese (Doutorado em Microbiologia Agrícola) – Universidade Federal de Lavras, Programa de Pós-Graduação em Microbiologia Agrícola, Lavras, 2019.
SABINO, R. et al. Molecular epidemiology of Aspergillus collected from cystic fibrosis patients. Journal of Cystic Fibrosis, v. 14, n. 4, 2015.
SETTE, Lara Durães; PAGNOCCA, Fernando Carlos; RODRIGUES, André. Microbial culture collections as pillars for promoting fungal diversity, conservation and exploitation. Fungal Genetics and Biology, v. 60, p. 2-8, 2013.
WALSH, T. J.; HAYDEN, R. T.; LARONE, D. H. Larone’s Medically Important Fungi. 6o ed. Washington: [2018]
WHITE, Thomas J. et al. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. PCR protocols: a guide to methods and applications, v. 18, n. 1, p. 315-322, 1990.
