GENES DE RESISTÊNCIAAOS ANTIMICROBIANOS EM Escherichia coli ISOLADAS DE ÁGUAS DESTINADAS AO CONSUMO HUMANO
- Home
- /
- Blog
- /
- Livros
- /
- Capítulo de Livro IV Mostra
- /
- GENES DE RESISTÊNCIAAOS ANTIMICROBIANOS EM Escherichia coli ISOLADAS DE ÁGUAS DESTINADAS AO CONSUMO HUMANO
Capítulo de livro publicado no Livro da IV Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
doi.org/10.53934/9786599965821-43
Este trabalho foi escrito por:
Natália Roberto Faria da Silva¹; Ruth Estela Gravato Rowlands²; Cecília Geraldes Martins²; Christiane Asturiano Ristori Costa²
1Estudante do Curso de Vigilância Laboratorial em Saúde Pública – IAL Central; E-mail: [email protected]
2Pesquisador do Núcleo de Microbiologia – IAL Central
Resumo: A resistência antimicrobiana representa uma ameaça sanitária global crescente e deve ser abordada com urgência e de forma integrada (saúde humana, animal e meio ambiente) em virtude do risco de tratamento ineficaz de infecções causadas por microrganismos, resultando em enfermidades mais prolongadas e com maior mortalidade. Existem dois mecanismos capazes de atribuir resistência, por mutações no cromossomo ou transferência horizontal entre bactérias de genes presentes em plasmídeos, integrons ou transposons. O integron de classe 1 é um dos mecanismos mais comuns que confere resistência aos antibióticos como β-lactâmicos, cloranfenicol, sulfonamidas, aminoglicosídeos, entre outros. Atualmente, as cepas de Escherichia coli são consideradas importantes reservatórios de genes de resistência. O objetivo do presente estudo foi pesquisar genes de resistência aos antimicrobianos (blaCTX-M-15, blaOXA-23, erm(B), sul1, tetA e tetB) e o integron de classe 1 (intl1), pela técnica de reação em cadeia da polimerase, em cepas de E. coli isoladas de amostras de água para consumo humano, no estado de São Paulo. Do total de cepas analisadas (n=50), 10% apresentou ao menos um dos seis genes de resistência, sendo tetA (6%) o de maior ocorrência seguido do tetB (4%) e sul1 (2%). Uma cepa (2%) isolada de água de poço apresentou positividade, simultaneamente, para os genes tetB e sul1. Os genes blaCTX-M-15, blaOXA-23 e erm(B) não foram detectados. O gene do elemento genético móvel intl1 foi encontrado em um isolado (2%). Os resultados encontrados demonstram que E. coli isoladas de água de fontes alternativas podem disseminar genes de resistência e elementos genéticos associados à expressão de resistência aos antimicrobianos.
Palavras–chave: Escherichia coli, Resistência a antibióticos, Integrons, Água para consumo humano.
INTRODUÇÃO
O acesso à água limpa e segura é considerado um recurso essencial para a vida, entretanto, no Brasil, 15,9% da população não tem acesso à água tratada (SNIS, 2020). Em regiões sem acesso ao sistema de abastecimento público é comum o uso de poços, minas e nascentes, porém tais fontes podem representar risco à população, uma vez que pode haver a contaminação pelo escoamento de águas de terras agrícolas e depósito de efluentes sem tratamento (CABRAL, 2010; FAYER; MORGAN; UPTON, 2000).
No Brasil, a avaliação da qualidade microbiológica da água é realizada por meio da pesquisa de Escherichia coli (BRASIL, 2021). A bactéria é amplamente utilizada como indicador de contaminação fecal em amostras de água e sua presença pode estar associada à presença de microrganismos patogênicos (NAVAB-DANESHMAND et. al., 2018).
A resistência antimicrobiana (RAM) representa uma das maiores ameaças à saúde global e deve ser abordada de forma integrada (saúde humana, animal e meio ambiente) como conceito de Saúde Única ou One Health (WHO, 2021). Estima-se que 1,27 milhão de mortes ocorreram em decorrência da RAM e as bactérias responsáveis pela maioria das infecções foram: E. coli, Staphylococcus aureus, Klebsiella pneumoniae, Staphylococcus pneumoniae, Acinetobacter baumannii e Pseudomonas aeruginosa (MURRAY et al., 2022).
E. coli têm sido descritas como reservatórios de genes de resistência e, em ambientes aquáticos, desempenham papel importante na disseminação de resistência entre bactérias patogênicas e comensais, o que gera preocupação para a saúde pública. Considerando a escassez de dados sobre o tema em cepas isoladas de água de consumo e a importância epidemiológica de cepas resistentes aos antimicrobianos, o presente estudo teve como objetivo pesquisar genes de resistência aos antimicrobianos e o integron de classe 1 em cepas de E. coli isoladas de amostras de água para o consumo humano.
MATERIAL E MÉTODOS
Cepas
Foram analisadas 50 cepas de E. coli provenientes de 50 amostras de água para o consumo humano, sendo 32 de água de poço, 13 de mina, 3 de nascente e 2 de abastecimento público. As amostras foram coletadas no Estado de São Paulo, no período de 2010 a 2011 e analisadas para avaliação da potabilidade no Núcleo de Microbiologia, do Instituto Adolfo Lutz.
O isolamento de E. coli foi realizado a partir dos métodos de presença-ausência ou substrato enzimático cromogênico e fluorogênico e a identificação bioquímica da espécie realizada por meio do IMViC (APHA, 2005). Os 50 isolados de E. coli foram testados pela técnica de reação em cadeia de polimerase para detecção de cepas diarreiogênicas e apenas uma, proveniente de água de poço, foi identificada como E. coli enteropatogênica (EPEC) típica (DE MELLO, 2014). As cepas foram armazenadas em Brain Heart Infusion (BHI) com glicerol, a -20°C.
Reação em cadeia de polimerase (PCR)
A extração de DNA foi efetuada utilizando kit de extração QIAamp® DNA Mini Kit (QiagenTM, USA), seguindo as instruções do fabricante. Os extraídos de DNA foram armazenados a -20ºC até o momento de uso. A pesquisa dos genes de resistência blaCTX-M-15, blaOXA-23, erm(B), tetA, tetB e do integron da classe 1 intl1 foi realizada pela PCR. Os genes tetA e tetB, blaCTX-M-15, blaOXA-23, erm(B) e intl1 foram pesquisados segundo metodologias descritas na literatura (Randall et al., 2004; Liaqat et al., 2022; Pajand et al., 2013; Knapp et al., 2010; Barraud et al., 2010), com modificações. Como controle positivo foram utilizadas cepas positivas para os genes estudados e como controle negativo água ultrapura livre de DNases e RNases. Os produtos amplificados foram revelados em gel de agarose (UltrapureTM, InvitrogenTM, EUA) a 1,5% em tampão TBE 1x, contendo 0,5 µL de brometo de etídio. A eletroforese foi realizada a 100V/ 60 minutos em cuba horizontal contendo TBE 1x e o gel foi visualizado em transiluminador de luz UV.
RESULTADOS E DISCUSSÃO
Do total de 50 cepas analisadas de E. coli isoladas de água para consumo humano, cinco (10%) apresentaram pelo menos um dos genes de resistência estudados. Dentre estas, a maior ocorrência foi observada em cepas isoladas de amostras de água de nascente (66,7%), seguido de poço (12,5%) e mina (7,69%). Nas amostras de água de abastecimento público nenhum gene foi detectado (Tabela 1). A cepa de E. coli diarreiogênica, EPEC, avaliada não apresentou nenhum dos genes de resistência e nem do integron, provavelmente porque a resistência antimicrobiana está significantemente associada à ausência de genes de virulência (SUKUMARAN; MOHAMED HATHA, 2015).
Uma cepa (2%) isolada de água de poço apresentou positividade, simultaneamente, para os genes tetB e sul1. A presença de E. coli multirresistente em ambiente aquático pode favorecer a disseminação de genes de resistência para diferentes ambientes e microrganismos e dificultar o tratamento de infecções no homem e nos animais (TALUKDAR et al., 2013).
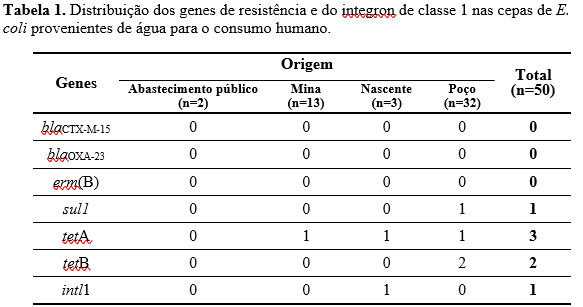
Os genes de resistência à tetraciclina, tetA e tetB foram os de maior ocorrência com 6% e 4% , respectivamente. A resistência à tetraciclina é amplamente observada em ambientes naturais e também em água potável (ARAÚJO et al., 2017; CHATURVEDI et al., 2021; FALGENHAUER et al., 2021). Na medicina veterinária, as tetraciclinas são amplamente utilizadas e o contato próximo de ambiente aquáticos com o solo contaminado com estrume dos animais pode favorecer alta pressão seletiva exercida sobre o desenvolvimento de resistência e a disseminação mediada por elementos móveis na água (AGERSØ; SANDVANG, 2005; WOAH, 2020).
Com relação ao gene sul1, a ocorrência observada foi de 2%, a menor entre os genes detectados no presente estudo. Resultado semelhante foi encontrado em amostras de água coletadas de torneiras de residências na Escócia, em que 5,4% das cepas continham o gene (KHAN; KNAPP; BEATTIE, 2016). Por outro lado, uma alta ocorrência (95,8%) foi relatada por Amarasiri et al. (2022), em amostras de água de poços e nascentes do Nepal, possivelmente pelos locais de coleta serem próximos de efluentes de farmácias e açougues. Quanto à pesquisa do gene intl1, que codifica o integron de classe 1, 2% das cepas analisadas carreavam o gene. Ozgumus et al. (2007) obteve percentual similar, detectando intl1 em 2,5% dos isolados de E. coli. Resultados diferentes foram relatados por Ramalho et al. (2022), que não detectaram o gene em amostras de água potável pós tratamento no sul do Brasil e Amarasiri et al. (2022) que observaram uma ocorrência significativa de 87,5% nas amostras de água de poços e nascentes no Nepal.
Vale ressaltar, que a cepa que carreava o gene intl1 também apresentava o gene tetA, entretanto, dentro dos limites do estudo, não foi possível afirmar que o isolado tenha adquirido o gene de resistência à tetraciclina por meio do integron. Mas, diversos estudos tem demonstrado que intl1 pode desempenhar papel fundamental na disseminação de genes de resistência em água potável (KOCZURA et al., 2016; ZHANG et al., 2020; ZHAO et al., 2001).
Na presente pesquisa não foi observada a presença simultânea do gene sul1 com o intl1, sugerindo que o integron de classe 1 encontrado em uma das amostras se trata, possivelmente, de um não clássico, forma em que a presença do gene de resistência à sulfonamida não é obrigatória (SU et al., 2012).
Nenhuma cepa de E. coli analisada apresentou os genes blaCTX-M-15 e blaOXA-23 que codificam a resistência à β-lactâmicos. Talukdar et al. (2013) obtiveram resultado distinto ao analisar isolados de E. coli de amostras de água de torneiras de uma comunidade em Bangladesh, sendo que dos 22 isolados positivos para ESBL, 20 apresentaram o gene blaCTX- M-15. Estudos vêm indicando o aumento de β-lactamases do tipo CTX-M em Enterobacteriaceae e, em algumas regiões, já é predominante (EJAZ et al., 2021; FALAGAS; KARAGEORGOPOULOS, 2009; PAVEZ et al., 2019) portanto, a pesquisa desses genes se faz necessária.
Não foram encontrados estudos sobre a pesquisa do gene blaOXA-23 em água para consumo humano, apenas em amostras de água de rios e águas residuais, nos quais não foi detectado (AZAM et al., 2018; SHAMSIZADEH et al., 2021), demonstrando a baixa ocorrência em ambiente aquático.
O gene erm(B) também não foi detectado entre as cepas estudadas, o que corrobora com os resultados de outros estudos com cepas isoladas de água para o consumo humano (BERGERON et al., 2015; RAMALHO et al., 2022).
CONCLUSÃO
Os resultados encontrados demonstram que E. coli isoladas de água de fontes alternativas podem disseminar genes de resistência e elementos genéticos associados à expressão de resistência aos antimicrobianos. Portanto, o monitoramento do perfil de resistência em microrganismos oriundos de ambientes aquáticos é fundamental para uma melhor compreensão dos mecanismos envolvidos na resistência e na sua disseminação, além de poder contribuir para a implementação de estratégias de prevenção e controle com foco no conceito de Saúde Única (saúde humana, animal e meio ambiente).
AGRADECIMENTOS
Os autores agradecem aos pesquisadores Dr. Carlos Henrique Camargo e Dr. Luis Fernando dos Santos, do Núcleo de Doenças Entéricas e Infecções por patógenos Especiais, do Centro de Bacteriologia e Dra. Tânia Sueli de Andrade, do Núcleo de Coleção de Micro- organismos, do Instituto Adolfo Lutz, pelo auxílio na obtenção das cepas utilizadas como controle positivo para os genes estudados.
REFERÊNCIAS
AGERSØ, Y.; SANDVANG, D. Class 1 Integrons and Tetracycline Resistance Genes in Alcaligenes, Arthrobacter, and Pseudomonas spp. Isolated from Pigsties and Manured Soil. Applied and Environmental Microbiology, v. 71, n. 12, p. 7941-7947, 2005.
AMARASIRI, M. et al. Prevalence of antibiotic resistance genes in drinking and environmental water sources of the Kathmandu Valley, Nepal. Frontiers in Microbiology, v. 13, 2022.
AMERICAN PUBLIC HEALTH ASSOCIATION (APHA). Standard Methods for the Examination of Water and Wastewater. 2.ed. New York: American Public Health Association, 2005.
ARAÚJO, S. et al. Characterization of antibiotic resistant and pathogenic Escherichia coli in irrigation water and vegetables in household farms. International Journal of Food Microbiology, v. 257, p. 192-200, 2017.
AZAM, M. et al. Study of pandrug and heavy metal resistance among E. coli from anthropogenically influenced Delhi stretch of river Yamuna. Brazilian Journal of Microbiology, v. 49, n. 3, p. 471-480, 2018.
BARRAUD, O. et al. Quantitative multiplex real-time PCR for detecting class 1, 2 and 3 integrons. Journal of Antimicrobial Chemotherapy, v. 65, n. 8, p. 1642-1645, 2010.
BERGERON, S. et al. Presence of antibiotic resistant bacteria and antibiotic resistance genes in raw source water and treated drinking water. International Biodeterioration & Biodegradation, v. 102, p. 370-374, 2015.
BRASIL. MINISTÉRIO DA SAÚDE (MS). Portaria nº 888, de 04 de maio de 2021. Portaria GM/MS Nº 888. Brasília, 2021.
CABRAL, J.P.S. Water Microbiology. Bacterial Pathogens and Water. International Journal of Environmental Research and Public Health, v. 7, n. 10, p. 3657-3703, 2010.
CHATURVEDI, P. et al. Occurrence of emerging sulfonamide resistance (sul1 and sul2) associated with mobile integrons-integrase (intI1 and intI2) in riverine systems. Science of the Total Environment, v. 751, e 142217 2021.
DE MELLO, L.S.R. Qualidade bacteriológica e ocorrência de Escherichia coli patogênicas em amostras de água para o consumo humano. Trabalho de Conclusão de Curso (Programa de Aprimoramento Profissional) – IAL, São Paulo, 2014.
EJAZ, H. et al. Molecular analysis of blaSHV, blaTEM, and blaCTX-M in extended-spectrum β-lactamase producing Enterobacteriaceae recovered from fecal specimens of animals. Plos One, v. 16, n. 1, e0245126, 2021.
FALAGAS, M.E.; KARAGEORGOPOULOS, D.E. Extended-spectrum β-lactamase-producing organisms. Journal of Hospital Infection, v. 73, n. 4, p. 345-354, 2009.
FALGENHAUER, L. et al. Clonal CTX-M-15-Producing Escherichia coli ST-949 Are Present in German Surface Water. Frontiers In Microbiology, v. 12, e 617349, 2021.
FAYER, R.; MORGAN, U.; UPTON, S.J. Epidemiology of Cryptosporidium: transmission, detection and identification. International Journal for Parasitology, v. 30, n. 12-13, p. 1305- 1322, 2000.
KHAN, S.; KNAPP, C.W.; BEATTIE, T.K. Antibiotic Resistant Bacteria Found in Municipal Drinking Water. Environmental Processes, v. 3, n. 3, p. 541-552, 2016.
KNAPP, C.W. et al. Differential fate of erythromycin and beta-lactam resistance genes from swine lagoon waste under different aquatic conditions. Environmental Pollution, v. 158, n. 5, p. 1506-1512, 2010.
KOCZURA, R. et al. Abundance of Class 1 Integron-Integrase and Sulfonamide Resistance Genes in River Water and Sediment Is Affected by Anthropogenic Pressure and Environmental Factors. Microbial Ecology, v. 72, n. 4, p. 909-916, 2016.
LIAQAT, Z. et al. Isolation and molecular characterization of extended spectrum beta lactamase producing Escherichia coli from chicken meat in Pakistan. Plos One, v. 17, n. 6, e0269194, 2022.
MURRAY, C.J.L. et al. Global burden of bacterial antimicrobial resistance in 2019: a systematic analysis. The Lancet, v. 399, n. 10325, p. 629-655, 2022.
NAVAB-DANESHMAND, T. et al. Escherichia coli Contamination across Multiple Environmental Compartments (Soil, Hands, Drinking Water, and Handwashing Water) in Urban Harare: correlations and risk factors. The American Journal of Tropical Medicine and Hygiene, v. 98, n. 3, p. 803-813, 2018.
OZGUMUS, O.B. et al. Molecular Characterization of Antibiotic Resistant Escherichia coli Strains Isolated from Tap and Spring Waters in a Coastal Region in Turkey. Journal of Microbiology, v. 45, n. 5, p. 379-387, 2007.
PAJAND, O. et al. Study of the carbapenem resistance mechanisms in clinical isolates of Acinetobacter baumannii: comparison of burn and non-burn strains. Burns, v. 39, n. 7, p. 1414- 1419, 2013.
PAVEZ, M. et al. High prevalence of CTX-M-1 group in ESBL-producing enterobacteriaceae infection in intensive care units in southern Chile. The Brazilian Journal of Infectious Diseases, v. 23, n. 2, p. 102-110, 2019.
RAMALHO, R. et al. The occurrence of antimicrobial residues and antimicrobial resistance genes in urban drinking water and sewage in Southern Brazil. Brazilian Journal of Microbiology, v. 53, n. 3, p. 1483-1489, 2022.
RANDALL, L.P. et al. Antibiotic resistance genes, integrons and multiple antibiotic resistance in thirty-five serotypes of Salmonella enterica isolated from humans and animals in the UK. Journal of Antimicrobial Chemotherapy, v. 53, n. 2, p. 208-216, 2004.
SHAMSIZADEH, Z. et al. Tracking antibiotic resistance genes and class 1 integrons in Escherichia coli isolates from wastewater and agricultural fields. Water Science and Technology, v. 84, n. 5, p. 1182-1189, 2021.
SISTEMA NACIONAL DE INFORMAÇÕES SOBRE SANEAMENTO (SNIS). Abastecimento de Água: 2020. 2020. Disponível em: http://www.snis.gov.br/painel- informacoes-saneamento-brasil/web/painel-abastecimento-agua. Acesso em: 11 out. 2022.
SU, H. et al. Class 1 and 2 integrons, sul resistance genes and antibiotic resistance in Escherichia coli isolated from Dongjiang River, South China. Environmental Pollution, v. 169, p. 42-49, 2012.
SUKUMARAN, D.; MOHAMED HATHA, A.A. Antibiotic resistance and virulence genes of extraintestinal pathogenic Escherichia coli from tropical estuary. The Journal of Infection in Developing Countries, v. 9; n. 5, p. 496-504, 2015.
TALUKDAR, P.K. et al. Antimicrobial Resistance, Virulence Factors and Genetic Diversity of Escherichia coli Isolates from Household Water Supply in Dhaka, Bangladesh. Plos One, v. 8, n. 4, 2013.
WORLD HEALTH ORGANIZATION (WHO). Global antimicrobial resistance and use surveillance system (GLASS): report 2021. Genebra, 2021.
WORLD ORGANIZATION FOR ANIMAL HEALTH (WOAH). 2020. OIE annual report on antimicrobial agents intended for use in animals – fourth report. Paris, 2020.
ZHANG, K. et al. Antibiotic Resistance Genes in drinking water of China: occurrence, distribution and influencing factors. Ecotoxicology and Environmental Safety, v. 188, e109837, 2020.
ZHAO, S. et al. Identification and Characterization of Integron-Mediated Antibiotic Resistance among Shiga Toxin-Producing Escherichia coli Isolates. Applied and Environmental Microbiology, v. 67, n. 4, p. 1558-1564, 2001.
