PRESERVAÇÃO E AUTENTICAÇÃO DE LINHAGENS GRAM- NEGATIVAS DO ACERVO DO NÚCLEO DE COLEÇÃO DE MICRO- ORGANISMOS DO INSTITUTO ADOLFO LUTZ
- Home
- /
- Blog
- /
- Livros
- /
- Capítulo de Livro IV Mostra
- /
- PRESERVAÇÃO E AUTENTICAÇÃO DE LINHAGENS GRAM- NEGATIVAS DO ACERVO DO NÚCLEO DE COLEÇÃO DE MICRO- ORGANISMOS DO INSTITUTO ADOLFO LUTZ
Capítulo de livro publicado no Livro da IV Mostra dos Trabalhos de Conclusão de Curso da Especialização em Vigilância Laboratorial em Saúde Pública. Para acessa-lo clique aqui.
doi.org/10.53934/9786599965821-22
Este trabalho foi escrito por:
Letícia de Carvalho dos Santos1, Elizabeth Harummyy Takagi2
1 Estudante do Curso de Vigilância Epidemiológica em Saúde Pública – NCMO – Instituo Adolfo Lutz. [email protected]
2Docente/pesquisadora do Depto de Coleção de Micro-organismos – NCMO.
RESUMO
Os micro-organismos desempenham um importante papel na manutenção da vida no planeta devido à sua imensa biodiversidade, fornecendo recursos ilimitados para o desenvolvimento de pesquisas e criação de novos conhecimentos. O presente estudo tem como objetivo realizar a autenticação de nove linhagens bacterianas preservadas pelo método de liofilização no Núcleo de Coleção de Micro-organismos (NCMO) do Instituto Adolfo Lutz. Como metodologias foram adotadas técnicas bioquímicas fenotípicas e técnicas moleculares como MALDI-TOF e sequenciamento do gene codificante da porção 16S ribossomal. Duas linhagens de Acinetobacter baumanni foram caracterizadas por provas bioquímicas, MALDI-TOF, sequenciamento e amplificação do gene codificador da beta-lactamase do subgrupo OXA-51. Quatro linhagens da bactéria Citrobacter sp., uma linhagem de Edwardsiella tarda, uma linhagem de Klebsiella sp e uma linhagem de Raoultella planticolla foram autenticadas com o uso do MALDI-TOF complementado com provas bioquímicas. Ao término do estudo, as linhagens autenticadas ficaram disponíveis para fornecimento público, permitindo seu uso em pesquisas de grande relevância, em trabalhos industriais e controle de qualidade em Laboratórios Centrais de Saúde Pública (LACENs), contribuindo com a vigilância epidemiológica e consequentemente fazendo parte da história da saúde pública do país.
Palavras-chave: Autenticação, Bactérias Gram-negativas, Coleção, MALDI, Saúde Pública
INTRODUÇÃO
Os micro-organismos desempenham um papel significativo na manutenção da vida no planeta devido à sua imensa biodiversidade, fornecendo recursos ilimitados para o desenvolvimento de pesquisas e criação de novos conhecimentos (JANSSENS, D. et al., 2010).
Os centros de coleção de cultura atuam como depósitos de linhagens para fins de processamento de patentes no campo de biotecnologia, preservando os micro-organismos de forma confidencial e segura, disponibilizando-os após o período de sigilo, além de fornecer linhagens usadas em pesquisas para confirmações de resultados (SMITH, 2003).
O Núcleo de Coleção de Micro-organismos (NCMO) do Instituto Adolfo Lutz atua como coleção de serviço, contando com mais de dez mil linhagens de importância em saúde armazenadas desde a década de 40 em acervo, conservando e distribuindo materiais biológicos com critérios reconhecidos para indústrias e pesquisas (ANDRADE, T. S. et al, 2008). No entanto, devido à diversidade do acervo e tempo de armazenamento muitas linhagens ainda necessitam de autenticação por novas metodologias de identificação e caracterização, por isso o presente trabalho visa autenticar algumas linhagens de bactérias Gram negativas depositadas há anos, sem caracterização anterior, garantindo desta forma o fornecimento futuro de material com qualidade assegurada e contribuindo na saúde pública e vigilância epidemiológica.
As linhagens abordadas no presente trabalho são importantes patógenos associados às Infecções Relacionadas à Assistência à Saúde (IRAS), sendo elas: Acinetobacter baumannii, Citrobacter sp., Edwardsiella tarda, Klebsiella sp. e Raoultella sp.
MATERIAL E MÉTODOS
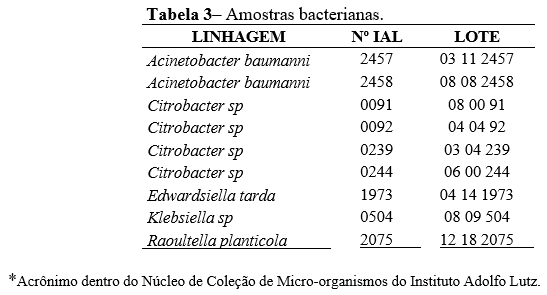
Foram selecionadas nove linhagens de bactérias Gram-negativas conservadas pelo método de liofilização, descritas na Tabela 3.
Foi realizada a análise macroscópica através da observação do crescimento dos micro- organismos em repique em placa de ágar comum, incubada por 24 horas a 37 ºC. A análise microscópica foi analisada por coloração de Gram e a leitura em aumento de 1000x.
A autenticação foi realizada por três métodos: MALDI-TOF (espectrometria de massa por ionização com dessorção a laser assistido por matriz e analisador de tempo de voo), avaliação bioquímica e sequenciamento Sanger.
A identificação por MALDI-TOF foi realizada por extração direta conforme estabelecido pelo fabricante Bruker. As proteínas do crescimento bacteriano depositado na placa do MALDI-TOF foram extraídas com ácido fórmico 95%, aplicando uma matriz HCCA (α- cyano-4-hydroxycinnamic Acid) que sofreu ionização pela incidência de um feixe de laser do equipamento e estabeleceu um espectograma de massa para leitura das proteínas.
Para as linhagens de Acinetobacter baumannii foram realizadas as provas bioquímicas: malonato, glicose, oxidase, lactose, hemólise em ágar sangue, degradação da gelatina, indol, sacarose, H2S, ureia e lisina. Para as enterobactérias foram feitas as análises de: nitrato, lisina, malonato, ornitina, ureia, citrato, glicose, oxidase, catalase, lactose, indol, sacarose, H2S, motilidade e dulcitol.
A interpretação dos resultados foi realizada através da mudança de cores dos meios pelo indicador de pH. A assimilação de carboidratos torna o meio ácido, gerando alteração de cor e sinalizando um resultado positivo. Nas provas de assimilação de aminoácidos, o meio se torna básico pela geração de um grupo amina e a cor permanece a mesma, indicando a positividade.
Para avaliação bioquímica também foi utilizado o Sistema Bactray I, seguindo as orientações do fabricante para execução e leitura dos resultados. Cada linhagem foi inoculada em água ultrapura, obtendo turvação final nº 0,5 na escala de McFarland , e dispensada no painel de provas direcionando aos depósitos de substrato. A leitura foi realizada após 24h de incubação a 37 ºC em condições de umidade 40-60%. Os resultados foram interpretados pela alteração da coloração dos substratos.
Foram realizadas reações em cadeia polimerase (PCR) com o objetivo de amplificar o material genômico das amostras a serem analisadas por sequenciamento. O alvo escolhido foi a porção do gene codificante 16S ribossomal com o uso dos oligoiniciadores 27F / 518R e 341F / 1492R para todas as amostras.
Para as linhagens de Acinetobacter baumannii também foi realizado PCR utilizando os oligoiniciadores OXA-51 F e OXA-51 R, que são específicos para esta espécie e auxiliam no direcionamento da identificação (TURTON, J. F. et al., 2006).
A reação do sequenciamento foi realizada com o uso do BigDye™ Terminator v3.1 Cycle Sequencing Kit. As amostras foram sequenciadas pelo aparelho 3500 Genetic Analyzer (ThermoFisher).
RESULTADOS E DISCUSSÃO MALDI-TOF
Os resultados do MALDI-TOF são classificados de acordo com a pontuação conferida pela pesquisa em banco de dados da Bruker, sendo a pontuação de 2,00 a 3,00 considerada uma identificação de alta confiança, 1,70 a 1,99 identificações de baixa confiabilidade e abaixo de 1,69 sem identificação.
A leitura dos padrões de proteína das linhagens de Acinetobacter baumanii, Citrobacter sp. e Raoultella planticola apresentaram diferentes resultados com um score superior a 2,0 e por isso não foi possível chegar à uma identificação conclusiva por essa técnica.
Para a linhagem Edwardsiella tarda foi concluída a identificação de gênero e espécie, com um score de 2,16. O IAL 504 Klebsiella sp. foi identificado como Klebsiella pneumoniae, com score 2,33, apresentando duas identificações para subespécie com scores considerados de alta confiança.
PERFIL BIOQUÍMICO
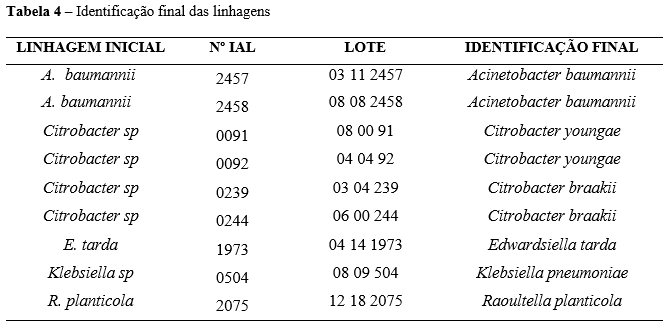
O resultado das provas para Acinetobacter baumannii foi compatível com o perfil do gênero e espécie (ANVISA, 2004), exceto pelo consumo do malonato da linhagem A. baumanni IAL 2458 que foi negativo.
Para a identificação das linhagens de Citrobacter foi utilizado o fluxograma desenvolvido por O’Hara e colaboradores (O’HARA, C. M. et al, 1995). Através da comparação dos resultados obtidos com o fluxograma, foi possível determinar que as linhagens IAL 239 e IAL 244 são da espécie braakii, enquanto o IAL 91 e IAL 92 são da espécie youngae.
Os resultados das provas bioquímicas direcionaram a identificação do IAL 1973 para Edwardsiella tarda e do IAL 504 como Klebsiella pneumoniae.
Segundo Walckenaer et al. (2008), isolados de R. planticola são negativos para ODC (ornitina) e variáveis para a produção de indol, enquanto a R. ornithinolytica é positiva para o teste de descarboxilase da ornitina e obrigatoriamente não produz indol. Considerando que a linhagem 2075 apresentou resultado negativo para a prova de ornitina e positivo para indol, foi possível concluir que esta linhagem pertence à espécie R. plancitcola..
BACTRAY
O sistema Bactray I é direcionado para bactérias Gram-negativas que são oxidase negativa. Os resultados foram analisados na plataforma online Bactray, sendo considerado um resultado aceitável aqueles com semelhança acima de 80%.
O sistema determinou 76,35% de semelhança com Acinetobacter baumannii para a linhagem 2457, sendo que para um perfil com 100% de semelhança pelo sistema, a bactéria deveria ser capaz de transformar fenilalanina em ácido fenilpirúvico. Contudo, a literatura demostra que apenas 87% das linhagens de A. baumannii apresentam positividade para esta prova (HOLT, J. G. 1994), levantando então o questionamento da assertividade desta plataforma na análise desta espécie. Não foi possível identificar o IAL 2458 com este sistema, apresentando diferentes relações com resultados abaixo de 50%.
As linhagens de Citrobacter sp., IAL 91 e IAL 92, apresentaram 99,97% de compatibilidade com Citrobacter freundii. Todavia, o sistema Bactray I não tem em sua composição o substrato “dulcitol”, prova importante para diferenciar as espécies C. freundii e C. youngae, e por isso este resultado não pode ser considerado como final sem a prova complementar citada.
A linhagem de Citrobacter sp. IAL 239 chegou a uma identificação com 100% de similaridade para a espécie C. braakii e o IAL 244 apresentou 97,76% de semelhança para C. braakii.
Para a linhagem IAL 1973, foi observado semelhança de 99,94% para Edwardsiella tarda, correspondendo à identificação original. Para a linhagem Klebsiella sp., IAL 504, foi observada semelhança de 97,63% para a identificação de Klebsiella pneumoniae.
Não foi possível concluir a identificação com este sistema para a linhagem de Raoultella planticola, pois este gênero não é abordado pelo Bactray I.
SEQUENCIAMENTO
Devido a não leitura das duas cadeias de polinucleotídeos das amostras, os resultados obtidos neste processo de sequenciamento não foram decisivos para concluir a autenticação das linhagens, tornando necessária a associação deste resultado com os demais obtidos pelas técnicas anteriores.
OXA-51
O resultado da PCR feita com os oligoiniciadores OXA-51F e OXA-51R foi visualizado em gel de Agarose 1,5%, apresentando bandas claras e únicas e confirmando a espécie das linhagens trabalhadas, pois, de acordo com Turton (2006), o gene codificante da beta- lactamase do subgrupo OXA-51 é intrínseco das linhagens de Acinetobacter baumannii.
IDENTIFICAÇÃO FINAL DAS LINHAGENS
Após a realização das técnicas de autenticação, bioquímicas e moleculares, a compilação dos resultados permitiu caracterizar as linhagens para gênero e espécie, como pode ser observado na Tabela 4.
CONCLUSÃO
As provas bioquímicas, convencionais e do Sistema Bactray, foram úteis na identificação das bactérias Gram-negativas em conjunto com outras técnicas complementares. Neste trabalho, a caracterização fenotípica foi fundamental no direcionamento da identificação das espécies das linhagens de Citrobacter sp e Raoultella planticolla., que em conjunto com as demais técnicas permitiu a conclusão da autenticação.
A técnica de MALDI-TOF foi capaz de concluir a identificação de gênero e espécie das linhagens Edwardsiella tarda e Klebsiella pneumoniae e auxiliou no direcionamento das espécies das demais linhagens, como no caso da Raoultella planticolla e espécies de Citrobacter sp.
Não foi possível concluir a identificação das linhagens através do sequenciamento devido a não leitura das duas cadeias de polinucleotídeos das amostras, não formando um consenso. Todavia, não se pode determinar que este não é um bom método de autenticação das bactérias Gram-negativas pois a falha na classificação pode ter ocorrido devido à falhas do desenvolvimento do trabalho, necessitando confirmar os resultados.
A amplificação do gene OXA-51, possibilitou a identificação específica das amostras de A. baumannii (IAL 2457 e IAL 2458) em razão de sua inerência para com a espécie.
Ao final dos procedimentos, as linhagens autenticadas passaram a estar disponíveis para fornecimento, permitindo seu uso em pesquisas de grande relevância, em trabalhos industriais e em Laboratórios Centrais de Saúde Pública (LACENs), contribuindo com a vigilância epidemiológica e consequentemente fazendo parte da história da saúde pública do país.
REFERÊNCIAS
ANDRADE, T. S. et al. Seção de Coleção de Culturas do Instituto Adolfo Lutz – 68 Anos de história dedicados à saúde pública. BEPA – Boletim Epidemiológico Paulista.
DETECÇÃO e Identificação de Bactérias de Importância Médica. Agência Nacional de Vigilância Sanitária, Mod V, 2004. 95p.
HOLT, J. G. Bergey’s Manual of Determinative Bacteriology. Ninth Edition – 1994.
JANSSENS, D. et al. The role of public biological resource centers in providing a basic infrastructure for microbial research. Research in microbiology v. 161,6 (2010): 422-9. doi:10.1016/j.resmic.2010.03.009.
O’HARA, C. M.; ROMAN, S. B.; MILLER, J. M. Ability of Commercial Identification Systems To Identify Newly Recognized Species of Citrobacter. Journal of Clinical Microbiology, v.33, n.1, p. 242–245, 1995.
SMITH, D. “Culture collections over the world.” International microbiology : the official journal of the Spanish Society for Microbiology v. 6,2 (2003): 95-100. doi:10.1007/s10123- 003-0114-3
TURTON, J. F. et al. Identification of Acinetobacter baumannii by Detection of the blaOXA-51-like Carbapenemase Gene Intrinsic to This Species. Journal of Clinical Microbiology. London, V. 44, No. 8, , p. 2974–2976, 2006.
WALCKENAER, E. et al. How to identify Raoultella spp. including R. ornithinolytica isolates negative for ornithine decarboxylase? The reliability of the chromosomal bla gene. Journal of microbiological methods v. 75,3 (2008): 405-10. doi:10.1016/j.mimet.2008.07.01.

